34 Gráficos de calor
Os gráficos de calor, também conhecidos como “mapas de calor” ou “blocos/ladrilhos de calor” (do inglês heat tiles) , podem ser visualizações úteis ao tentar exibir 3 variáveis (eixo x, eixo y e preenchimento). Abaixo, demonstramos dois exemplos:
- Uma matriz visual de eventos de transmissão por idade (“quem infectou quem”)
- Acompanhamento de métricas de relatórios em muitas instalações / jurisdições ao longo do tempo
34.1 Preparação
Carregar pacotes
Este pedaço de código mostra o carregamento de pacotes necessários para as análises. Neste manual, enfatizamos p_load() do pacman, que instala o pacote se necessário e o carrega para uso. Você também pode carregar pacotes instalados com library() do R base. Veja a página em Introdução ao R para mais informações sobre pacotes R.
pacman :: p_load(
tidyverse, # manipulação e visualização de dados
rio, # importando dados
lubridate # trabalhando com datas
)Conjuntos de dados
Esta página utiliza a lista de casos de um surto simulado para a seção de matriz de transmissão e um conjunto de dados separado de contagens diárias de casos de malária por instalação para a seção de rastreamento de métricas. Eles são carregados e limpos em suas seções individuais.
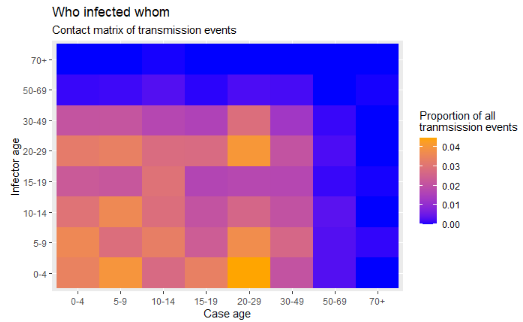
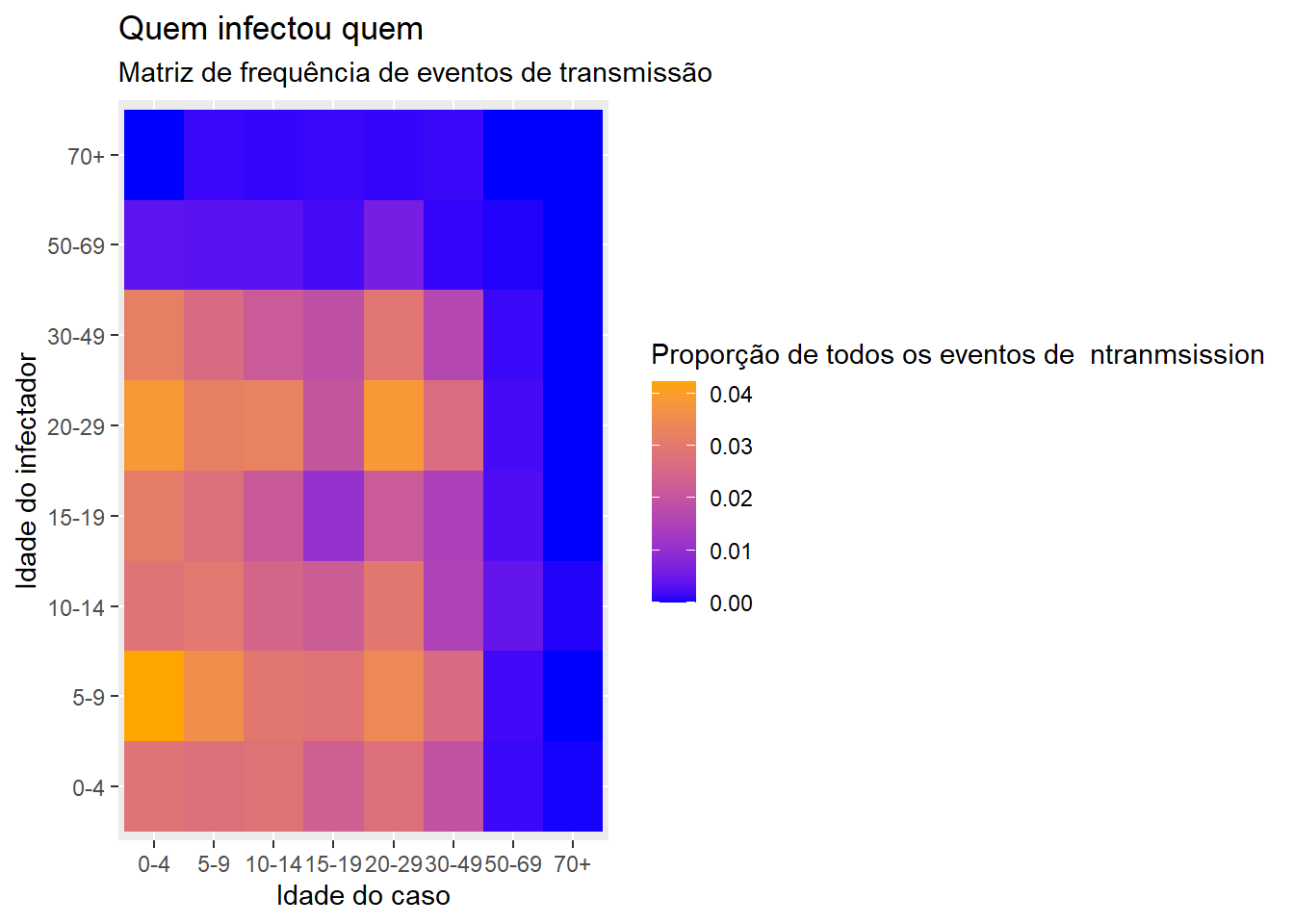
34.2 Matriz de transmissão
Os quadrados de um mapa de calor podem ser úteis para visualizar matrizes. Um exemplo é exibir “quem infectou quem” em um surto. Isso pressupõe que você tenha informações sobre os eventos de transmissão.
Observe que a página Rastreamento de contato contém outro exemplo de criação de uma matriz de contato de do tipo blocos/quadrados térmicos, usando um conjunto de dados diferente (talvez até mais simples) onde as idades dos casos e suas fontes estão perfeitamente alinhadas na mesma linha de observação do quadro de dados (data frame). Esses mesmos dados são usados para fazer um mapa de densidade na página dicas do ggplot. O exemplo abaixo começa com uma linelist de caso e, portanto, envolve uma manipulação considerável de dados antes de obter um data frame que possa ser utilizado em um gráfico. Portanto, existem muitos cenários para escolher…
Começamos com a lista de casos de uma simulação de epidemia de Ebola. Se você quiser acompanhar, clique para baixar o “clean” linelist (as .rds file). Importe dados com a função import() do pacote rio (ele lida com muitos tipos de arquivo como .xlsx, .csv, .rds - veja a página Importar e exportar para detalhes).
As primeiras 50 linhas da linelist são mostradas abaixo para demonstração:
linelist <- import("linelist_cleaned.rds")Nesta linelist:
- Existe uma linha por caso, conforme identificado por
case_id
- Existe uma coluna posterior
infectorque contém ocase_iddo infectador, que também é um caso na linelist
Preparação
Objetivo: Precisamos alcançar um data frame de estilo “longo” que contenha uma linha por rota de transmissão de idade a idade possível, com uma coluna com valores numéricos contendo a proporção dessa linha de todos os eventos de transmissão observados na linelist.
Isso exigirá várias etapas de manipulação de dados para alcançar:
Criar um data frame dos casos
Para começar, criamos um data frame dos casos, suas idades e seus infectantes - chamamos o data frame de idades_de_caso. As primeiras 50 linhas são exibidas abaixo.
case_ages <- linelist%>%
select(case_id, infector, age_cat)%>%
rename("case_age_cat" = "age_cat")Criar um data frame de infectantes
A seguir, criamos um data frame dos infectantes - no momento, ele consiste em uma única coluna. Estas são as IDs de infecção da linelist. Nem todos os casos têm um infectante conhecido, por isso removemos os valores ausentes. As primeiras 50 linhas são exibidas abaixo.
infectors <- linelist %>%
select(infector) %>%
drop_na(infector)Em seguida, usamos junções para obter as idades dos infectantes. Isso não é simples, pois na linelist, as idades do infectador não são listadas como tal. Alcançamos esse resultado juntando a ‘linelist’ dos casos a dos infectantes. Começamos com os infectantes e fazemos um left_join() ( ou seja, o adicionamos) com o linelist, de forma que o data frame de infectates seja a “linha de base” e a coluna infector id do lado esquerdo se junte à coluna case_id na linelist a direita.
Assim, os dados do registro do caso do infectante na linelist (incluindo a idade) são adicionados à linha do infectante. As 50 primeiras linhas são exibidas abaixo.
infector_ages <- infectors%>% # começam com infectores
left_join(# adiciona os dados da linelist para cada infectador
linelist,
by = c("infector" = "case_id"))%>% # corresponde ao infector às suas informações como um caso
select(infector, age_cat)%>% # mantém apenas as colunas de interesse
rename("infector_age_cat" = "age_cat") # rename for clarityEm seguida, combinamos os casos e suas idades com os infectantes e suas idades. Cada um desses data frame tem a coluna infector, então ela é usada para a junção. As primeiras linhas são exibidas abaixo:
ages_complete <- case_ages%>%
left_join(
infector_ages,
by = "infector")%>% # utilizando a coluna `infector`coomo chave para a junção
drop_na() # excluir linhas com qualquer dado faltanteWarning in left_join(., infector_ages, by = "infector"): Detected an unexpected many-to-many relationship between `x` and `y`.
ℹ Row 1 of `x` matches multiple rows in `y`.
ℹ Row 6 of `y` matches multiple rows in `x`.
ℹ If a many-to-many relationship is expected, set `relationship =
"many-to-many"` to silence this warning.Abaixo, uma tabulação cruzada simples de contagens entre os casos e os grupos de idade dos infectantes. Rótulos foram adicionadas para maior clareza.
table(cases = ages_complete$case_age_cat,
infectors = ages_complete$infector_age_cat) infectors
cases 0-4 5-9 10-14 15-19 20-29 30-49 50-69 70+
0-4 105 156 105 114 143 117 13 0
5-9 102 132 110 102 117 96 12 5
10-14 104 109 91 79 120 80 12 4
15-19 85 105 82 39 75 69 7 5
20-29 101 127 109 80 143 107 22 4
30-49 72 97 56 54 98 61 4 5
50-69 5 6 15 9 7 5 2 0
70+ 1 0 2 0 0 0 0 0Podemos converter esta tabela em um dataframe com data.frame() do R base, que também converte automaticamente para o formato “longo”, que é desejado para o ggplot(). As primeiras linhas são mostradas abaixo.
long_counts <- data.frame(table(
cases = ages_complete$case_age_cat,
infectors = ages_complete$infector_age_cat))Agora fazemos o mesmo, mas aplicamos prop.table() do R base para a tabela de forma que, em vez de contagens, obtenhamos proporções do total. As primeiras 50 linhas são mostradas abaixo.
long_prop <- data.frame(prop.table(table(
cases = ages_complete$case_age_cat,
infectors = ages_complete$infector_age_cat)))Criar gráfico de calor
Agora, finalmente, podemos criar o gráfico de calor com o pacote ggplot2, usando a função geom_tile(). Veja a página de dicas do ggplot para aprender mais extensivamente sobre as escalas de cor / preenchimento, especialmente a função scale_fill_gradient().
- Na estética
aes()degeom_tile()defina x e y como a idade do caso e idade infectante
- Também em
aes()defina o argumentofill =para a colunaFreq- este é o valor que será convertido para uma cor de bloco
- Defina uma cor de escala com
scale_fill_gradient()- você pode especificar as cores altas / baixas- Observe que
scale_color_gradient()é diferente! Neste caso, você quer o preenchimento
- Observe que
- Como a cor é feita por meio de “preenchimento”, você pode usar o argumento
fill =emlabs()para alterar o título da legenda
ggplot(data = long_prop) + # usa um dataframe no formato longo, com proporções como Freq
geom_tile(# visualizar em quadrados
aes(
x = cases, # eixo x é a idade do caso
y = infectors, # eixo y é a idade do infectador
fill = Freq)) + # cor de cada quadrado é a coluna Freq nos dados
scale_fill_gradient(# ajusta a cor de preenchimento dos quadrados
low = "blue",
high = "orange")+
labs(# rótulos
x = "Idade do caso",
y = "Idade do infectador",
title = "Quem infectou quem",
subtitle = "Matriz de frequência de eventos de transmissão",
fill = "Proporção de todos os eventos de \ ntranmsission" # título da legenda
)34.3 Métricas de relatório ao longo do tempo
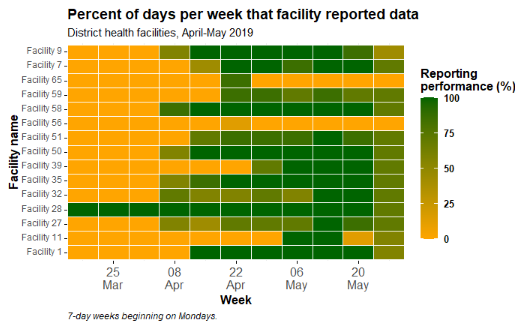
Frequentemente, na saúde pública, um objetivo é avaliar as tendências ao longo do tempo para muitas entidades (instalações, jurisdições, etc.). Uma maneira de visualizar essas tendências ao longo do tempo é um gráfico de calor em que o eixo x é o tempo e no eixo y estão as várias entidades.
Preparação
Começamos importando um conjunto de dados de relatórios diários da malária de muitos estabelecimentos. Os relatórios contêm uma data, província, distrito e contagens de malária. Consulte a página em Baixar manual e dados para obter informações sobre como baixar esses dados. Abaixo estão as primeiras 30 linhas:
facility_count_data <- import("malaria_facility_count_data.rds")Agregar e resumir
O objetivo neste exemplo é transformar as contagens diárias de casos de malária total dos estabelecimentos (visto na guia anterior) em estatísticas resumidas semanais de desempenho de relatórios das instalações - neste caso a proporção de dias por semana que a instalação/estabelecimento relatou quaisquer dados. Para este exemplo, mostraremos dados apenas para Spring District.
Para conseguir isso, faremos as seguintes etapas de gerenciamento de dados:
- Filtre os dados conforme apropriado (por local, data)
- Crie uma coluna de semana usando
floor_date()do pacote lubridate- Esta função retorna a data de início da semana de uma determinada data, usando uma data de início especificada de cada semana (por exemplo, “Segundas”)
- Esta função retorna a data de início da semana de uma determinada data, usando uma data de início especificada de cada semana (por exemplo, “Segundas”)
- Os dados são agrupados pelas colunas “local” e “semana” para criar unidades de análise de “semana-estabelecimento”
- A função
resumir()cria novas colunas para refletir as estatísticas de resumo por grupo de semana-estabelecimento:- Número de dias por semana (7 - um valor estático)
- Número de relatórios recebidos da semana-estabelecimento (pode ser mais de 7!)
- Soma dos casos de malária relatados pela semana-estabelecimento (apenas por interesse)
- Número de dias únicos na semana-estabelecimento para os quais há dados relatados
-
Porcentagem dos 7 dias por semana-estabelecimento para os quais os dados foram relatados
- Número de dias por semana (7 - um valor estático)
- Odata frame é unido com
right_join()a uma lista abrangente de todas as combinações possíveis de semana-estabelecimento, para tornar o conjunto de dados completo. A matriz de todas as combinações possíveis é criada aplicandoexpand()a essas duas colunas dodata frame, como está naquele momento na cadeia de pipes (representado por.). Como umright_join()é usado, todas as linhas no data frameexpand()são mantidas e adicionadas aagg_weeksse necessário. Essas novas linhas aparecem com valores resumidosNA(ausentes).
Abaixo, demonstramos passo a passo:
# Crie um conjunto de dados de resumo semanal
agg_weeks <- facility_count_data%>%
# filtrar os dados conforme apropriado
filter(
District == "Spring",
data_date < as.Date("2020-08-01")) Agora o conjunto de dados tem nrow(agg_weeks) linhas, quando anteriormente tinha nrow(facility_count_data).
Em seguida, criamos uma coluna semana refletindo a data de início da semana para cada registro. Isso é obtido com o pacote lubridate e a função floor_date(), que é definida como “semana” e para as semanas com início às segundas-feiras (dia 1 da semana - domingos seria 7). As linhas superiores são mostradas abaixo.
agg_weeks <- agg_weeks %>%
# Crie a coluna da semana a partir de data_date
mutate(
week = lubridate::floor_date(# criar uma nova coluna de semanas
data_date, # date
unit = "week", # dá o início da semana
week_start = 1)) # semanas para começar às segundas-feiras A nova coluna da semana pode ser vista na extremidade direita do quadro de dados
Agora agrupamos os dados em semana-instalação e os resumimos para produzir estatísticas por semana-instalação. Consulte a página em Tabelas descritivas para dicas. O próprio agrupamento não altera o quadro de dados, mas impacta como as estatísticas de resumo subsequentes são calculadas.
As linhas superiores são mostradas abaixo. Observe como as colunas mudaram completamente para refletir as estatísticas de resumo desejadas. Cada linha reflete uma semana-instalação.
agg_weeks <- agg_weeks %>%
# Grupo em semana-estabelecimento
group_by(location_name, week) %>%
# Crie colunas de estatísticas de resumo nos dados agrupados
summarise(
n_days = 7, # 7 dias por semana
n_reports = dplyr::n(), # número de relatórios recebidos por semana (pode ser> 7)
malaria_tot = sum (malaria_tot, na.rm = T), # total de casos de malária relatados
n_days_reported = length(unique (data_date)), # número de dias únicos de relatórios por semana
p_days_reported = round(100*(n_days_reported / n_days))) # por cento de relatórios de diasPor fim, executamos o comando abaixo para garantir que TODAS as semanas-estabelecimento possíveis estejam presentes nos dados, mesmo que não existissem antes.
Estamos usando um right_join() em si mesmo (o conjunto de dados é representado por “.”), Mas foi expandido para incluir todas as combinações possíveis das colunas semana e localização_nome. Veja a documentação sobre a função expand() na página Pivoteando Dados. Antes de executar este código, o conjunto de dados contém linhas nrow(agg_weeks).
# Crie um dataframe com todas as possibilidades de combinação semana-estabelecimento
expanded_weeks <- agg_weeks%>%
tidyr::expand(week) # expanda data frame para incluir todas as combinações possíveis de semana-estabelecimentoAqui está expanded_weeks:
Antes de executar este código, agg_weeks contém linhas nrow(agg_weeks).
# Use uma junção à direita com a lista expandida semana-estabelecimento para preencher as lacunas que faltam nos dados
agg_weeks <- agg_weeks %>%
right_join(expanded_weeks)%>% # Certifique-se de que todas as combinações possíveis de estabelecimento-semana apareçam nos dados
mutate(p_days_reported = replace_na(p_days_reported, 0)) # converter valores ausentes para 0 Joining with `by = join_by(location_name, week)`Depois de executar este código, agg_weeks contém linhas nrow(agg_weeks).
Criar gráfico de calor
O ggplot() é feito usando geom_tile() do pacote ggplot2:
- Semanas no eixo x são transformadas em datas, permitindo o uso de
scale_x_date()
-
location_nameno eixo y mostrará todos os nomes de estabelecimentos - O
fillép_days_reported, o desempenho para aquela semana-estabelecimento(numérico)
-
scale_fill_gradient()é usado no preenchimento numérico, especificando cores para alto, baixo eNA
-
scale_x_date()é usado no eixo x especificando rótulos a cada 2 semanas e seu formato
- Temas de exibição e rótulos podem ser ajustados conforme necessário
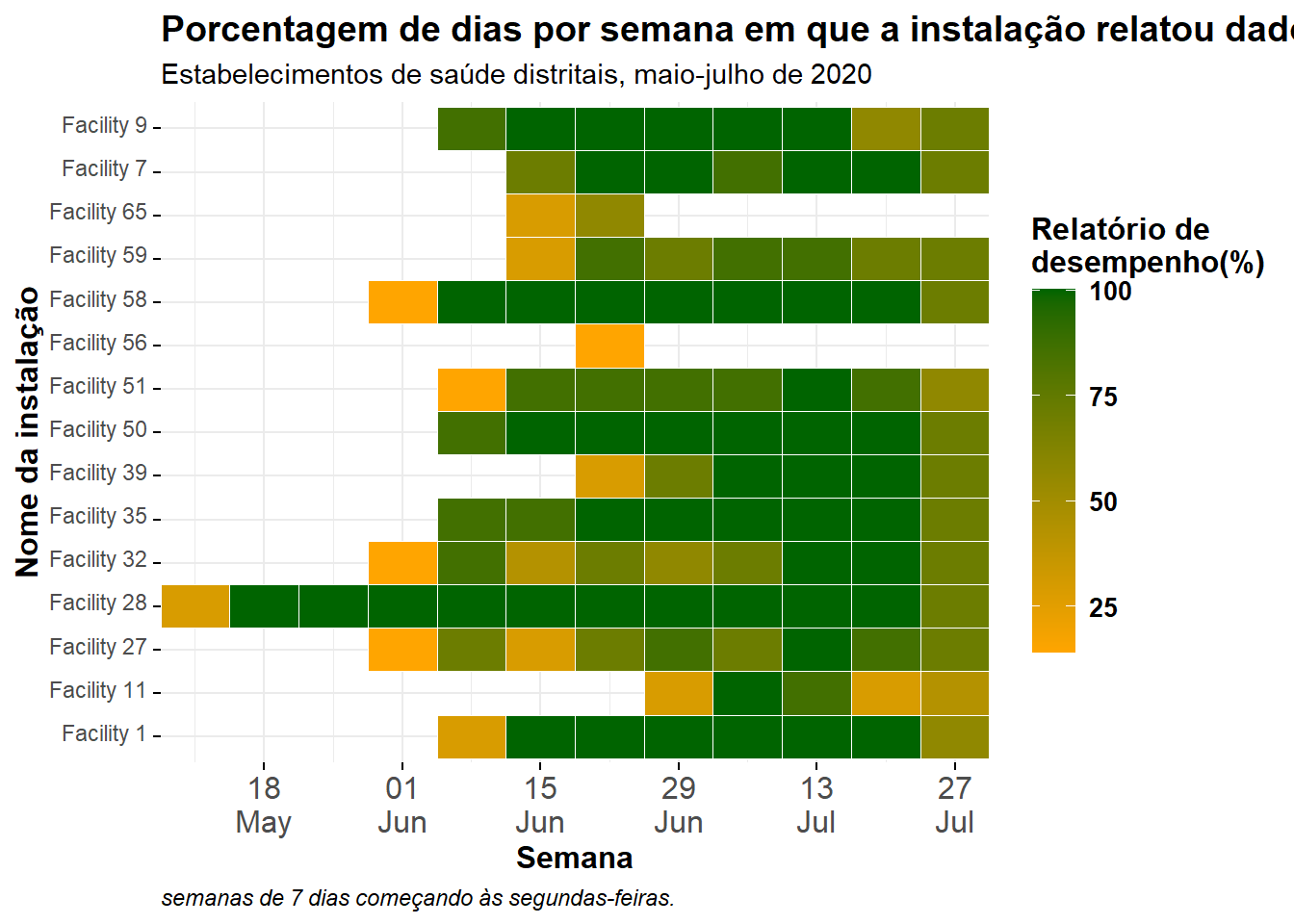
Básico
Um gráfico de calor básico é produzido abaixo, usando as cores e escalas padrão. Como explicado acima, dentro de aes() para geom_tile() você deve fornecer uma coluna do eixo x, coluna do eixo y e uma coluna para o fill =. O preenchimento é o valor numérico apresentado como cor do bloco.
Gráfico limpo
Podemos fazer esse gráfico parecer melhor adicionando funções ggplot2 adicionais, conforme mostrado abaixo. Veja a página em dicas do ggplot para detalhes.
ggplot(data = agg_weeks)+
# mostrar dados como quadrados
geom_tile(
aes(x = week,
y = location_name,
fill = p_days_reported),
color = "white") + # linhas de grade brancas
scale_fill_gradient(
low = "orange",
high = "darkgreen",
na.value = "grey80")+
# eixo de data
scale_x_date(
expand = c(0,0), # remove espaço extra nas laterais
date_breaks = "2 weeks", # rótulos a cada 2 semanas
date_labels = "%d\n%b") + # formato é dia após mês (\n em nova linha)
# temas estéticos
theme_minimal() + # fundo simplificado
theme(
legend.title = element_text(size=12, face="bold"),
legend.text = element_text(size=10, face="bold"),
legend.key.height = grid::unit(1, "cm"), # altura da chave da legenda
legend.key.width = grid::unit(0.6, "cm"), # largura da chave da legenda
axis.text.x = element_text(size=12), # axis text size
axis.text.y = element_text(vjust = 0.2), # alinhamento do texto do eixo
axis.ticks = element_line(size=0.4),
axis.title = element_text(size = 12, face = "bold"), # tamanho do título do eixo e negrito
plot.title = element_text(hjust=0,size=14,face="bold"), # title right-aligned, large, bold
plot.caption = element_text(hjust = 0, face = "italic") # legenda alinhado à direita e itálico
)+
# rótulos de gráfico
labs(x = "Semana",
y = "Nome da instalação",
fill = "Relatório de \ndesempenho(%)", # título da legenda, porque a legenda mostra preenchimento
title = "Porcentagem de dias por semana em que a instalação relatou dados",
subtitle = "Estabelecimentos de saúde distritais, maio-julho de 2020",
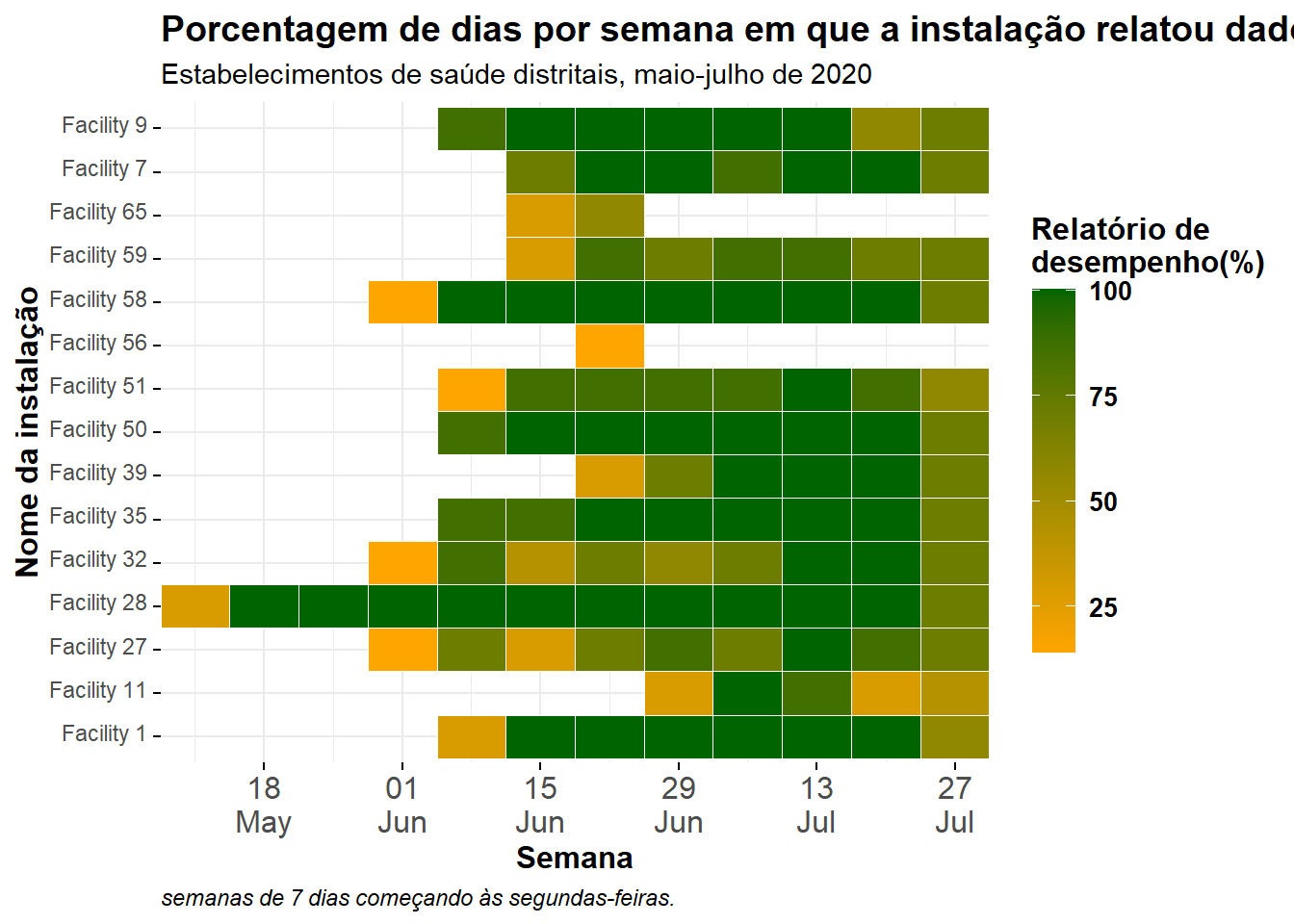
caption = "semanas de 7 dias começando às segundas-feiras.")Eixo y ordenado
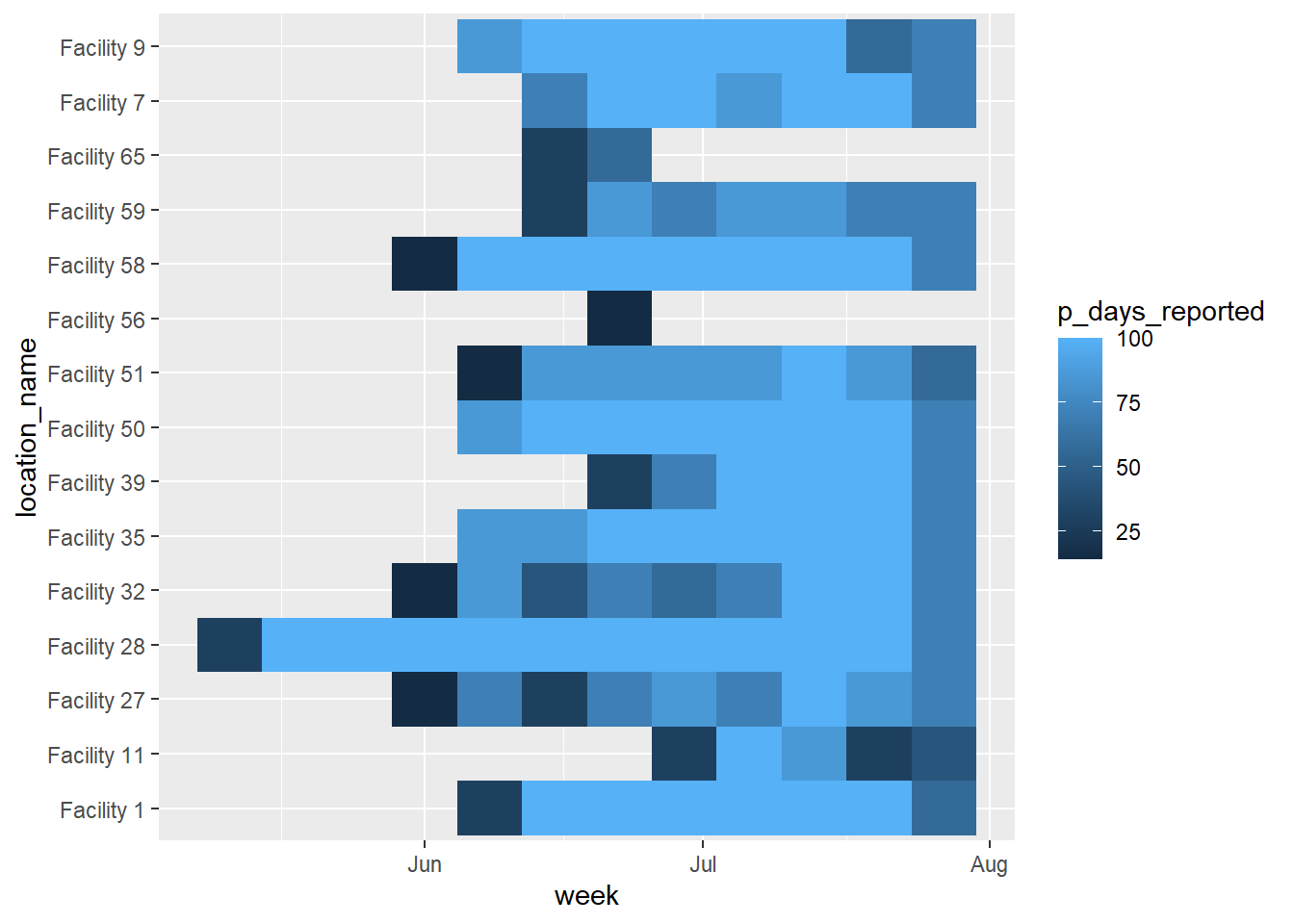
Atualmente, as instalações são ordenadas “alfanumericamente” de baixo para cima. Se você quiser ajustar a ordem das facilidades do eixo y, converta-as em fator de classe e forneça a ordem. Veja a página em Fatores para dicas.
Uma vez que existem muitos recursos e não queremos escrevê-los todos, tentaremos outra abordagem - ordenar os recursos em um data frame e usar a coluna de nomes resultante como a ordem dos níveis do fator. Abaixo, a coluna location_name é convertida em um fator, e a ordem de seus níveis é definida com base no número total de dias de relatório arquivados pela instalação/estabelecimento ao longo de todo o período de tempo.
Para fazer isso, criamos um data frame que representa o número total de relatórios por instalação, organizados em ordem crescente. Podemos usar este vetor para ordenar os níveis dos fatores no gráfico.
facility_order <- agg_weeks %>%
group_by(location_name)%>%
summarize(tot_reports = sum(n_days_reported, na.rm=T)) %>%
arrange(tot_reports) # ordem crescenteVeja o data frame abaixo:
Agora use uma coluna do data frame acima (facility_order$location_name) para ser a ordem dos níveis de fator de location_name no quadro de dados agg_weeks:
E agora os dados são adicionados a um gráfico novamente, com location_name sendo um fator ordenado:
ggplot(data = agg_weeks)+
# mostrar dados como quadrados
geom_tile(
aes(x = week,
y = location_name,
fill = p_days_reported),
color = "white") + # linhas de grade brancas
scale_fill_gradient(
low = "orange",
high = "darkgreen",
na.value = "grey80")+
# eixo de data
scale_x_date(
expand = c(0,0), # remove espaço extra nas laterais
date_breaks = "2 weeks", # rótulos a cada 2 semanas
date_labels = "%d\n%b") + # formato é dia após mês (\n em nova linha)
# temas estéticos
theme_minimal() + # fundo simplificado
theme(
legend.title = element_text(size=12, face="bold"),
legend.text = element_text(size=10, face="bold"),
legend.key.height = grid::unit(1, "cm"), # altura da chave da legenda
legend.key.width = grid::unit(0.6, "cm"), # largura da chave da legenda
axis.text.x = element_text(size=12), # axis text size
axis.text.y = element_text(vjust = 0.2), # alinhamento do texto do eixo
axis.ticks = element_line(size=0.4),
axis.title = element_text(size = 12, face = "bold"), # tamanho do título do eixo e negrito
plot.title = element_text(hjust=0,size=14,face="bold"), # title right-aligned, large, bold
plot.caption = element_text(hjust = 0, face = "italic") # legenda alinhado à direita e itálico
)+
# rótulos de gráfico
labs(x = "Semana",
y = "Nome da instalação",
fill = "Relatório de \ndesempenho(%)", # título da legenda, porque a legenda mostra preenchimento
title = "Porcentagem de dias por semana em que a instalação relatou dados",
subtitle = "Estabelecimentos de saúde distritais, maio-julho de 2020",
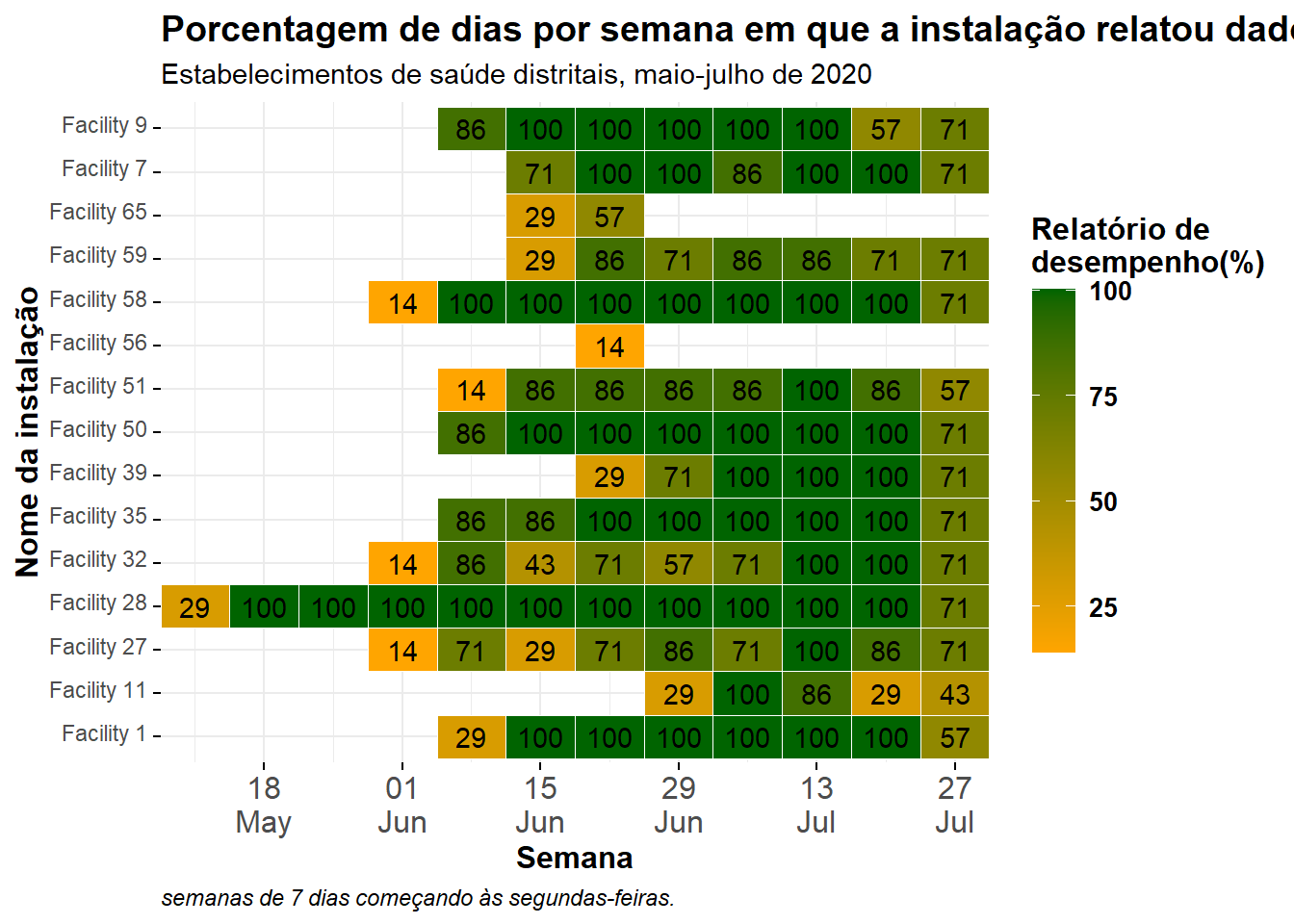
caption = "semanas de 7 dias começando às segundas-feiras.")Valores expostos
Você pode adicionar uma camada geom_text() no topo dos quadrados, para exibir os números reais de cada quadrado. Esteja ciente de que isso pode não parecer muito elegante se você tiver muitos quadradinhos pequenos!
O seguinte código foi adicionado: geom_text(aes(label = p_days_reported)). Isso adiciona texto a cada bloco. O texto exibido é o valor atribuído ao argumento label =, que neste caso foi definido para a mesma coluna numérica p_days_reported que também é usada para criar o gradiente de cor.
ggplot(data = agg_weeks)+
# mostrar dados como quadrados
geom_tile(
aes(x = week,
y = location_name,
fill = p_days_reported),
color = "white") + # linhas de grade brancas
# texto
geom_text(
aes(
x = week,
y = location_name,
label = p_days_reported)) + # adicionar texto no topo do quadrado
# escala de preenchimento
scale_fill_gradient(
low = "orange",
high = "darkgreen",
na.value = "grey80")+
# eixo de data
scale_x_date(
expand = c(0,0), # remove espaço extra nas laterais
date_breaks = "2 weeks", # rótulos a cada 2 semanas
date_labels = "%d\n%b") + # formato é dia após mês (\n em nova linha)
# temas estéticos
theme_minimal() + # fundo simplificado
theme(
legend.title = element_text(size=12, face="bold"),
legend.text = element_text(size=10, face="bold"),
legend.key.height = grid::unit(1, "cm"), # altura da chave da legenda
legend.key.width = grid::unit(0.6, "cm"), # largura da chave da legenda
axis.text.x = element_text(size=12), # axis text size
axis.text.y = element_text(vjust = 0.2), # alinhamento do texto do eixo
axis.ticks = element_line(size=0.4),
axis.title = element_text(size = 12, face = "bold"), # tamanho do título do eixo e negrito
plot.title = element_text(hjust=0,size=14,face="bold"), # title right-aligned, large, bold
plot.caption = element_text(hjust = 0, face = "italic") # legenda alinhado à direita e itálico
)+
# rótulos de gráfico
labs(x = "Semana",
y = "Nome da instalação",
fill = "Relatório de \ndesempenho(%)", # título da legenda, porque a legenda mostra preenchimento
title = "Porcentagem de dias por semana em que a instalação relatou dados",
subtitle = "Estabelecimentos de saúde distritais, maio-julho de 2020",
caption = "semanas de 7 dias começando às segundas-feiras.")