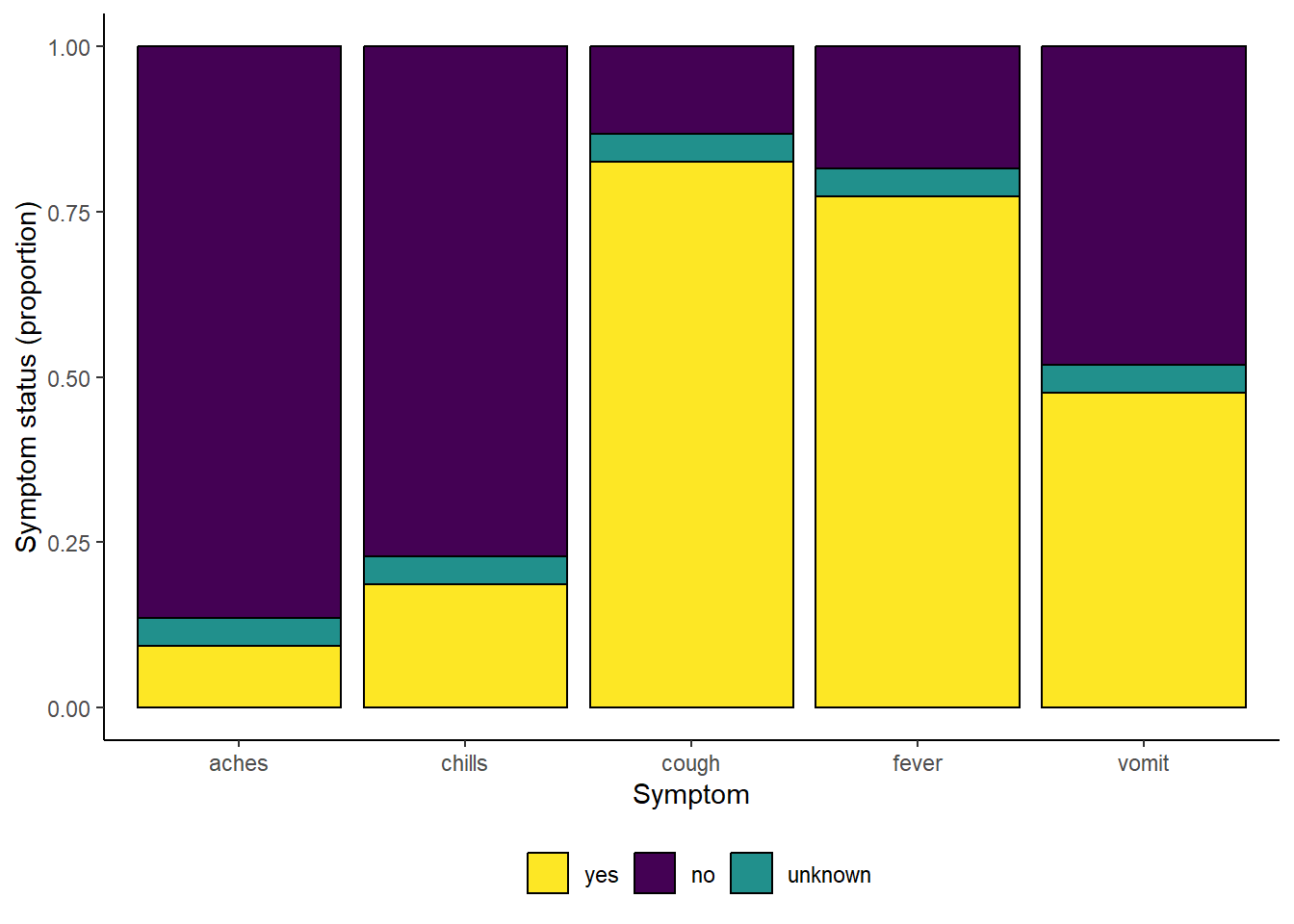pacman::p_load(
tidyverse, # включает в себя ggplot2 и другие
rio, # импорт/экспорт
here, # локатор файлов
stringr, # работа с символами
scales, # преобразование чисел
ggrepel, # грамотно расставленные метки
gghighlight, # выделение одной части графика
RColorBrewer # цветовые шкалы
)31 Советы по использованию ggplot
На этой странице мы рассмотрим советы и приемы, позволяющие сделать ваши ggplots более четкими и красивыми. Основы работы с ggplot описаны на странице Основы ggplot.
В разделе “Ресурсы” есть несколько подробных учебных пособий по [ggplot2] (https://ggplot2.tidyverse.org/). Вы также можете загрузить шпаргалку Визуализация данных с помощью ggplot с сайта RStudio. Мы настоятельно рекомендуем обратиться за вдохновением к разделам галерея графиков R и От данных к визуализации.
31.1 Подготовка
Загрузка пакетов
В этом фрагменте кода показана загрузка пакетов, необходимых для проведения анализа. В данном руководстве мы делаем акцент на функции p_load() из pacman, которая при необходимости устанавливает пакет и загружает его для использования. Установленные пакеты можно также загрузить с помощью library() из базового R. Более подробную информацию о пакетах R см. на странице Основы R.
Импорт данных
Для этой страницы мы импортируем набор данных случаев из смоделированной эпидемии лихорадки Эбола. Если вы хотите выполнять действия параллельно, нажмите кнопку, чтобы загрузить “чистый” построчный список (в виде файла .rds). Импортируйте данные с помощью функции import() из пакета rio (она работает со многими типами файлов, такими как .xlsx, .csv, .rds - подробности см. на странице Импорт и экспорт).
linelist <- rio::import("linelist_cleaned.rds")Ниже отображаются первые 50 строк построчного списка.
31.2 Шкалы для цвета, заливки, осей и т.д.
В ggplot2, когда эстетические характеристики графических данных (например, размер, цвет, форма, заливка, ось графика) привязаны к столбцам данных, точное отображение можно настроить с помощью соответствующей команды “scale”. В этом разделе мы расскажем о некоторых распространенных способах настройки шкал.
31.2.1 Цветовые схемы
Одним из моментов, который поначалу может оказаться сложным для понимания в ggplot2, является управление цветовыми схемами. Обратите внимание на то, что в данном разделе рассматривается цвет объектов графика (геомов/фигур), таких как точки, отрезки, линии, плитки и т.д. Для настройки цвета текста, заголовков или цвета фона см. раздел Темы на странице Основы ggplot.
Для управления “цветом” объектов графика необходимо настраивать либо эстетику color = (внешний цвет), либо эстетику fill = (внутренний цвет). Исключением из этого правила является функция geom_point(), где действительно можно управлять только color =, который управляет цветом точки (внутренним и внешним).
При задании цвета или заливки можно использовать названия цветов, распознаваемые R, например, "red" (см. полный список или введите ?colors), или конкретный шестнадцатеричный код цвета, например, "#ff0505".
# гистограмма -
ggplot(data = linelist, mapping = aes(x = age))+ # set data and axes
geom_histogram( # отобразит гистаграмму
binwidth = 7, # ширина корзин
color = "red", # цвет линии корзины
fill = "lightblue") # внутренний цвет корзины (заливка) Как объясняется в разделе Основы ggplot, посвященном вопросу сопоставление данных с графиком, такие эстетические параметры, как fill = и color =, могут быть заданы как вне оператора mapping = aes(), так и внутри него. В случае внешнего оператора aes() присвоенное значение должно быть статичным (например, color = "blue") и будет применяться для всех данных, построенных с помощью геома. Если внутри, то эстетика должна быть сопоставлена со столбцом, например, color = hospital, и выражение будет меняться в зависимости от значения этой строки в данных. Несколько примеров:
# Статический цвет для точек и для линии
ggplot(data = linelist, mapping = aes(x = age, y = wt_kg))+
geom_point(color = "purple")+
geom_vline(xintercept = 50, color = "orange")+
labs(title = "Static color for points and line")
# Цвет сопоставлен с непрерывным столбцом
ggplot(data = linelist, mapping = aes(x = age, y = wt_kg))+
geom_point(mapping = aes(color = temp))+
labs(title = "Color mapped to continuous column")
# Цвет сопоставлен с дискретным столбцом
ggplot(data = linelist, mapping = aes(x = age, y = wt_kg))+
geom_point(mapping = aes(color = gender))+
labs(title = "Color mapped to discrete column")
# столбчатая диаграмма, заливка сопоставлена с дискретным столбцом, цвет - со статическим значением
ggplot(data = linelist, mapping = aes(x = hospital))+
geom_bar(mapping = aes(fill = gender), color = "yellow")+
labs(title = "Fill mapped to discrete column, static color")Шкалы
После сопоставления столбца с эстетикой графика (например, x =, y =, fill =, color =…) у графика появляется шкала/легенда. См. выше, что шкала может быть непрерывной, дискретной, датой и т.д. в зависимости от класса назначенного столбца. Если столбцам сопоставлено несколько эстетик, то участок будет иметь несколько шкал.
Управлять шкалами можно с помощью соответствующей функции scales_(). Функции шкал в ggplot() состоят из трех частей, которые записываются следующим образом: scale_AESTHETIC_METHOD()..
- Первая часть,
scale_(), является фиксированной.
- Вторая часть, ЭСТЕТИКА, должна быть эстетикой, для которой вы хотите настроить масштаб (
_fill_,_shape_,_color_,_size_,_alpha_…) - опции здесь также включают_x_и_y_.
- Третья часть, МЕТОД, будет либо
_discrete(),continuous(),_date(),_gradient(), либо_manual()в зависимости от класса столбца и того, как вы хотите им управлять. Существуют и другие, но эти наиболее часто используемые.
Убедитесь, что вы используете правильную функцию для шкалы! В противном случае окажется, что команда изменения шкалы ничего не меняет. Если у вас несколько шкал, то для их настройки можно использовать несколько функций шкалы! Например:
Аргументы шкалы
Каждый вид шкалы имеет свои аргументы, хотя они частично совпадают. Запросите функцию типа ?scale_color_discrete в консоли R, чтобы увидеть документацию по аргументам функции.
Для непрерывных шкал используйте breaks = и задайте последовательность значений с помощью seq() (возьмите to =, from = и by =, как показано в примере ниже. Установите expand = c(0,0), чтобы удалить наращивание пробелов вокруг осей (это может быть использовано для любой шкалы _x_ или _y_.
Для дискретных шкал можно настроить порядок появления уровней с помощью аргумента breaks =, а также порядок отображения значений с помощью аргумента labels =. Каждому из них присвойте символьный вектор (см. пример ниже). Также можно легко отказаться от NA, задав na.translate = FALSE.
Более подробно о тонкостях шкал дат рассказано на странице [Эпидемические кривые].
Ручные настройки
Одним из наиболее полезных приемов является использование функций ” Ручной” настройки шкалы для прямого назначения цветов по своему усмотрению. Это функции с синтаксисом scale_xxx_manual() (например, scale_colour_manual() или scale_fill_manual()). Каждый из перечисленных ниже аргументов демонстрируется в приведенном ниже примере кода.
- Назначьте цвета значениям данных с помощью аргумента
values =.
- Задайте цвет для
NAс помощью аргументаna.value =.
- Измените то, как значения будут записаны в легенде с помощью аргумента
labels =.
- Измените название легенды с помощью аргумента
name =. `
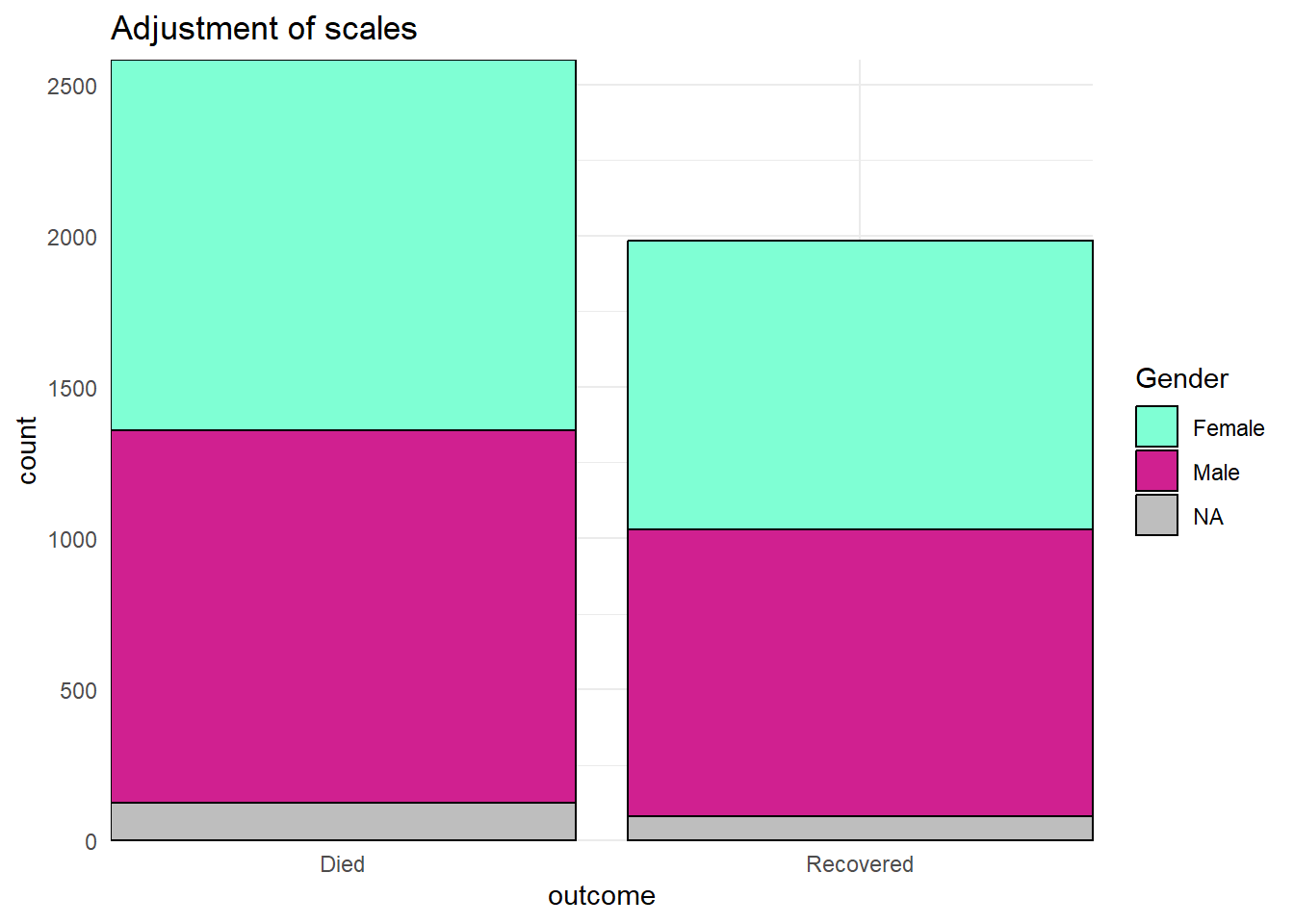
Ниже мы создадим столбчатую диаграмму и покажем, как она выглядит по умолчанию, а затем настроим три шкалы - непрерывную по оси y, дискретную по оси x и ручную настройку заливки (цвета внутреннего столбца).
# Исходная ситуация - без настройки шкалы
ggplot(data = linelist)+
geom_bar(mapping = aes(x = outcome, fill = gender))+
labs(title = "Baseline - no scale adjustments")# НАСТРОЙКА ШКАЛЫ
ggplot(data = linelist)+
geom_bar(mapping = aes(x = outcome, fill = gender), color = "black")+
theme_minimal()+ # упростить фон
scale_y_continuous( # непрерывная шкала для оси y (количество)
expand = c(0,0), # без наращивания
breaks = seq(from = 0,
to = 3000,
by = 500))+
scale_x_discrete( # дискретная шкала для оси x (пол)
expand = c(0,0), # без наращивания
drop = FALSE, # показать все уровни факторов (даже если их нет в данных)
na.translate = FALSE, # удалить исходы NA из графика
labels = c("Died", "Recovered"))+ # Изменение отображения значений
scale_fill_manual( # Вручную задать заливку (внутренний цвет столбика)
values = c("m" = "violetred", # референсные значения в данных для присвоения цветов
"f" = "aquamarine"),
labels = c("m" = "Male", # Изменить метки в легенде (во избежание ошибок используйте присваивание "=")
"f" = "Female",
"Missing"),
name = "Gender", # заголовок легенды
na.value = "grey" # назначение цвета для отсутствующего значенияs
)+
labs(title = "Adjustment of scales") # Настройка заголовка легенды заливкиШкалы непрерывных осей
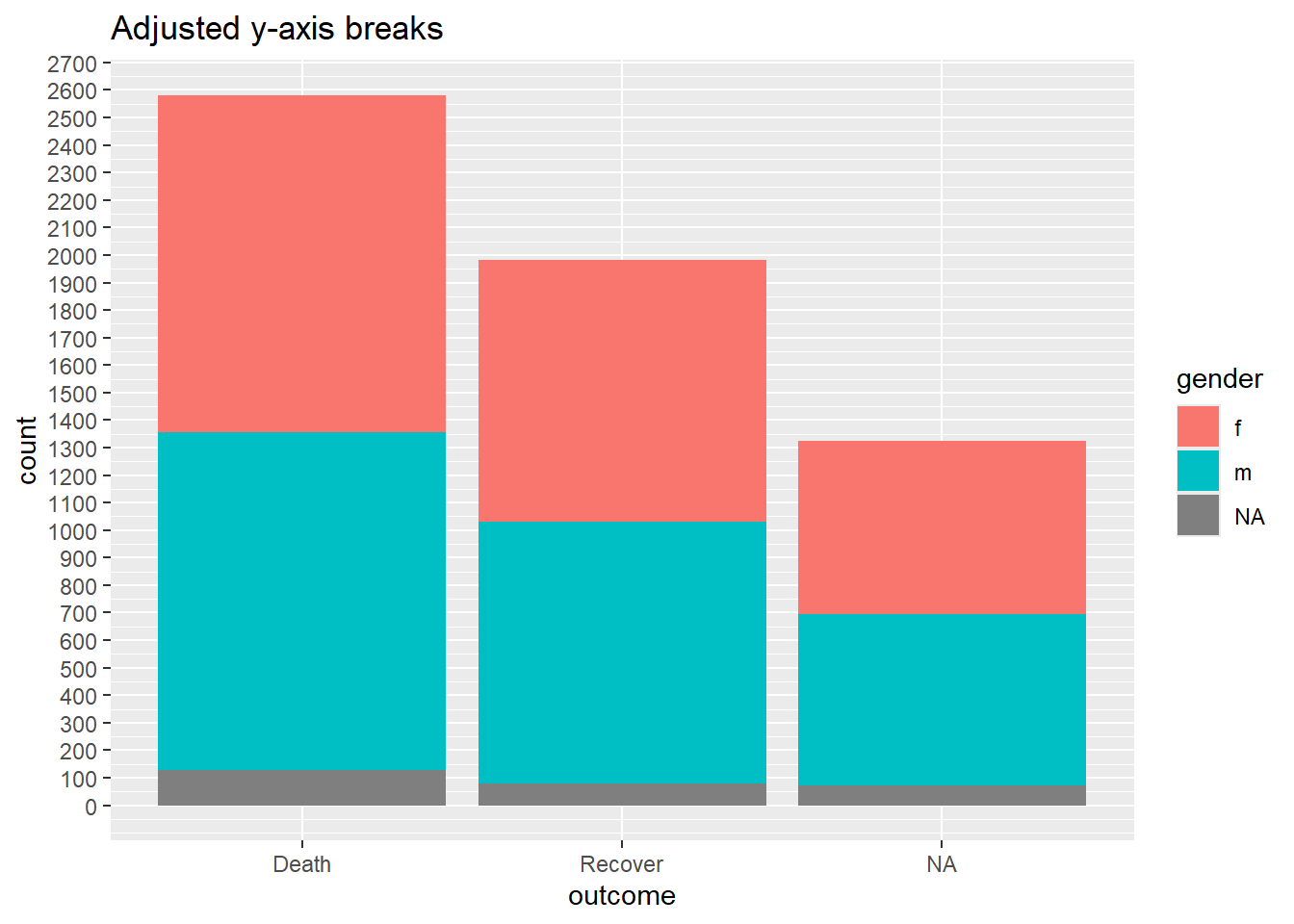
Когда данные сопоставляются с осями графика, они также могут быть настроены с помощью команд шкал. Частым примером является настройка отображения оси (например, оси y), которая сопоставлена столбцу с непрерывными данными.
Мы можем захотеть настроить разрывы или отображение значений в ggplot, используя команду scale_y_continuous(). Как отмечалось выше, с помощью аргумента breaks = можно задать последовательность значений, которые будут служить ” разрывами” вдоль шкалы. Это те значения, при которых будут отображаться числа. В качестве аргумента можно указать вектор c(), содержащий желаемые значения разрывов, либо задать обычную последовательность чисел с помощью базовой функции R seq(). Эта функция seq() принимает значения to =, from = и by =.
# Исходная ситуация - без настройки шкалы
ggplot(data = linelist)+
geom_bar(mapping = aes(x = outcome, fill = gender))+
labs(title = "Baseline - no scale adjustments")
#
ggplot(data = linelist)+
geom_bar(mapping = aes(x = outcome, fill = gender))+
scale_y_continuous(
breaks = seq(
from = 0,
to = 3000,
by = 100)
)+
labs(title = "Adjusted y-axis breaks")Отображение процентов
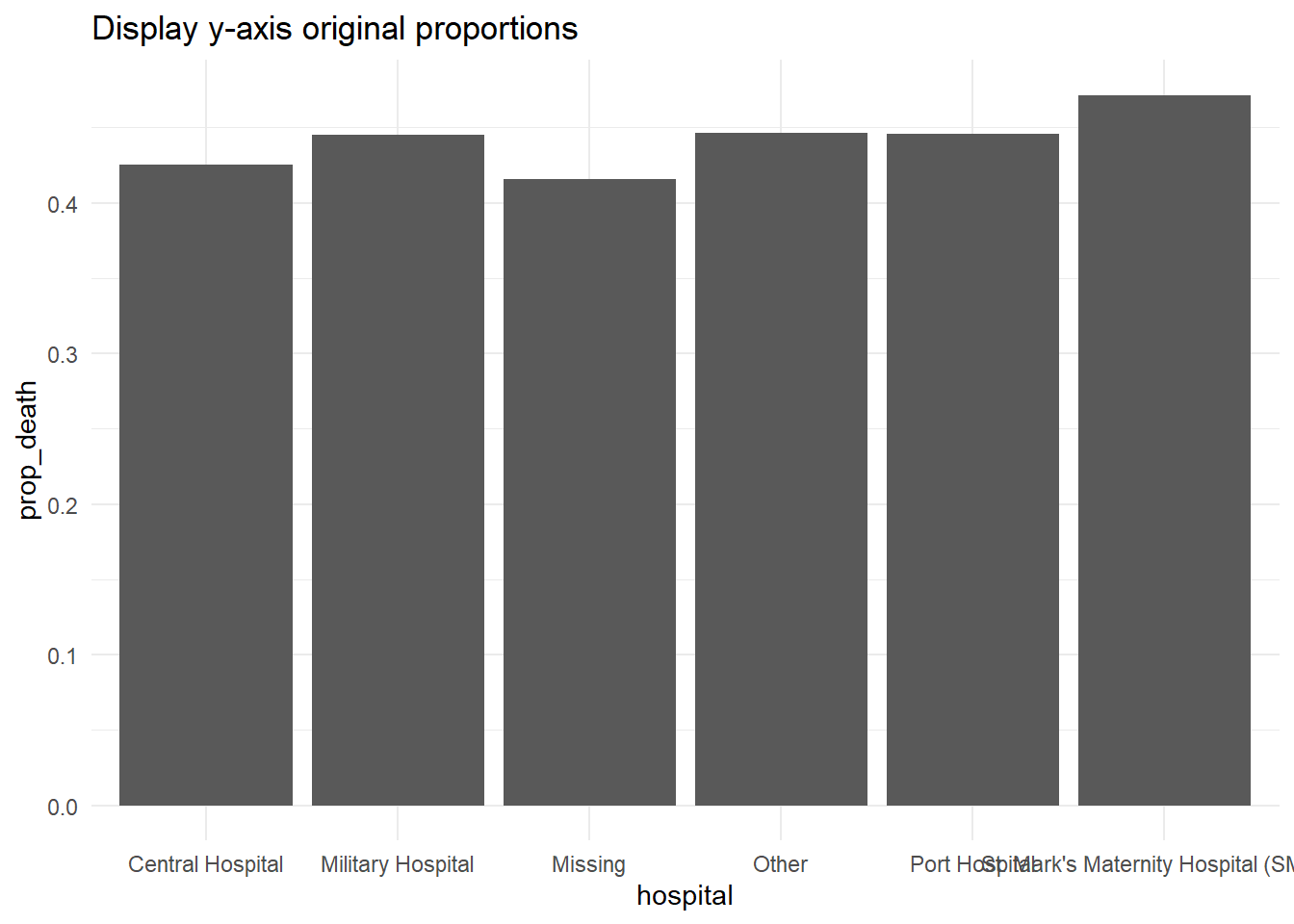
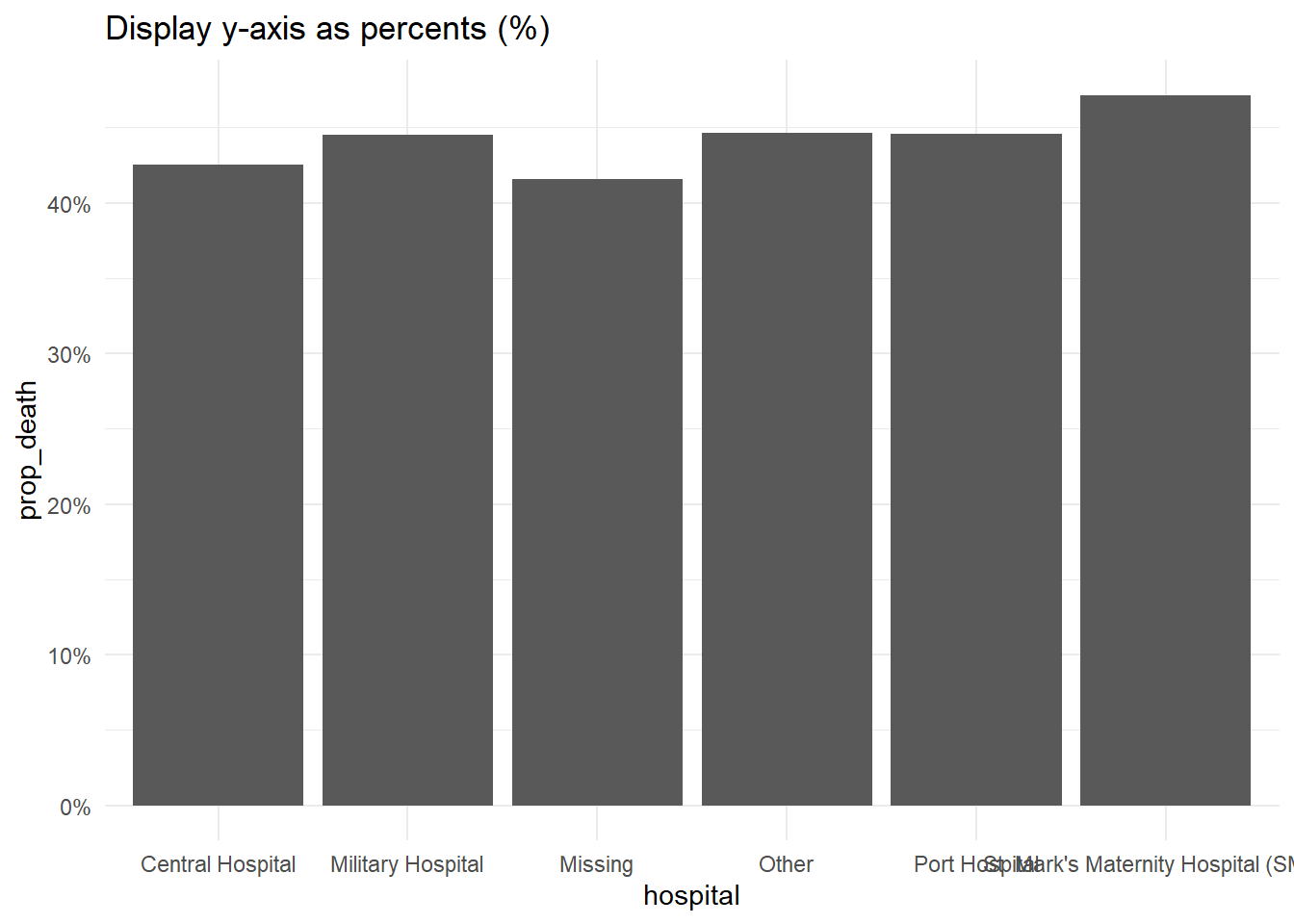
Если исходные значения данных представляют собой пропорции, то их можно легко отобразить в виде процентов с помощью “%”, указав в команде шкалы labels = scales::percent, как показано ниже.
Альтернативным вариантом может быть преобразование значений в символы и добавление в конце символа “%”, однако такой подход приведет к осложнениям, поскольку данные перестанут быть непрерывными числовыми значениями.
# Исходные пропорции оси y
#############################
linelist %>% # начать с построчного списка
group_by(hospital) %>% # сгруппировать данные по больницамl
summarise( # создать сводные столбцы
n = n(), # общее количество строк в группе
deaths = sum(outcome == "Death", na.rm=T), # количество смертельных исходов в группе
prop_death = deaths/n) %>% # доля смертельных исходов в группе
ggplot( # начать построение графика
mapping = aes(
x = hospital,
y = prop_death))+
geom_col()+
theme_minimal()+
labs(title = "Display y-axis original proportions")
# Отображение пропорций оси y в виде процентов
########################################
linelist %>%
group_by(hospital) %>%
summarise(
n = n(),
deaths = sum(outcome == "Death", na.rm=T),
prop_death = deaths/n) %>%
ggplot(
mapping = aes(
x = hospital,
y = prop_death))+
geom_col()+
theme_minimal()+
labs(title = "Display y-axis as percents (%)")+
scale_y_continuous(
labels = scales::percent # отображать пропорции в виде процентов
)Логарифмическая шкала
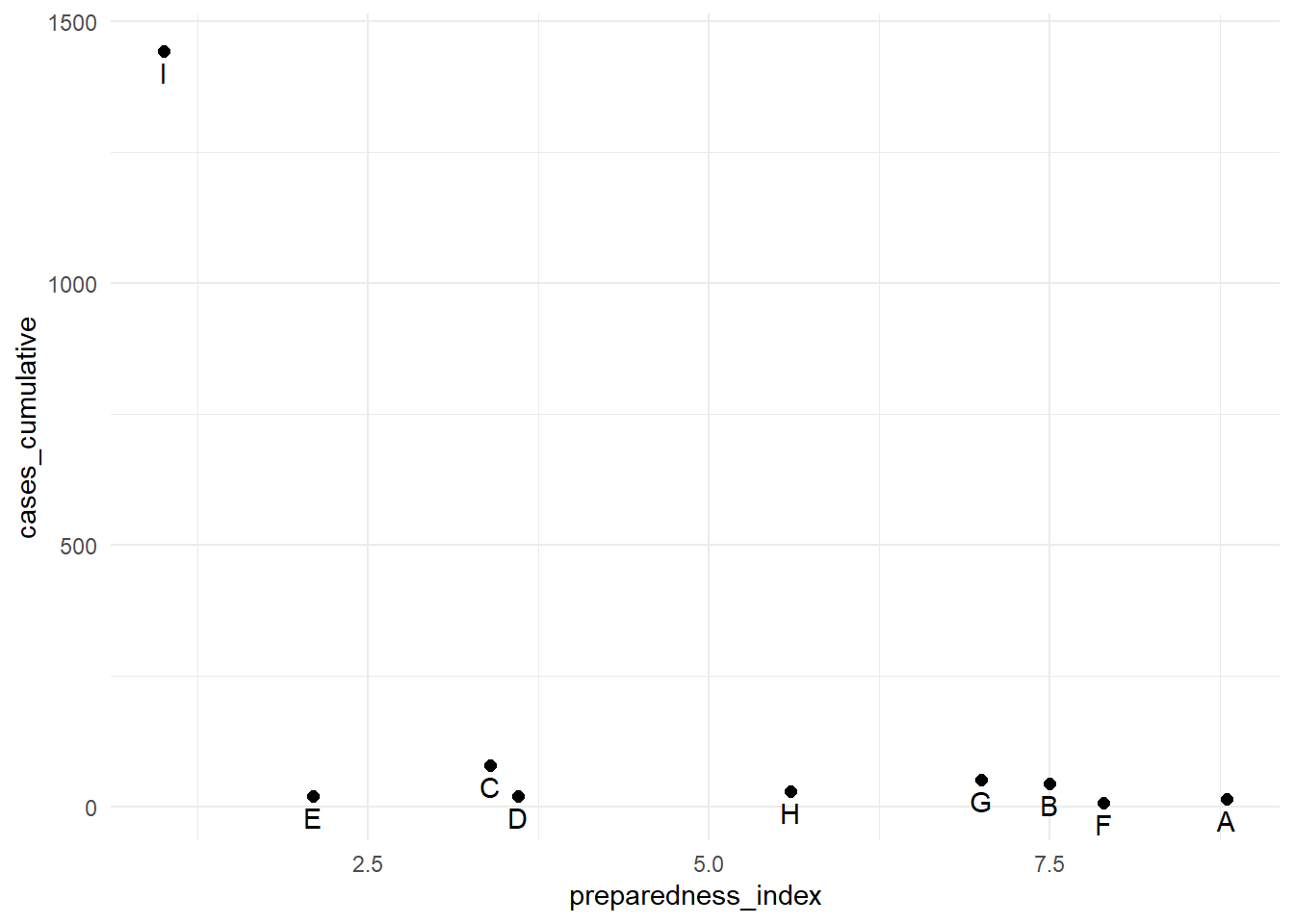
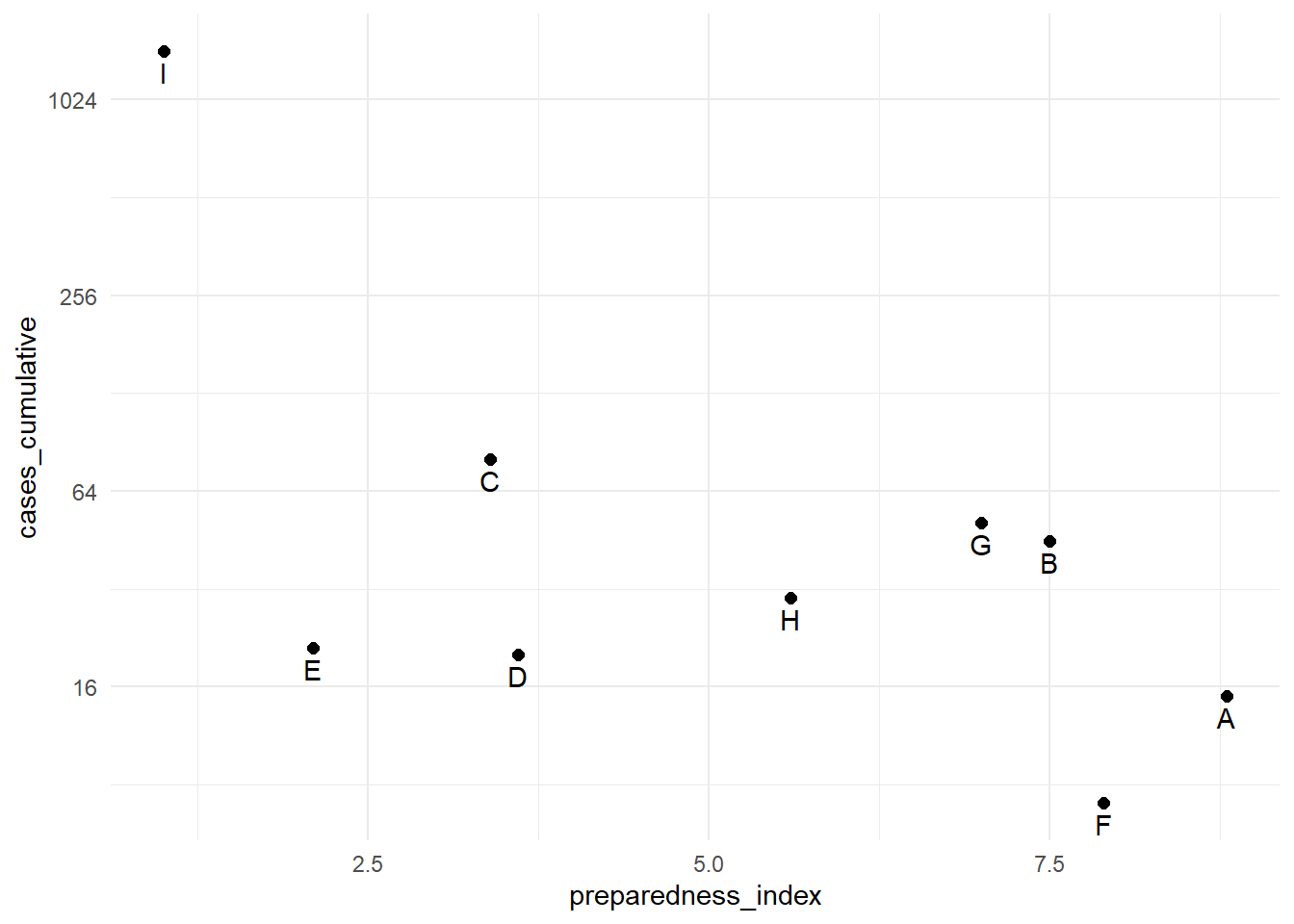
Чтобы преобразовать непрерывную ось в логарифмическую шкалу, добавьте к команде шкалы команду trans = "log2". Для примера создадим датафрейм регионов с соответствующими значениями preparedness_index и кумулятивных случаев.
plot_data <- data.frame(
region = c("A", "B", "C", "D", "E", "F", "G", "H", "I"),
preparedness_index = c(8.8, 7.5, 3.4, 3.6, 2.1, 7.9, 7.0, 5.6, 1.0),
cases_cumulative = c(15, 45, 80, 20, 21, 7, 51, 30, 1442)
)
plot_data region preparedness_index cases_cumulative
1 A 8.8 15
2 B 7.5 45
3 C 3.4 80
4 D 3.6 20
5 E 2.1 21
6 F 7.9 7
7 G 7.0 51
8 H 5.6 30
9 I 1.0 1442Кумулятивное число случаев в регионе “I” значительно превышает число случаев во всех других регионах. В подобных случаях можно использовать логарифмическую шкалу для отображения оси y, чтобы читатель мог увидеть различия между регионами с меньшим числом случаев.
# Исходная ось y
preparedness_plot <- ggplot(data = plot_data,
mapping = aes(
x = preparedness_index,
y = cases_cumulative))+
geom_point(size = 2)+ # точки для каждого региона
geom_text(
mapping = aes(label = region),
vjust = 1.5)+ # добавление текстовых меток
theme_minimal()
preparedness_plot # печать исходного графика
# печать с преобразованием оси y
preparedness_plot+ # начать с графика, сохраненного выше
scale_y_continuous(trans = "log2") # добавить преобразование для оси yГрадиентные шкалы
Шкалы градиента заливки могут иметь дополнительные нюансы. Как правило, значения по умолчанию вполне приемлемы, но вы можете захотеть подкорректировать значения, отсечения и т.д.
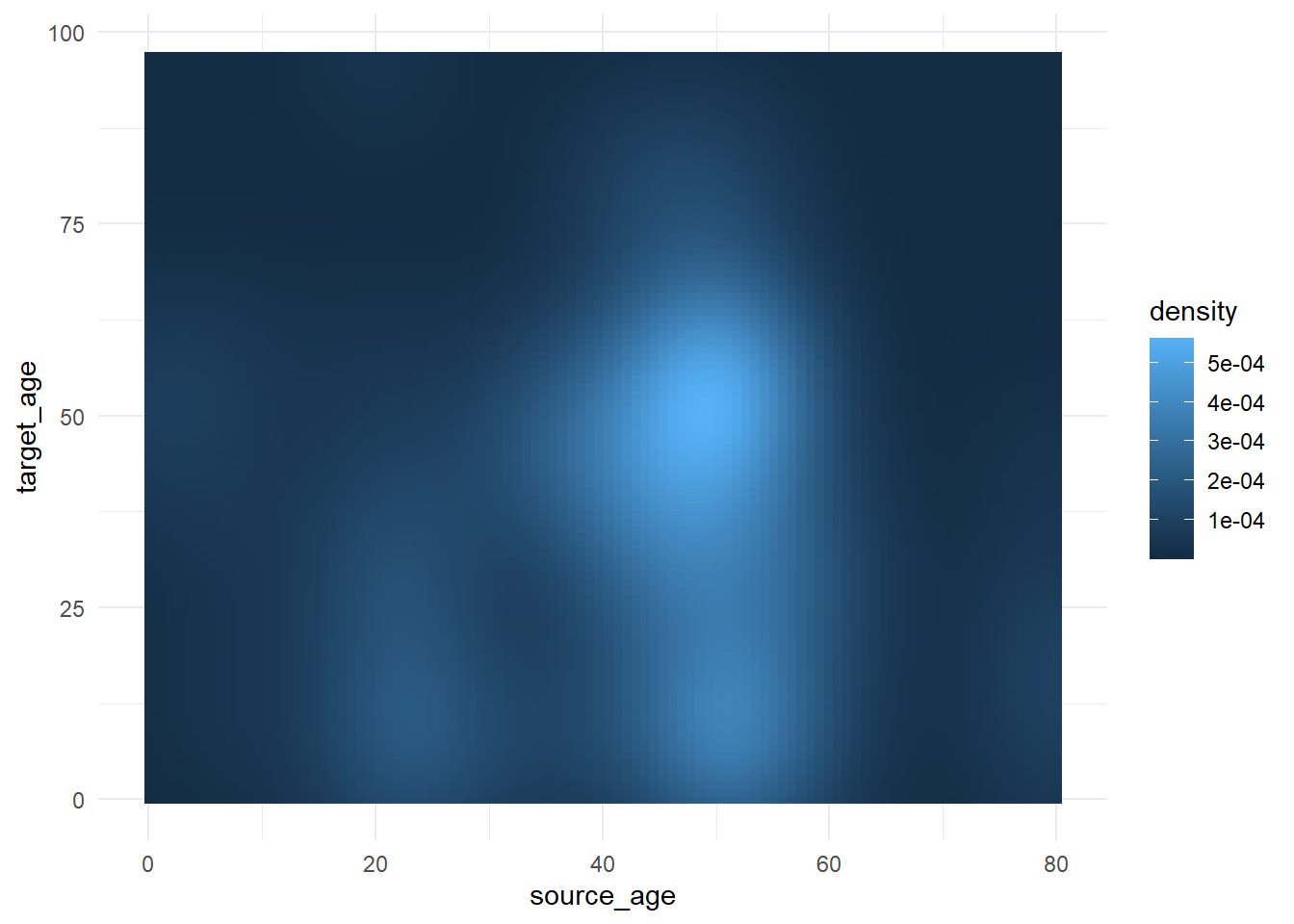
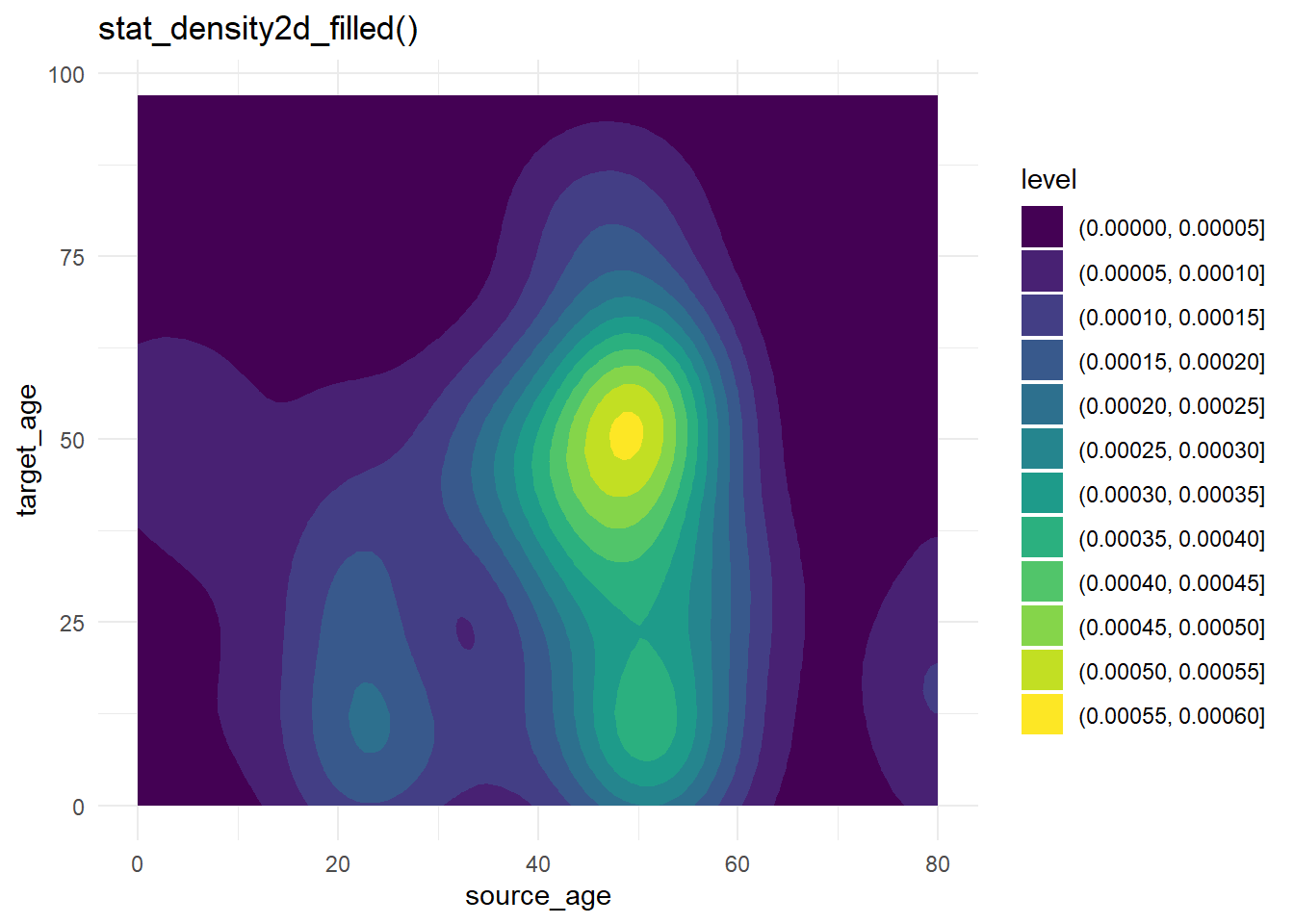
Для демонстрации настройки непрерывной цветовой шкалы мы воспользуемся набором данных со страницы [Отслеживание контактов], содержащим возраст случаев и их источников.
case_source_relationships <- rio::import(here::here("data", "godata", "relationships_clean.rds")) %>%
select(source_age, target_age) Ниже мы построим “растровый” график плотности тепловой плитки. Мы не будем подробно описывать, как это делается (см. ссылку в абзаце выше), а остановимся на том, как можно настроить цветовую шкалу. Подробнее о функции stat_density2d() ggplot2 можно прочитать здесь. Обратите внимание, что шкала fill является непрерывной.
trans_matrix <- ggplot(
data = case_source_relationships,
mapping = aes(x = source_age, y = target_age))+
stat_density2d(
geom = "raster",
mapping = aes(fill = after_stat(density)),
contour = FALSE)+
theme_minimal()Теперь мы покажем некоторые вариации шкалы заливки:
Теперь мы покажем несколько примеров настройки точек излома шкалы:
-
scale_fill_gradient()принимает два цвета (high/low)
-
scale_fill_gradientn()принимает вектор цветов произвольной длиныvalues =(промежуточные значения будут интерполированы)
- Используйте
scales::rescale()для настройки расположения цветов вдоль градиента; она изменяет вектор позиций на значения от 0 до 1.
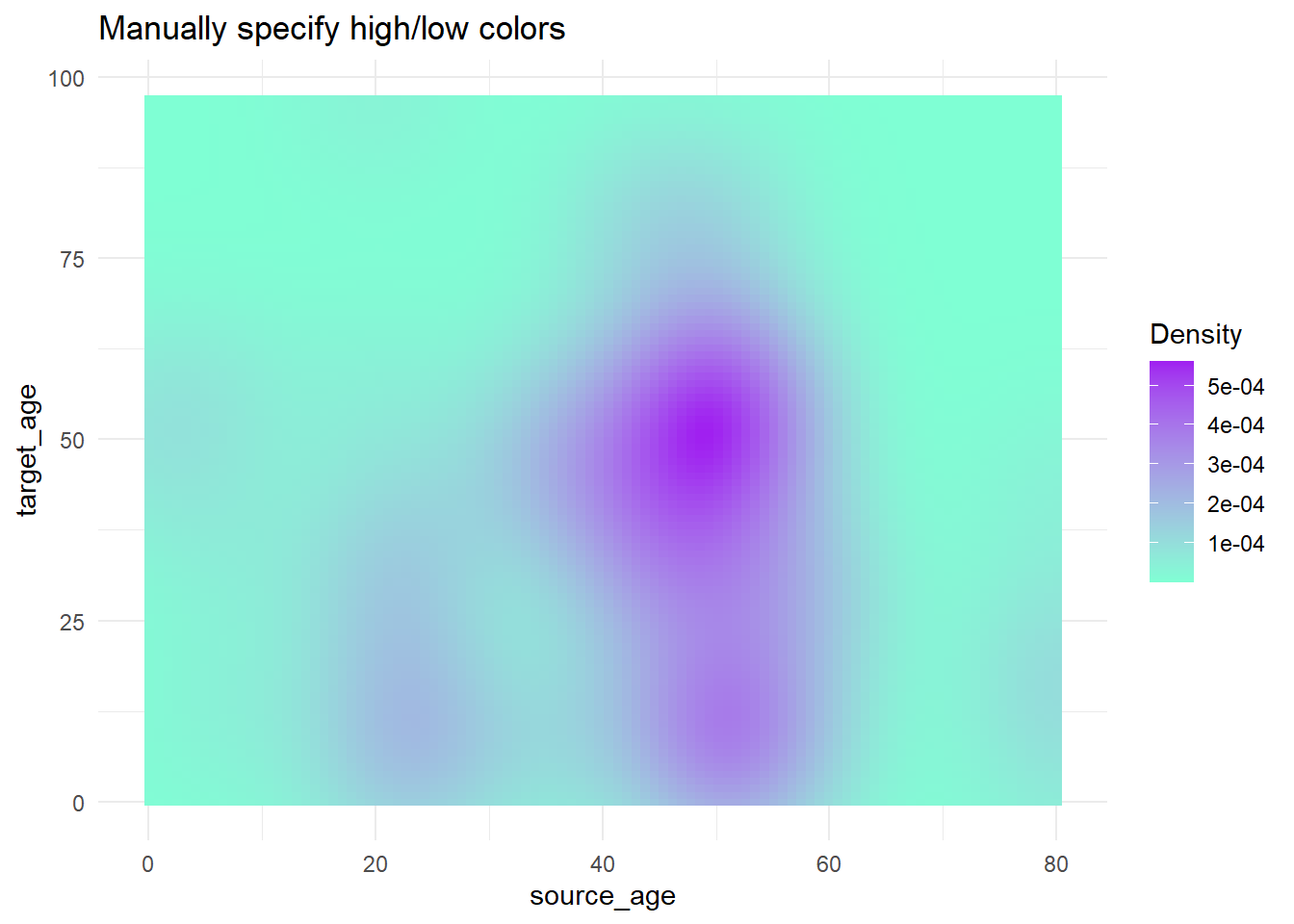
trans_matrix +
scale_fill_gradient( # Двусторонняя градиентная шкала
low = "aquamarine", # низкое значение
high = "purple", # высокое значение
na.value = "grey", # значение для NA
name = "Density")+ # Заголовок легенды
labs(title = "Manually specify high/low colors")
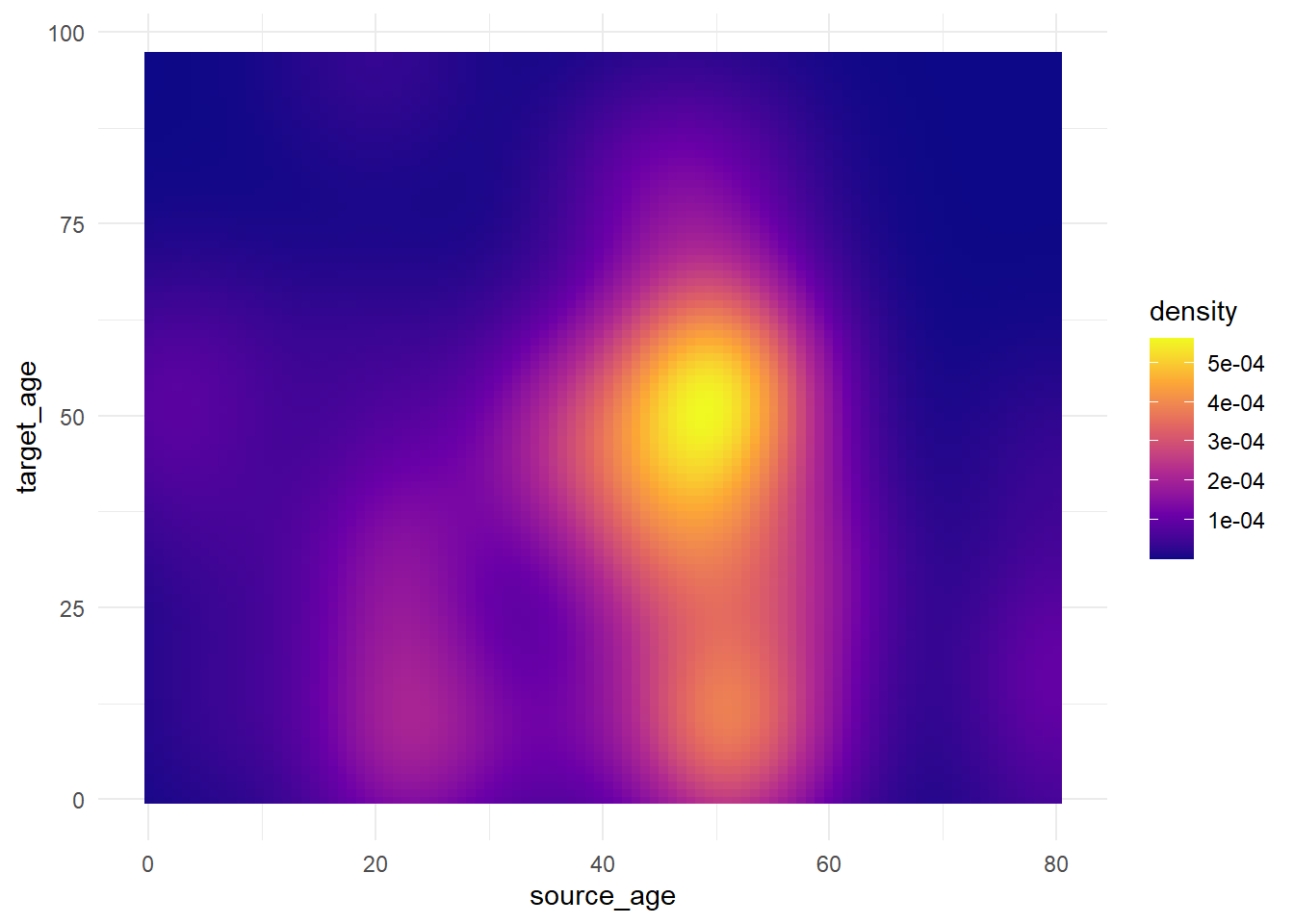
# 3+ цвета в шкале
trans_matrix +
scale_fill_gradientn( # 3-цветная шкала (низкий/средний/высокий уровень)
colors = c("blue", "yellow","red") # предоставить цвета в векторе
)+
labs(title = "3-color scale")
# Использование функции rescale() для изменения расположения цветов по шкале
trans_matrix +
scale_fill_gradientn( # задать любое количество цветов
colors = c("blue", "yellow","red", "black"),
values = scales::rescale(c(0, 0.05, 0.07, 0.10, 0.15, 0.20, 0.3, 0.5)) #позиции цветов изменяются в диапазоне от 0 до 1
)+
labs(title = "Colors not evenly positioned")
# использование ограничений для отсечения значений, получающих цвет заливки
trans_matrix +
scale_fill_gradientn(
colors = c("blue", "yellow","red"),
limits = c(0, 0.0002))+
labs(title = "Restrict value limits, resulting in grey space")Цветовые палитры
Colorbrewer и Viridis
В целом, если вам нужны предопределенные палитры, вы можете использовать функции scale_xxx_brewer или scale_xxx_viridis_y.
Функции ‘brewer’ могут рисовать из палитр colorbrewer.org.
Функции “viridis” основаны на палитрах “viridis” (с учетом особенностей зрения людей, не различающих цвета!), которые “обеспечивают цветовые карты, воспринимаемые одинаково как в цветном, так и в черно-белом варианте. Они также рассчитаны на восприятие пользователями с распространенными формами дальтонизма”. (подробнее здесь и здесь). Определите, является ли палитра дискретной, непрерывной или сгруппированной, указав это в конце функции (например, дискретная - scale_xxx_viridis_d).
Рекомендуется протестировать свой график в этом симуляторе цветовой слепоты. Если у вас красная/зеленая цветовая схема, попробуйте вместо нее использовать схему “горячий-холодный” (красно-синий), как описано здесь.
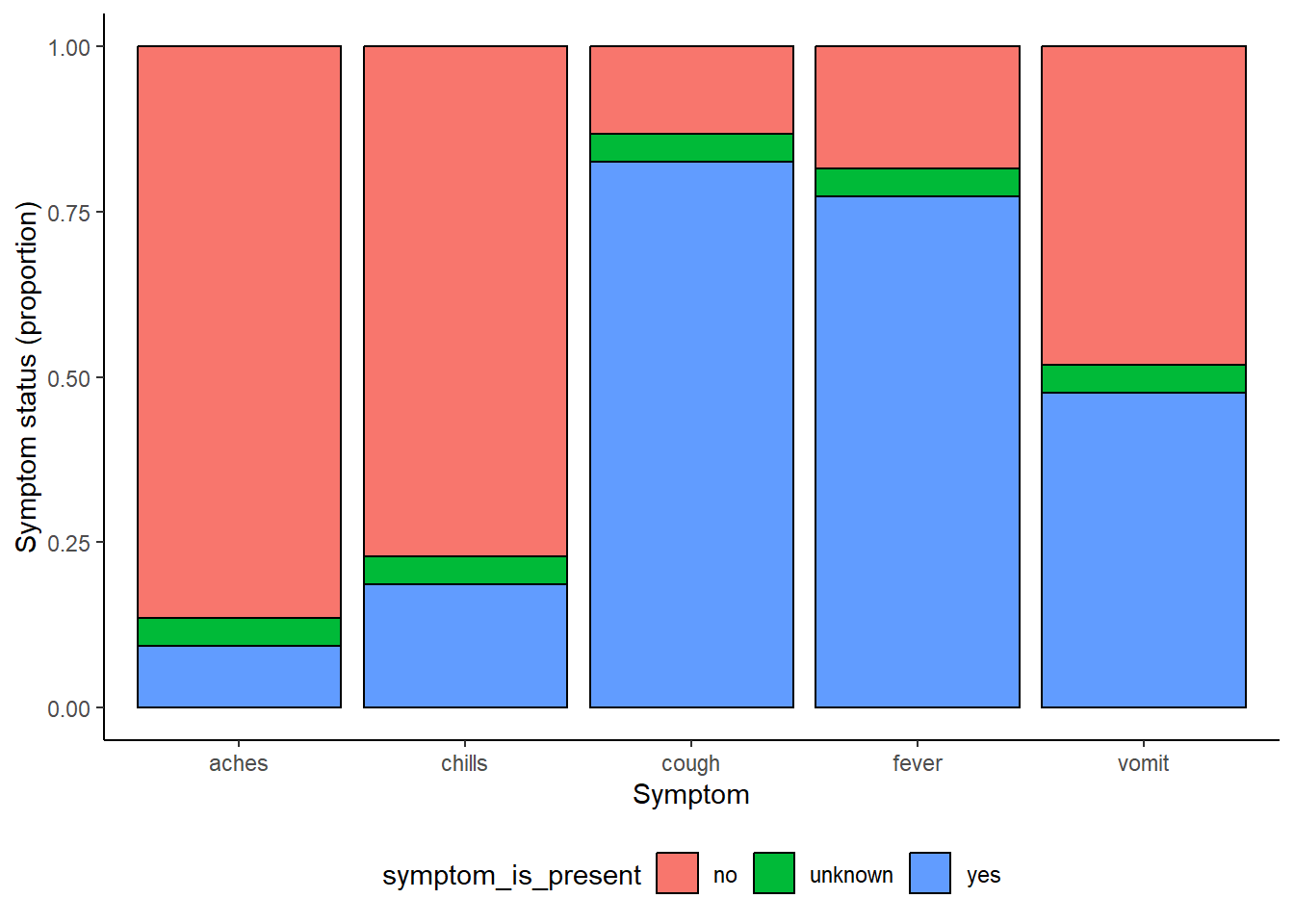
Вот пример со страницы [Основы ggplot] с использованием различных цветовых схем.
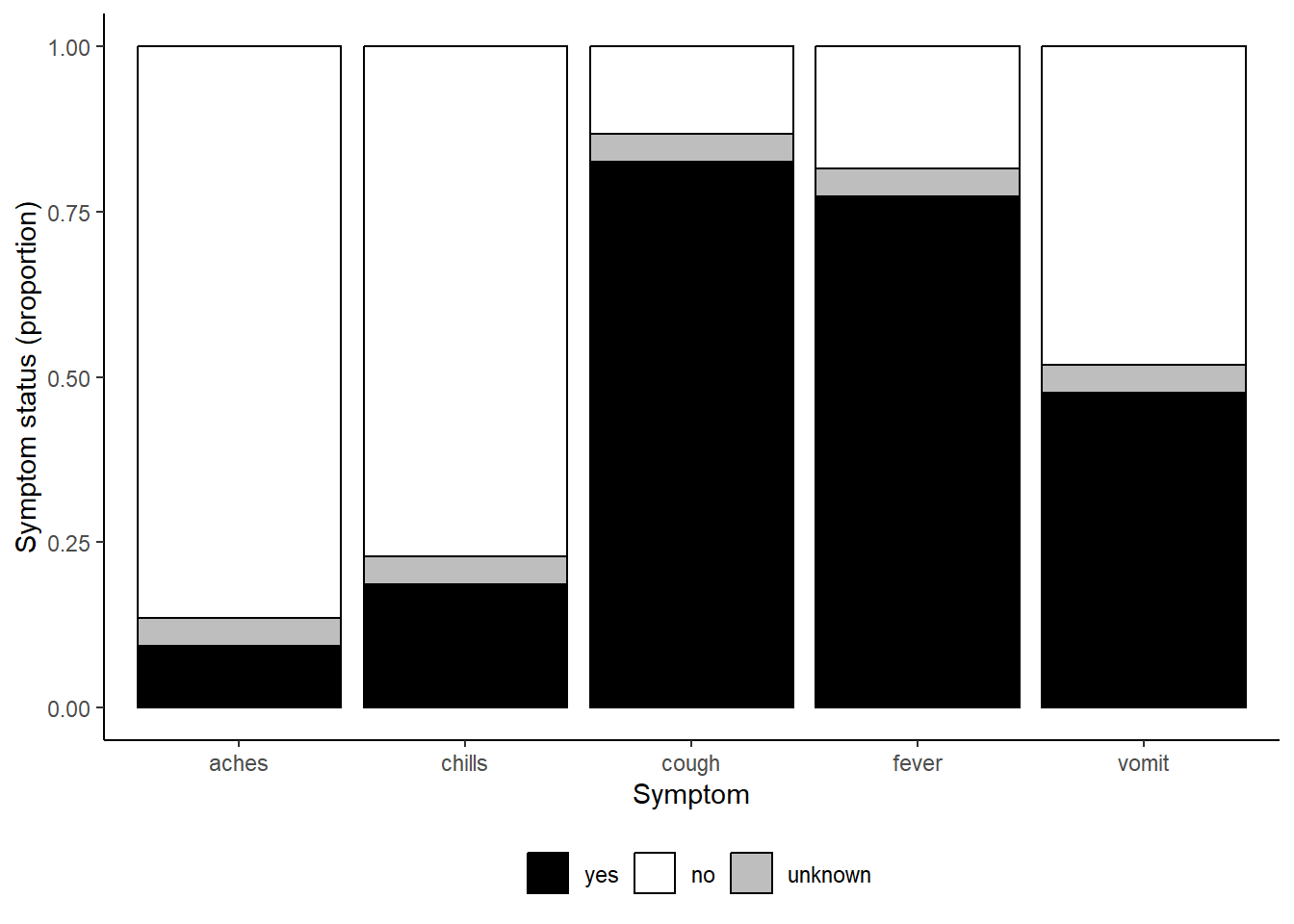
symp_plot <- linelist %>% # начать с построчного списка
select(c(case_id, fever, chills, cough, aches, vomit)) %>% # выбрать столбцы
pivot_longer( # повернуть вдлину
cols = -case_id,
names_to = "symptom_name",
values_to = "symptom_is_present") %>%
mutate( # заменить отсутствующие значения
symptom_is_present = replace_na(symptom_is_present, "unknown")) %>%
ggplot( # начать ggplot!
mapping = aes(x = symptom_name, fill = symptom_is_present))+
geom_bar(position = "fill", col = "black") +
theme_classic() +
theme(legend.position = "bottom")+
labs(
x = "Symptom",
y = "Symptom status (proportion)"
)
symp_plot # печать с цветами по умолчанию
#################################
# печать с заданными вручную цветами
symp_plot +
scale_fill_manual(
values = c("yes" = "black", # четко определить цвета
"no" = "white",
"unknown" = "grey"),
breaks = c("yes", "no", "unknown"), # правильно упорядочить факторы
name = "" # задать легенду без заголовка
)
#################################
# печать дискретными цветами viridis
symp_plot +
scale_fill_viridis_d(
breaks = c("yes", "no", "unknown"),
name = ""
)31.3 Изменение порядка дискретных переменных
Изменение порядка отображения дискретных переменных часто бывает трудно понять тем, кто впервые знакомится с графиками ggplot2. Однако понять, как это сделать, несложно, если разобраться в том, как ggplot2 работает с дискретными переменными. Вообще говоря, если используется дискретная переменная, то она автоматически преобразуется в тип фактор (factor), который по умолчанию упорядочивает факторы в алфавитном порядке. Чтобы справиться с этим, необходимо просто перестроить уровни факторов в соответствии с тем порядком, в котором они должны отображаться на графике. Более подробная информация о том, как переупорядочивать объекты типа factor, приведена в разделе руководства, посвященном факторам.
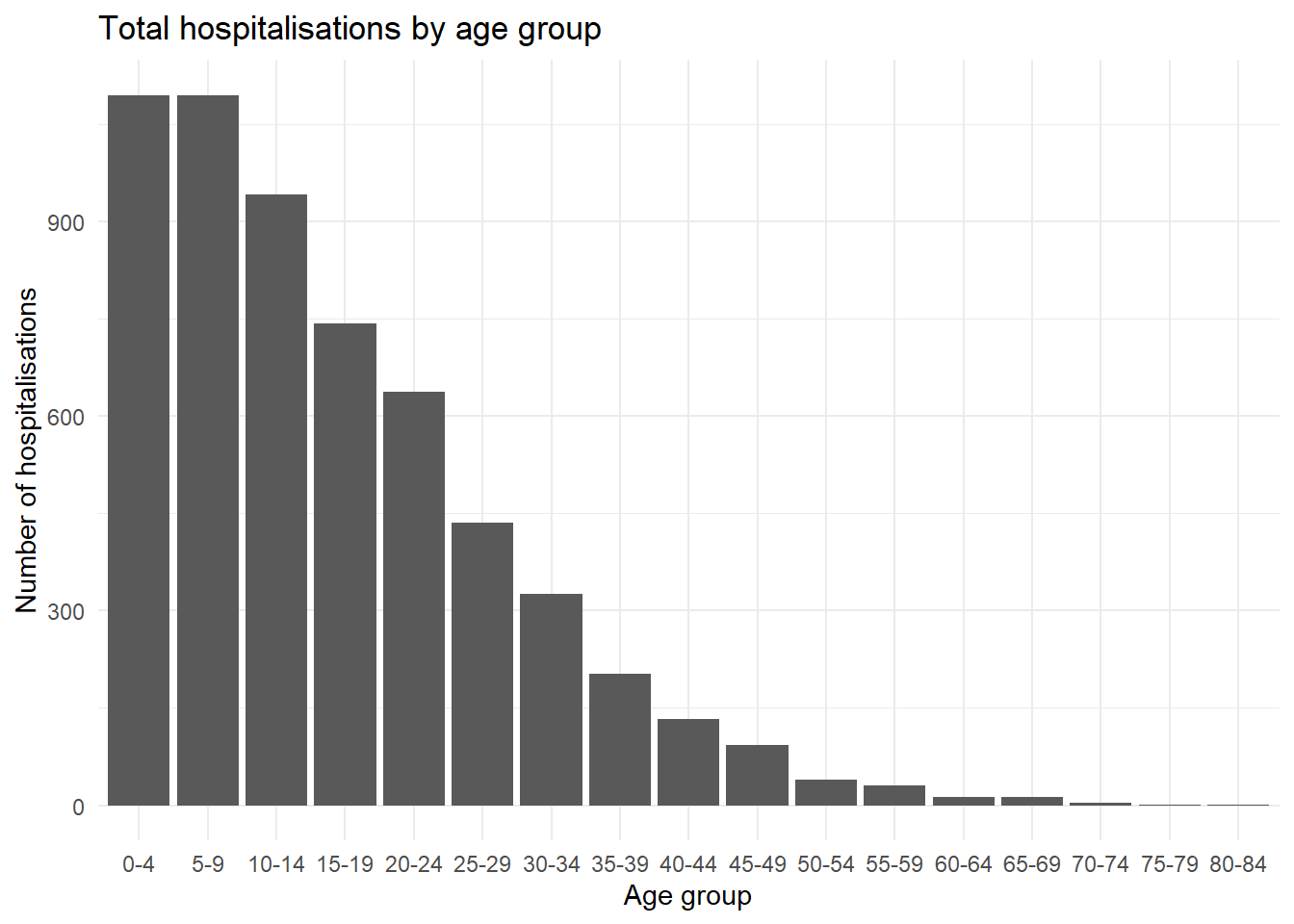
Мы можем рассмотреть распространенный пример с возрастными группами - по умолчанию возрастная группа 5-9 лет будет располагаться в середине возрастных групп (в алфавитно-цифровом порядке), но мы можем переместить ее за возрастную группу 0-4 на диаграмме, переупорядочив факторы.
ggplot(
data = linelist %>% drop_na(age_cat5), # удалить строки, в которых отсутствует age_cat5
mapping = aes(x = fct_relevel(age_cat5, "5-9", after = 1))) + # заново задать уровень фактора
geom_bar() +
labs(x = "Age group", y = "Number of hospitalisations",
title = "Total hospitalisations by age group") +
theme_minimal()31.3.0.1 ggthemr
Также можно рассмотреть возможность использования пакета ggthemr. Вы можете загрузить этот пакет с Github, используя инструкции здесь. Он предлагает очень эстетичные палитры, однако следует учитывать, что они обычно имеют максимальное количество значений, которое может быть ограничено, если вам нужно более 7 или 8 цветов.
31.4 Контурные линии
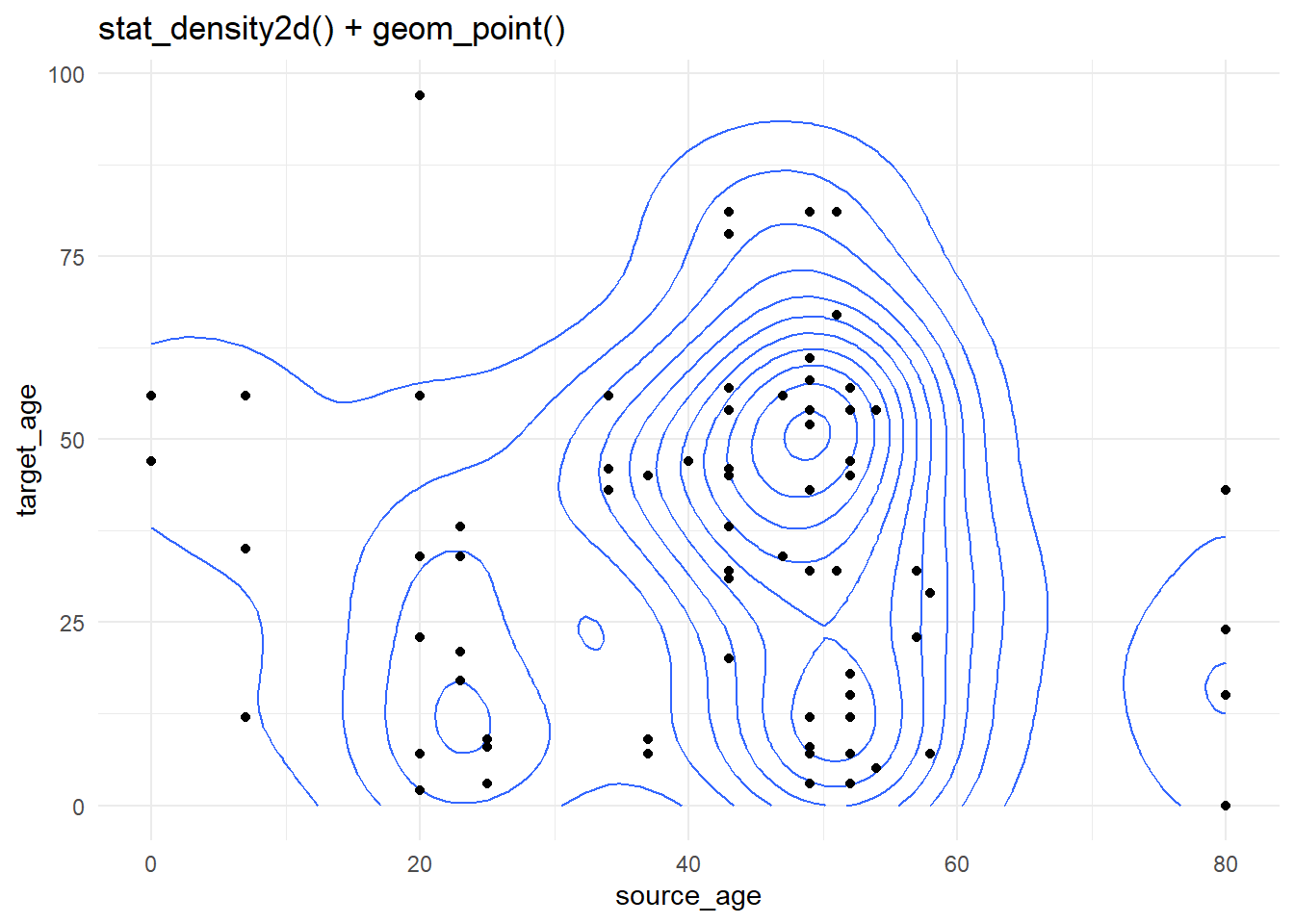
Контурные графики полезны при наличии большого количества точек, которые могут перекрывать друг друга (“наложение”). На примере исходных данных, использованных выше, снова строятся контурные графики, но более просто, с использованием stat_density2d() и stat_density2d_filled() для получения дискретных контурных уровней - как на топографической карте. Подробнее о статистике можно прочитать [здесь] (https://ggplot2.tidyverse.org/reference/geom_density_2d.html).
case_source_relationships %>%
ggplot(aes(x = source_age, y = target_age))+
stat_density2d()+
geom_point()+
theme_minimal()+
labs(title = "stat_density2d() + geom_point()")
case_source_relationships %>%
ggplot(aes(x = source_age, y = target_age))+
stat_density2d_filled()+
theme_minimal()+
labs(title = "stat_density2d_filled()")31.5 Предельные распределения
Для отображения распределений на краях графика рассеивания geom_point() можно использовать пакет ggExtra и его функцию ggMarginal(). Сохраните исходный ggplot как объект, а затем передайте его в ggMarginal(), как показано ниже. Вот основные аргументы:
- Вы должны указать
type =как ” гистограмма”, “плотность” “коробчатая диаграмма”, ” скрипичная диаграмма” или “денсиграмма”.
- По умолчанию для обеих осей отображаются предельные графики. Вы можете установить
margins =в значение “x” или “y”, если вам нужна только одна ось.
- Другие аргументы по выбору:
fill =(цвет столбиков),color =(цвет линий),Size =(размер графика относительно размера полей, поэтому большее число делает предельный график меньше).
- В качестве аргументов
xparams =иyparams =можно указывать другие аргументы, специфичные для конкретной оси. Например, чтобы иметь различные размеры корзин гистограммы, как показано ниже.
Вы можете сделать так, чтобы предельные графики отражали группы (столбцы, которым присвоен color = в эстетике отображения ggplot()). В этом случае установите аргумент ggMarginal() groupColour = или groupFill = в TRUE, как показано ниже.
Подробнее можно прочитать в разделе Эта виньетка, в галерее галерея R Graph или в документации по функции R ?ggMarginal.
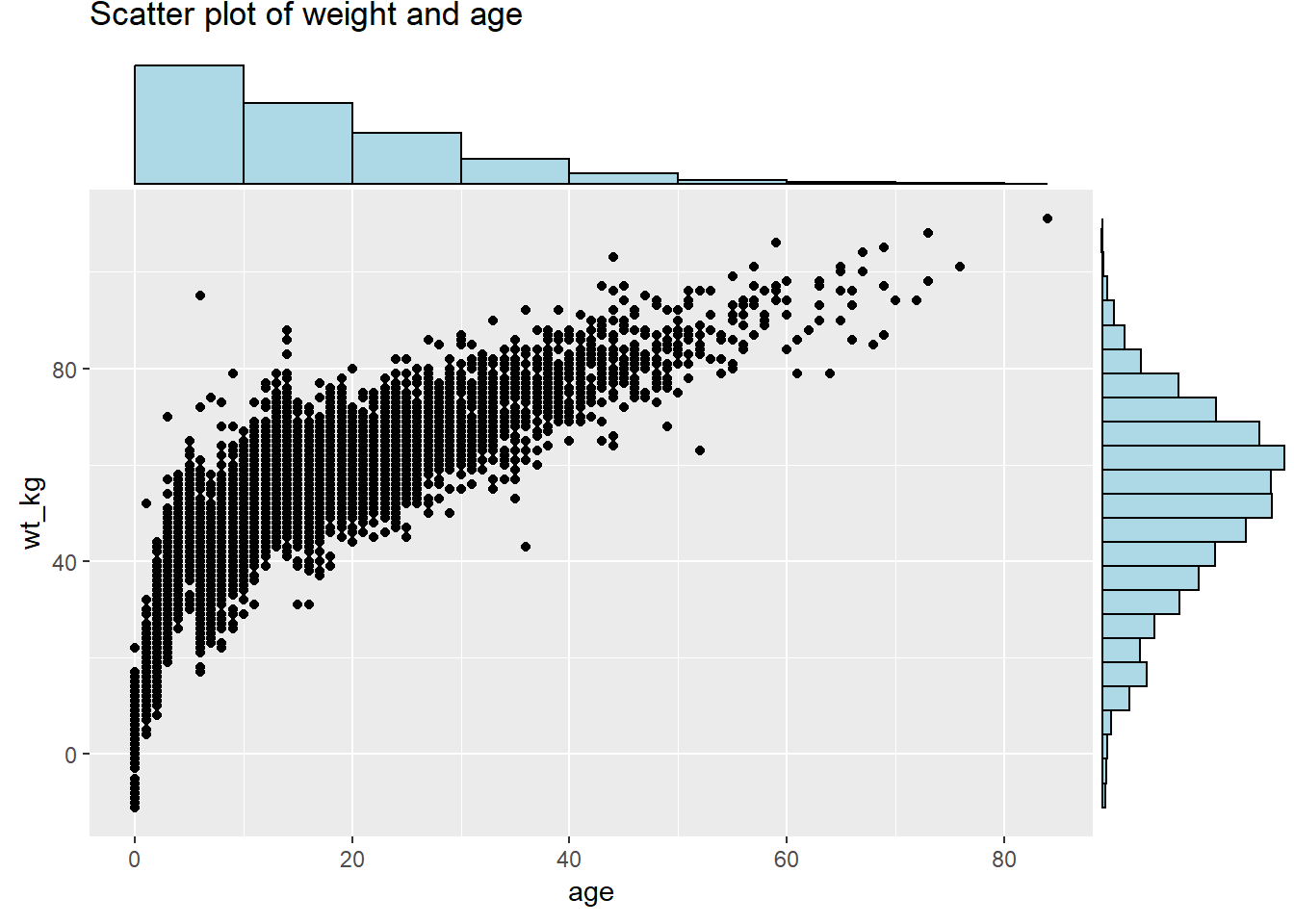
# Установка/загрузка ggExtra
pacman::p_load(ggExtra)
# Базовая диаграмма рассеивания веса и возраста
scatter_plot <- ggplot(data = linelist)+
geom_point(mapping = aes(y = wt_kg, x = age)) +
labs(title = "Scatter plot of weight and age")Для добавления предельных гистограмм используйте type = "histogram". Опционально можно задать groupFill = TRUE для получения сложенных гистограмм.
# с гистограммами
ggMarginal(
scatter_plot, # добавить предельные гистограммы
type = "histogram", # указать гистограммы
fill = "lightblue", # заливка столбика
xparams = list(binwidth = 10), # другие параметры для предельных значений по оси x
yparams = list(binwidth = 5)) # другие параметры для предельных значений по оси уГрафик предельной плотности с группированными/цветными значениями:
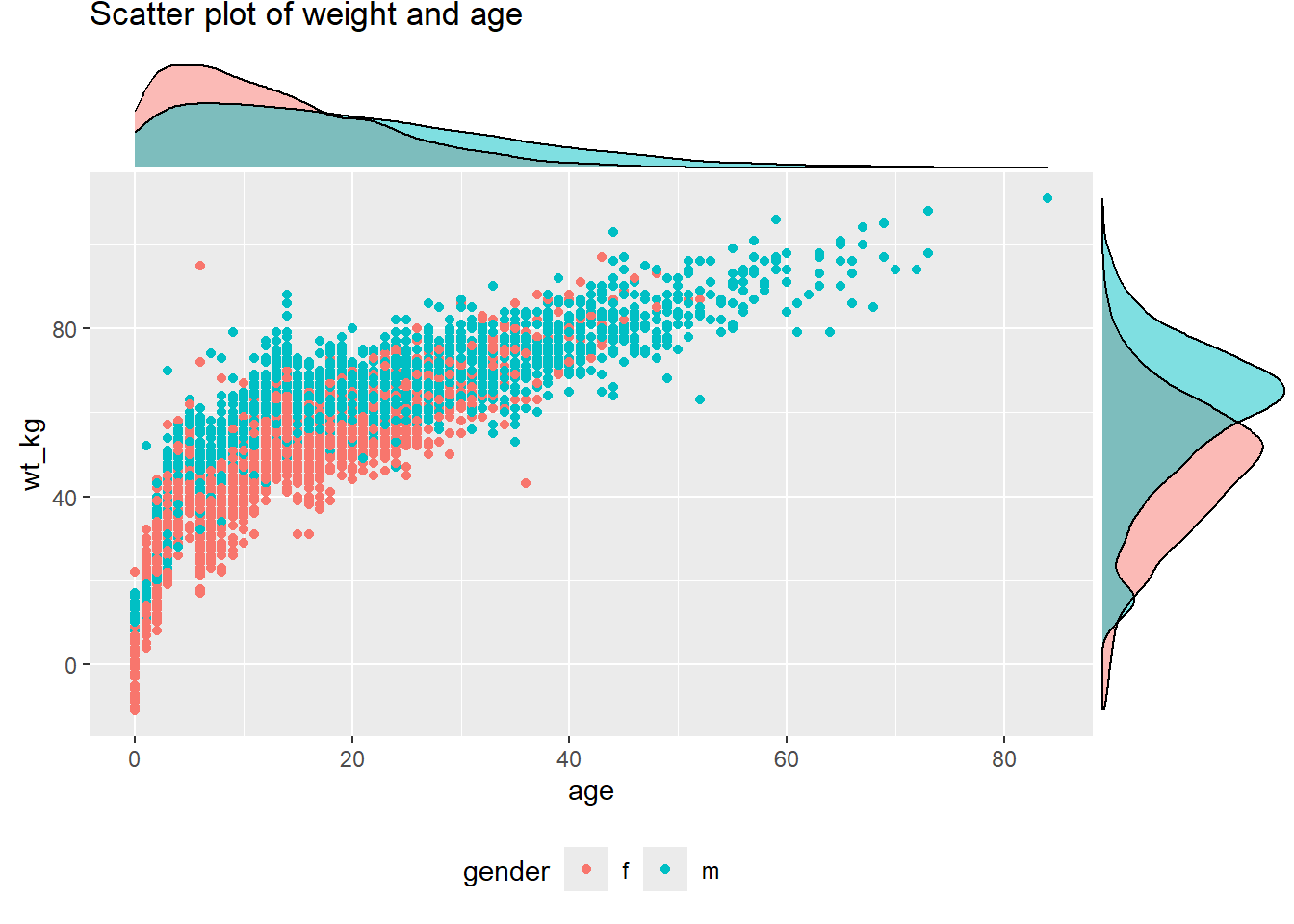
# График рассеивания, окрашенный по исходам
# Столбец исходов назначается цветом в ggplot. Значение groupFill в ggMarginal установлено как TRUE.
scatter_plot_color <- ggplot(data = linelist %>% drop_na(gender))+
geom_point(mapping = aes(y = wt_kg, x = age, color = gender)) +
labs(title = "Scatter plot of weight and age")+
theme(legend.position = "bottom")
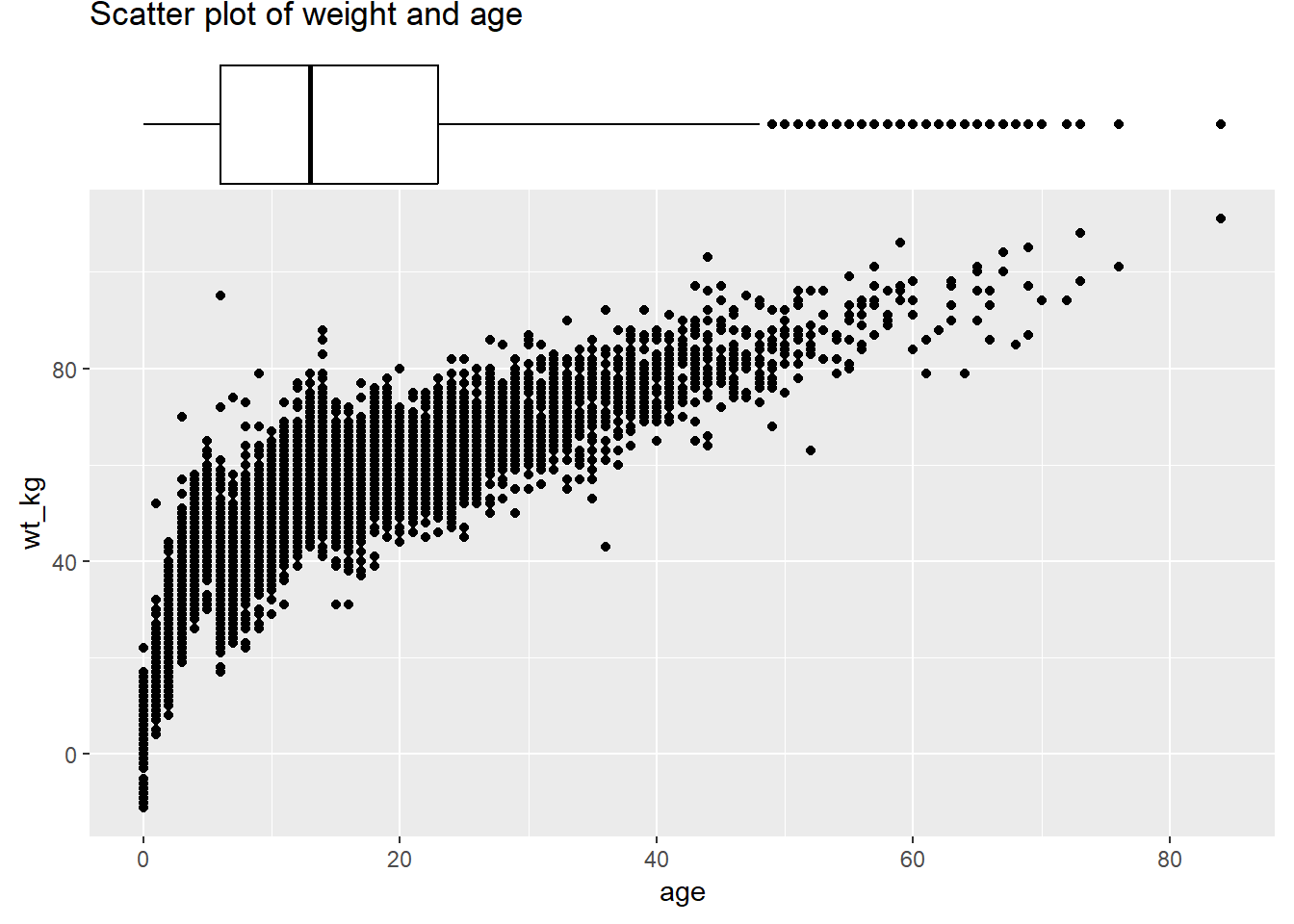
ggMarginal(scatter_plot_color, type = "density", groupFill = TRUE)Задайте аргумент size = для настройки относительного размера предельного графика. Меньшее число делает предельный график более крупным. Также можно задать color =. Ниже показан предельная коробчатая диаграмма, с демонстрацией аргумента margins =, чтобы она отображалась только на одной оси:
31.6 Умное наложение меток
В ggplot2 также возможно добавление текста на графики. Однако при этом возникает заметное ограничение: текстовые метки часто смешиваются с точками данных на графике, в результате чего они выглядят неаккуратно или плохо читаются. Идеального способа решения этой проблемы в базовом пакете нет, но существует дополнение ggplot2, известное как ggrepel, которое позволяет решить эту проблему очень просто!
Пакет ggrepel предлагает две новые функции, geom_label_repel() и geom_text_repel(), которые заменяют geom_label() и geom_text(). Просто используйте эти функции вместо базовых для получения аккуратных меток. Внутри функции, как обычно, используйте эстетику aes(), но включите аргумент label =, в котором укажите имя столбца, содержащего значения, которые вы хотите отобразить (например, идентификатор пациента, или имя, и т.д.). Можно создавать более сложные метки, комбинируя столбцы и новые строки (\n) в str_glue(), как показано ниже.
Несколько советов:
- Используйте
min.segment.length = 0, чтобы всегда рисовать сегменты линий, илиmin.segment.length = Inf, чтобы никогда их не рисовать
- Для задания размера текста используйте `
size =outside ofaes().
- Используйте
force =для изменения степени отталкивания между метками и соответствующими им точками (по умолчанию 1)
- Используйте
fill =outside ofaes()для окрашивания метки по значению.- В легенде может появиться буква “a” - добавьте
guides(fill = guide_legend(override.aes = aes(color = NA)))+для ее удаления
- В легенде может появиться буква “a” - добавьте
Подробнее об этом смотрите в этом подробном руководстве Обучение.
pacman::p_load(ggrepel)
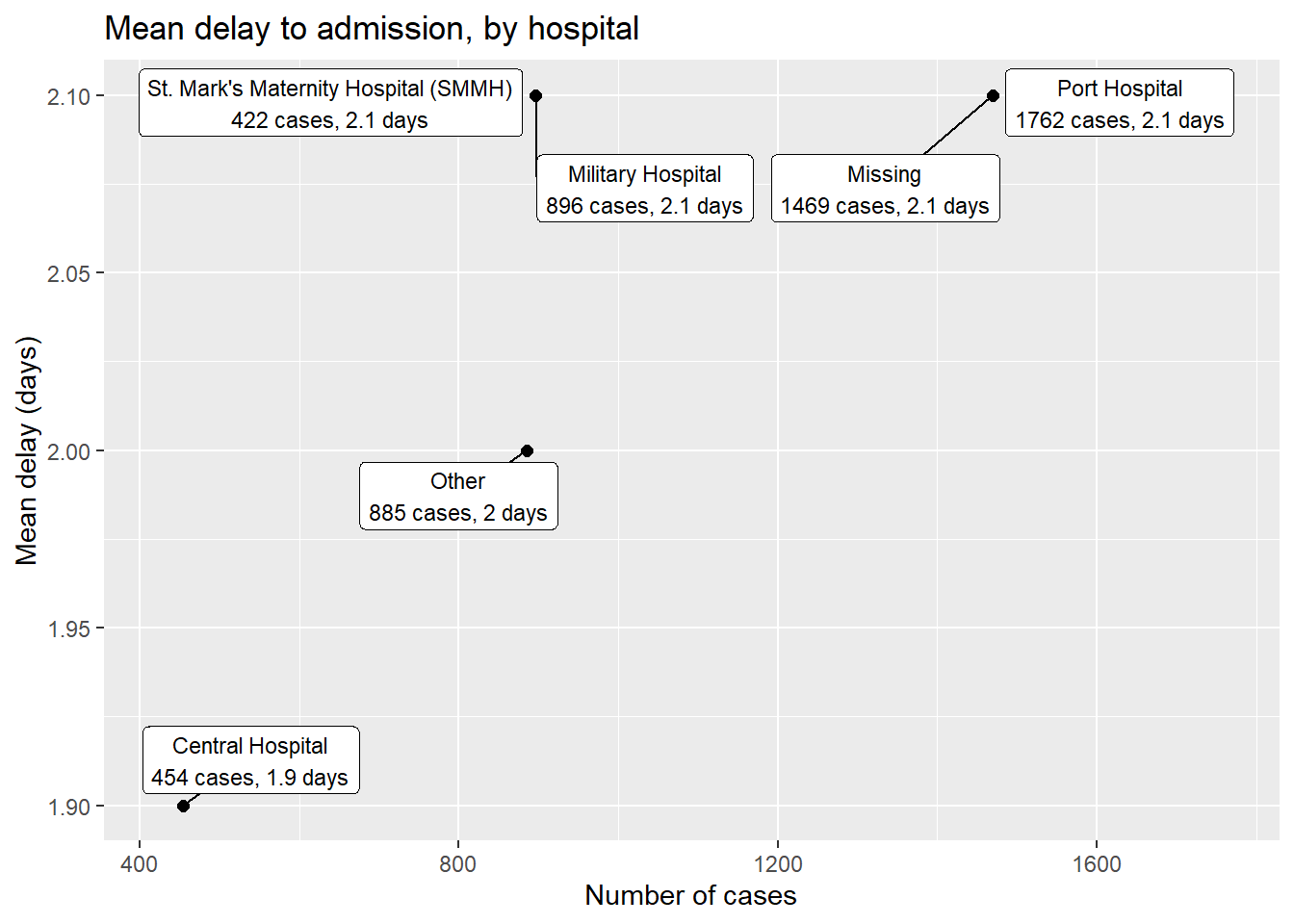
linelist %>% # начать с построчного списка
group_by(hospital) %>% # сгруппировать по больницам
summarise( # сформировать новый набор данных с сводными значениями по каждой больнице
n_cases = n(), # количество случаев на одну больницу
delay_mean = round(mean(days_onset_hosp, na.rm=T),1), # средняя задержка по больнице
) %>%
ggplot(mapping = aes(x = n_cases, y = delay_mean))+ # передать датафрейм в ggplot
geom_point(size = 2)+ # добавить точки
geom_label_repel( # добавить метки точек
mapping = aes(
label = stringr::str_glue(
"{hospital}\n{n_cases} cases, {delay_mean} days") # как отображаются метки
),
size = 3, # размер текста в метках
min.segment.length = 0)+ # показать все сегменты линии
labs( # добавить метки осей
title = "Mean delay to admission, by hospital",
x = "Number of cases",
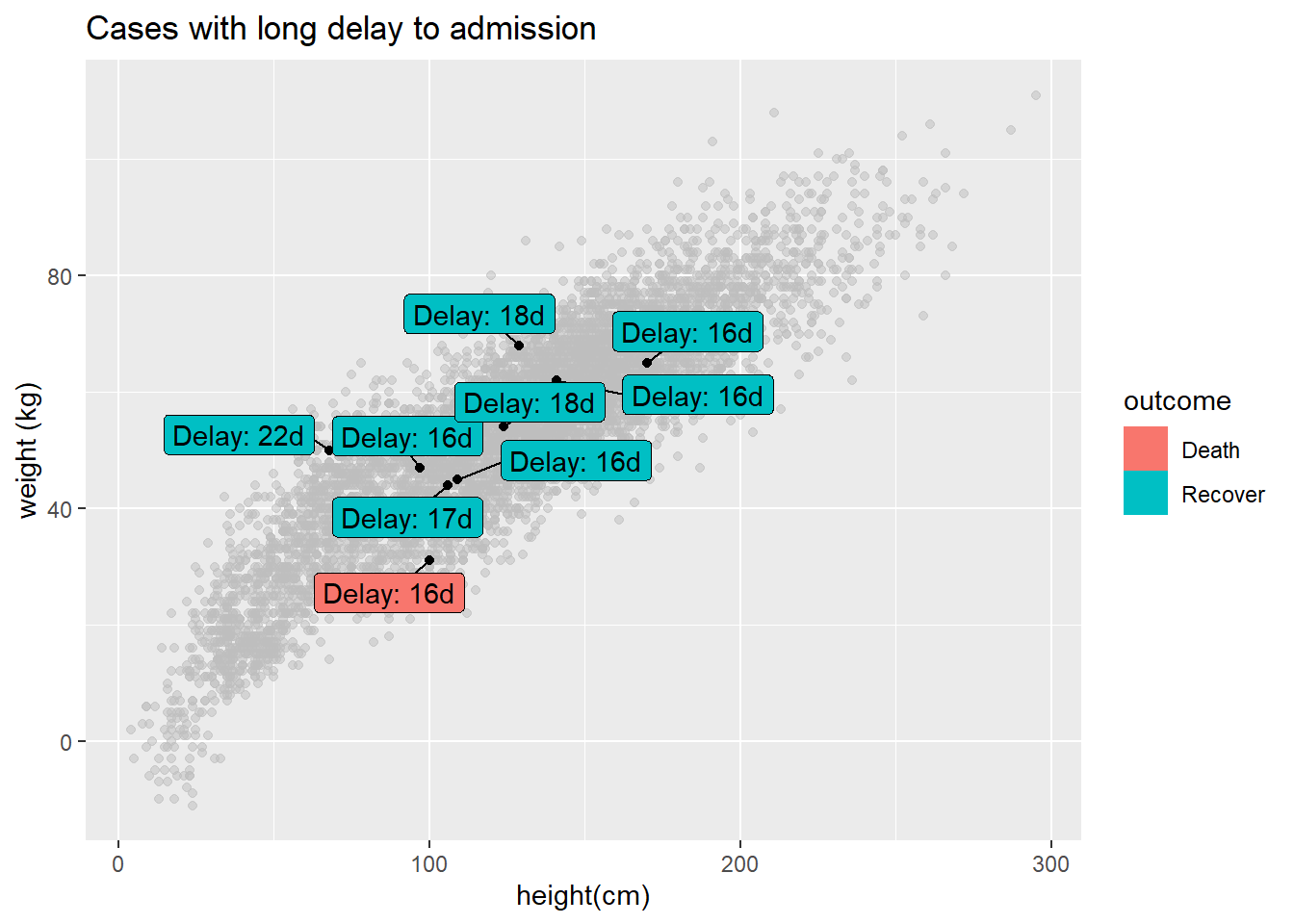
y = "Mean delay (days)")Вы можете пометить только подмножество точек данных - используя стандартный синтаксис ggplot(), чтобы обеспечить различные data = для каждого geom слоя графика. Ниже показаны все случаи, но помечены только некоторые из них.
ggplot()+
# Все точки выделены серым цветом
geom_point(
data = linelist, # все данные, представленные в этом слое
mapping = aes(x = ht_cm, y = wt_kg),
color = "grey",
alpha = 0.5)+ # серый и полупрозрачный
# Few points in black
geom_point(
data = linelist %>% filter(days_onset_hosp > 15), # фильтрованные данные, поступающие в этот слой
mapping = aes(x = ht_cm, y = wt_kg),
alpha = 1)+ # по умолчанию черный и не прозрачный
# point labels for few points
geom_label_repel(
data = linelist %>% filter(days_onset_hosp > 15), # отфильтровать данные для меток
mapping = aes(
x = ht_cm,
y = wt_kg,
fill = outcome, # обозначение цвета метками в зависимости от исхода
label = stringr::str_glue("Delay: {days_onset_hosp}d")), # метка, созданная с помощью функции str_glue()
min.segment.length = 0) + # показать сегменты линий для всех
# убрать букву "a" из внутренних полей легенды
guides(fill = guide_legend(override.aes = aes(color = NA)))+
# метки осей
labs(
title = "Cases with long delay to admission",
y = "weight (kg)",
x = "height(cm)")31.7 Оси времени
Работа с осями времени в ggplot может показаться сложной, но она очень проста благодаря нескольким ключевым функциям. Помните, что при работе с временем или датой необходимо убедиться, что нужные переменные отформатированы как класс дата или дата-время - примеры см. на странице [Работа с датами], а также на странице [Эпидемические кривые] (раздел ggplot).
Наиболее полезным набором функций для работы с датами в ggplot2 являются функции шкал (scale_x_date(), scale_x_datetime() и родственные им функции оси y). Эти функции позволяют определить частоту появления меток осей и способ форматирования меток осей. Чтобы узнать, как форматировать даты, обратитесь к разделу working with dates! С помощью аргументов date_breaks и date_labels можно указать, как должны выглядеть даты:
date_breaksпозволяет указать, как часто происходят разрывы оси - здесь можно указать последовательность (например,"3 месяца"или"2 дня")date_labelsпозволяет определить формат отображения дат. В эти аргументы можно передать строку формата даты (например,"%b-%d-%Y"):
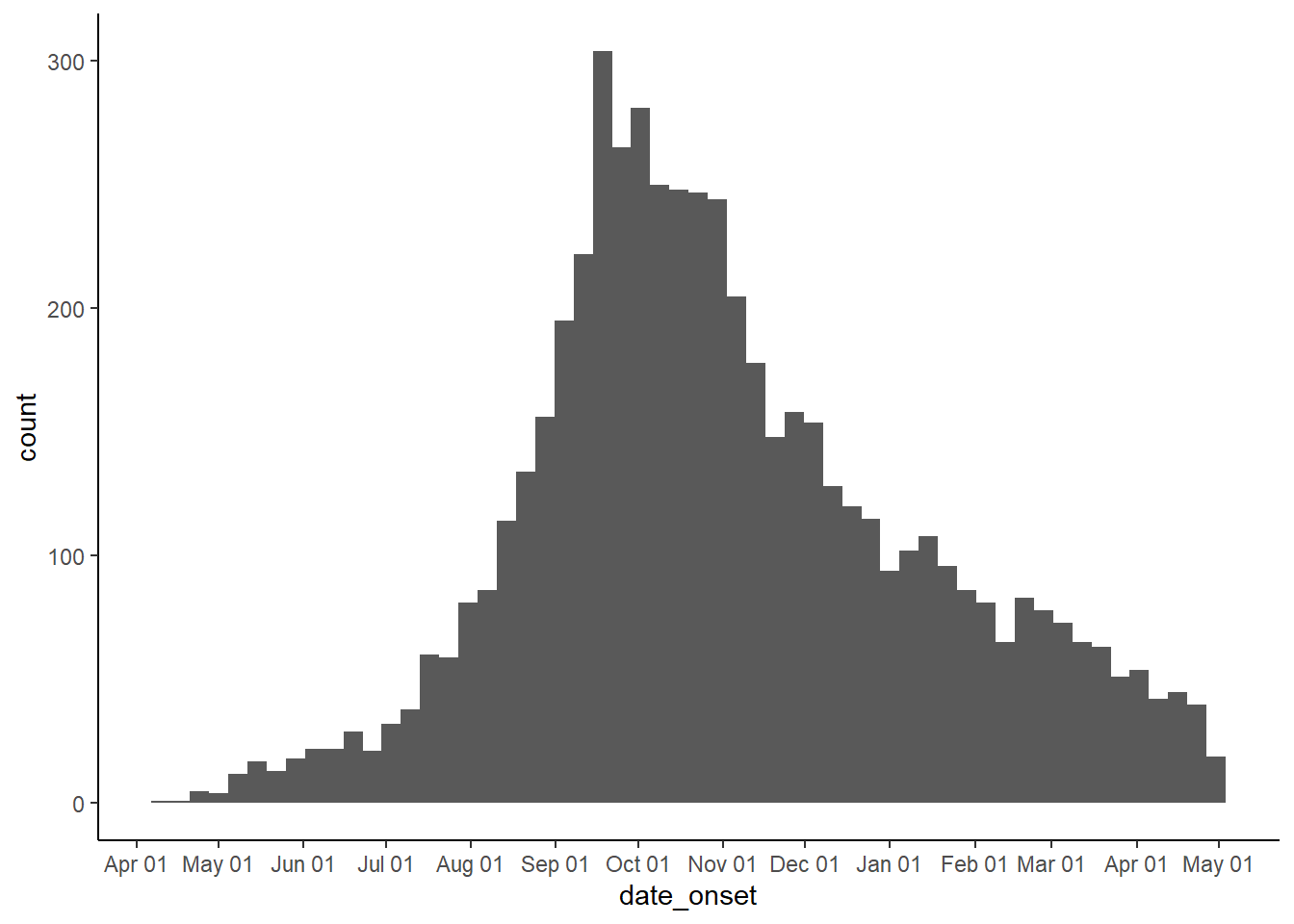
# построить эпидемическую кривую по дате начала заболевания, если это возможно
ggplot(linelist, aes(x = date_onset)) +
geom_histogram(binwidth = 7) +
scale_x_date(
# 1 разрыв каждый 1 месяц
date_breaks = "1 months",
# На метках должен отображаться месяц, а затем дата
date_labels = "%b %d"
) +
theme_classic()Одним из простых решений для получения эффективных меток даты на оси x является присвоение аргумента labels = в scale_x_date() функции label_date_short() из пакета scales. Эта функция автоматически построит эффективные метки даты (подробнее здесь). Дополнительным преимуществом этой функции является то, что метки будут автоматически корректироваться по мере увеличения объема данных во времени: от дней, недель, месяцев и лет.
Полный пример можно посмотреть в разделе страницы Эпидемические кривые, посвященном вопросу многоуровневые метки дат, а для справки ниже приведен краткий пример:
31.8 Выделение
Выделение определенных элементов на графике - полезный способ привлечь внимание к конкретному элементу переменной и одновременно предоставить информацию о дисперсии всего набора данных. Хотя это нелегко сделать в базовом ggplot2, существует внешний пакет gghighlight, который может помочь в этом. Его легко использовать в рамках синтаксиса ggplot.
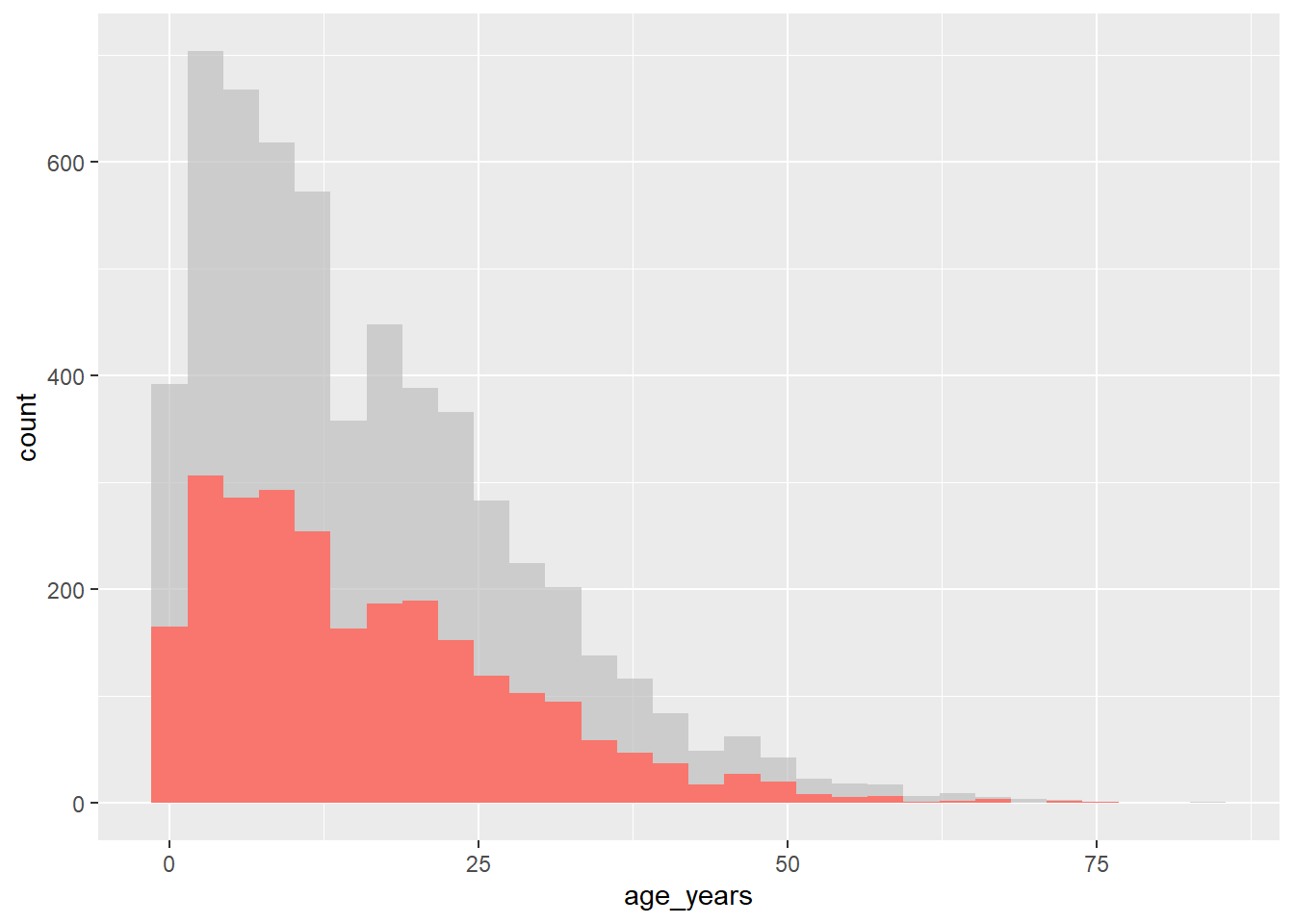
Для достижения этого эффекта пакет gghighlight использует функцию gghighlight(). Для использования этой функции необходимо задать ей логический оператор - это может привести к довольно гибким результатам, но здесь мы покажем пример распределения случаев по возрасту в нашем построчном списке, выделив их по исходу.
# загрузка gghighlight
library(gghighlight)
# заменить значения NA на неизвестные в переменной исхода
linelist <- linelist %>%
mutate(outcome = replace_na(outcome, "Unknown"))
# построить гистограмму всех случаев по возрасту
ggplot(
data = linelist,
mapping = aes(x = age_years, fill = outcome)) +
geom_histogram() +
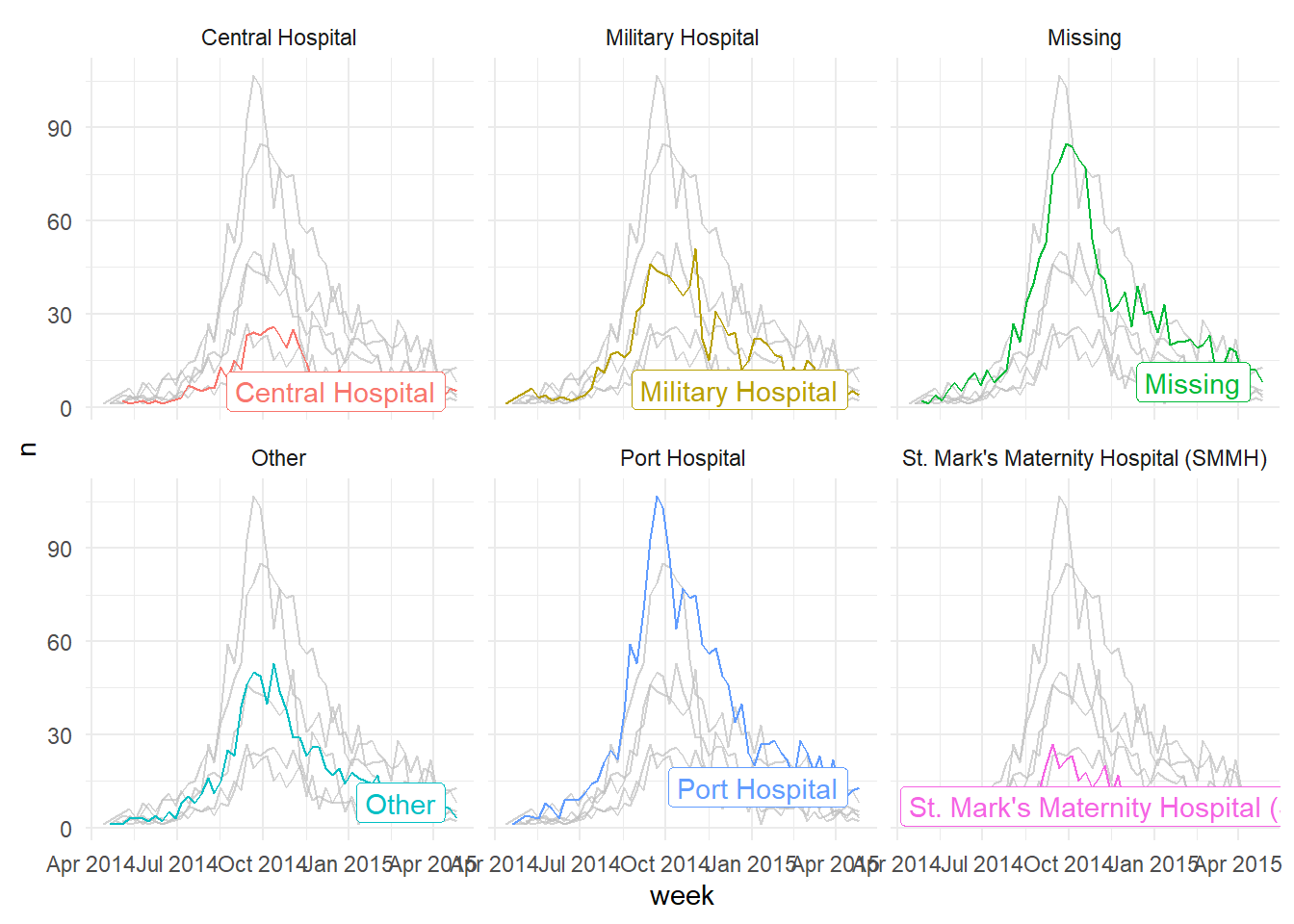
gghighlight::gghighlight(outcome == "Death") # выделить случаи, когда пациент умерЭто также хорошо работает с функциями фасетирования - позволяет строить графики фасетов с выделением фоновых данных, не относящихся к фасету! Ниже приведен подсчет случаев заболевания по неделям и построение эпидемических кривых по больницам (color = и facet_wrap() установлен на столбец hospital).
# построить гистограмму всех случаев по возрасту
linelist %>%
count(week = lubridate::floor_date(date_hospitalisation, "week"),
hospital) %>%
ggplot()+
geom_line(aes(x = week, y = n, color = hospital))+
theme_minimal()+
gghighlight::gghighlight() + # выделить случаи, когда пациент умер
facet_wrap(~hospital) # создавать фасеты по исходам31.9 Построение графиков для нескольких наборов данных
Обратите внимание на то, что правильное выравнивание осей для построения графиков из нескольких наборов данных в одном графике может быть затруднено. Рассмотрим одну из следующих стратегий:
- Объединить данные перед построением графика и преобразовать их в “длинный” формат со столбцом, отражающим набор данных
- Использовать cowplot или аналогичный пакет для объединения двух графиков (см. ниже)
31.10 Объединение графиков
Два пакета, которые очень полезны для объединения графиков, - это cowplot и patchwork. На этой странице мы будем рассматривать в основном cowplot, изредка используя patchwork.
Здесь находится онлайн введение в cowplot. Более подробную документацию по каждой функции можно прочитать здесь. Ниже мы рассмотрим несколько наиболее распространенных вариантов использования и функций.
Пакет cowplot работает в паре с ggplot2 - по сути, с его помощью вы упорядочиваете и объединяете ggplots и их легенды в составные фигуры. Он также может работать с базовыми графиками R.
pacman::p_load(
tidyverse, # работа с данными и их визуализация
cowplot, # объединение графиков
patchwork # объединение графиков
)Хотя фасет (описанный на странице [Основы ggplot]) является удобным подходом к построению графиков, иногда невозможно получить желаемые результаты с помощью его относительно ограниченного подхода. В этом случае можно объединить графики, склеив их в более крупный график. Есть три известных пакета, которые отлично подходят для этого - cowplot, gridExtra и patchwork. Однако эти пакеты выполняют в основном одни и те же задачи, поэтому в данном разделе мы остановимся на cowplot.
plot_grid()
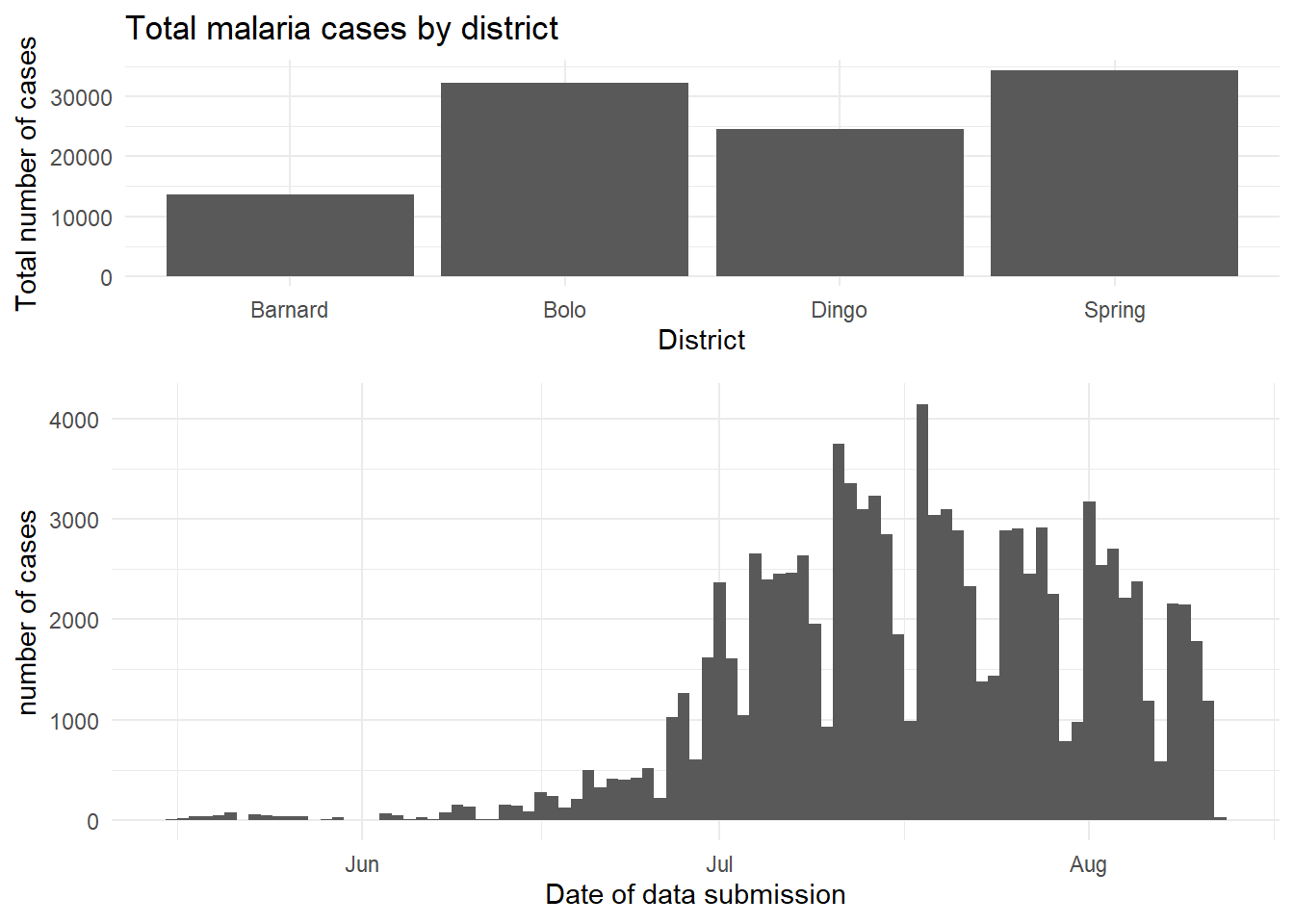
Пакет cowplot имеет достаточно широкий набор функций, но наиболее простое его использование достигается с помощью plot_grid(). Фактически это способ расположить предопределенные графики в виде сетки. Мы можем рассмотреть еще один пример с набором данных по малярии - здесь мы можем построить график общего количества случаев заболевания по районам, а также показать эпидемическую кривую в динамике.
malaria_data <- rio::import(here::here("data", "malaria_facility_count_data.rds"))
# столбчатая диаграмма общего количества случаев по районам
p1 <- ggplot(malaria_data, aes(x = District, y = malaria_tot)) +
geom_bar(stat = "identity") +
labs(
x = "District",
y = "Total number of cases",
title = "Total malaria cases by district"
) +
theme_minimal()
# эпидемическая кривая во времени
p2 <- ggplot(malaria_data, aes(x = data_date, y = malaria_tot)) +
geom_col(width = 1) +
labs(
x = "Date of data submission",
y = "number of cases"
) +
theme_minimal()
cowplot::plot_grid(p1, p2,
# 1 столбец и две строки - уложенные друг на друга
ncol = 1,
nrow = 2,
# верхний график на 2/3 выше второго
rel_heights = c(2, 3))Объединение легенд
Если графики имеют одинаковую легенду, их объединение не представляет сложности. Достаточно использовать описанный выше подход cowplot для объединения графиков, но при этом удалить легенду одного из них (удалить дублирование).
Если же графики имеют разные легенды, то необходимо использовать альтернативный подход:
- Создайте и сохраните графики без легенд, используя
theme(legend.position = "none").
- Извлеките легенды из каждого графика, используя
get_legend(), как показано ниже - но извлекайте легенды из графиков, видоизмененных так, чтобы они действительно показывали легенду.
- Объедините легенды в панель легенд
- Объединить графики и панель легенд
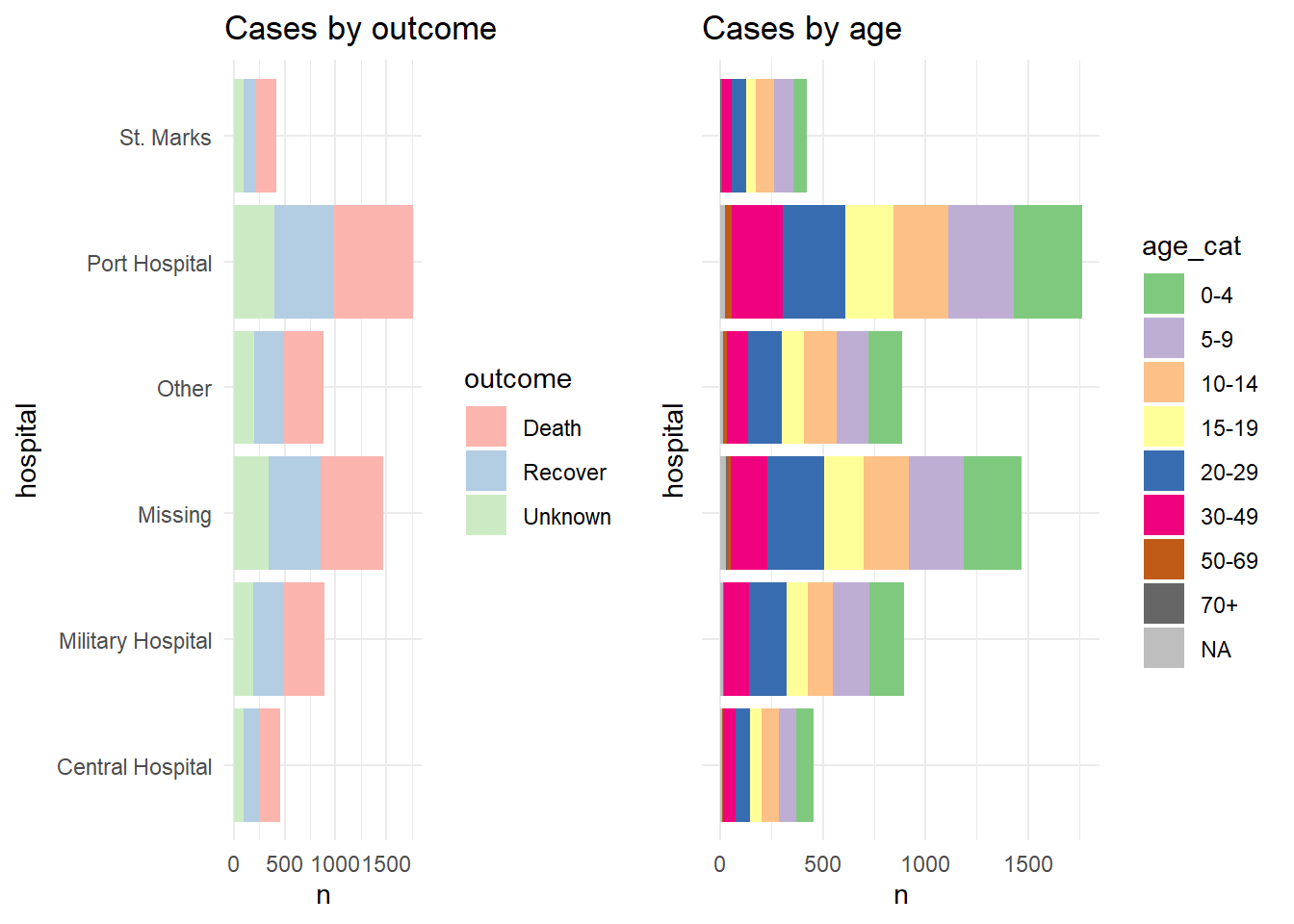
Для демонстрации мы показываем два графика отдельно, а затем располагаем их в сетке с указанием собственных легенд (некрасивое и неэффективное использование пространства):
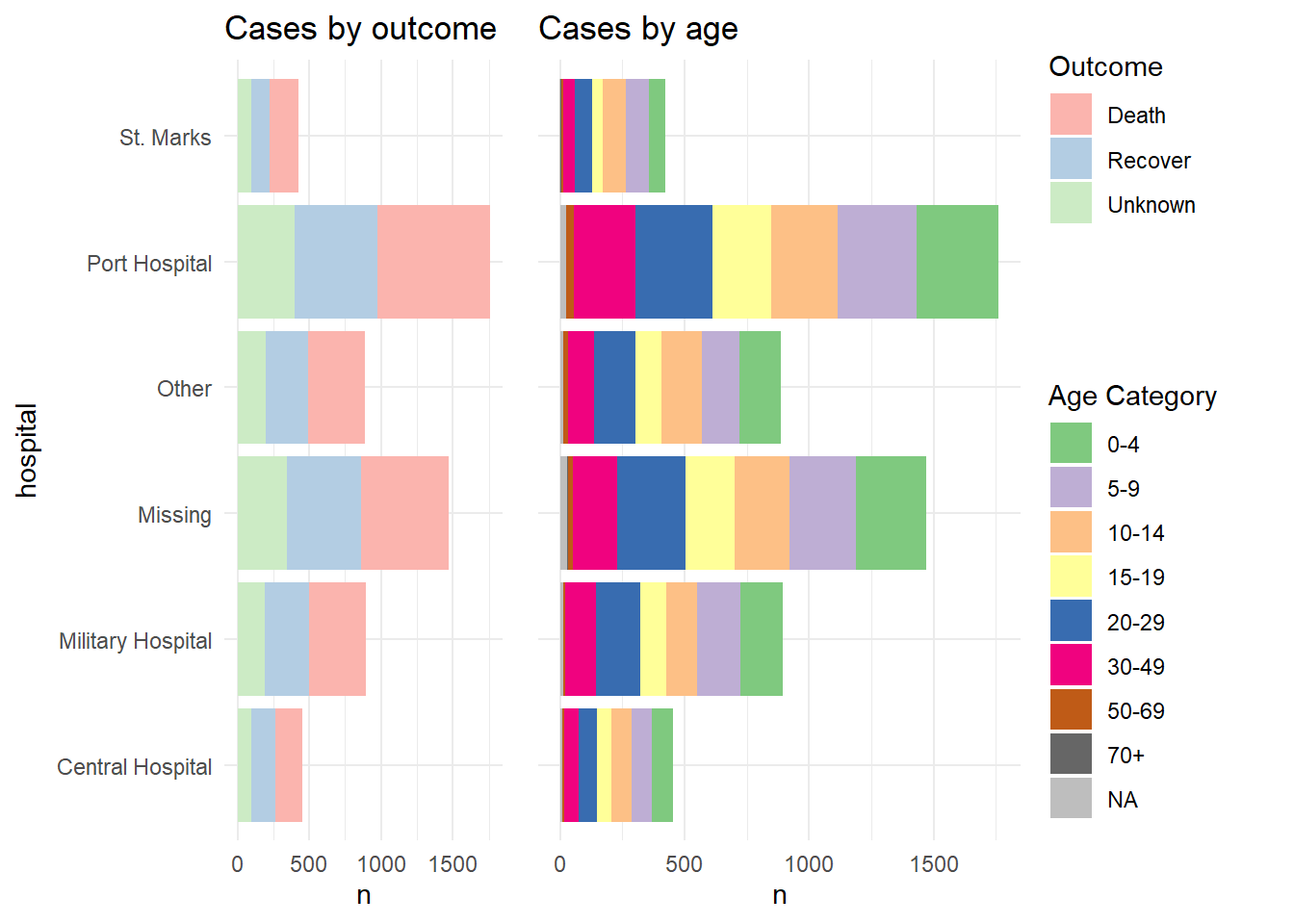
p1 <- linelist %>%
mutate(hospital = recode(hospital, "St. Mark's Maternity Hospital (SMMH)" = "St. Marks")) %>%
count(hospital, outcome) %>%
ggplot()+
geom_col(mapping = aes(x = hospital, y = n, fill = outcome))+
scale_fill_brewer(type = "qual", palette = 4, na.value = "grey")+
coord_flip()+
theme_minimal()+
labs(title = "Cases by outcome")
p2 <- linelist %>%
mutate(hospital = recode(hospital, "St. Mark's Maternity Hospital (SMMH)" = "St. Marks")) %>%
count(hospital, age_cat) %>%
ggplot()+
geom_col(mapping = aes(x = hospital, y = n, fill = age_cat))+
scale_fill_brewer(type = "qual", palette = 1, na.value = "grey")+
coord_flip()+
theme_minimal()+
theme(axis.text.y = element_blank())+
labs(title = "Cases by age")Вот как выглядят два графика при их объединении с помощью plot_grid() без объединения легенд:
А теперь покажем, как объединить легенды. По сути, мы определяем каждый график без легенды (theme(legend.position = "none")), а затем определяем легенду каждого графика отдельно, используя функцию get_legend() из cowplot. Когда мы извлекаем легенду из сохраненного графика, нам необходимо добавить + легенду обратно, включая указание размещения (““справа”“) и небольшие корректировки для выравнивания легенд и их заголовков. Затем мы объединяем легенды по вертикали, а затем объединяем два графика с объединенными легендами. Вуаля!
# Определить график 1 без легенды
p1 <- linelist %>%
mutate(hospital = recode(hospital, "St. Mark's Maternity Hospital (SMMH)" = "St. Marks")) %>%
count(hospital, outcome) %>%
ggplot()+
geom_col(mapping = aes(x = hospital, y = n, fill = outcome))+
scale_fill_brewer(type = "qual", palette = 4, na.value = "grey")+
coord_flip()+
theme_minimal()+
theme(legend.position = "none")+
labs(title = "Cases by outcome")
# Определить график 2 без легенды
p2 <- linelist %>%
mutate(hospital = recode(hospital, "St. Mark's Maternity Hospital (SMMH)" = "St. Marks")) %>%
count(hospital, age_cat) %>%
ggplot()+
geom_col(mapping = aes(x = hospital, y = n, fill = age_cat))+
scale_fill_brewer(type = "qual", palette = 1, na.value = "grey")+
coord_flip()+
theme_minimal()+
theme(
legend.position = "none",
axis.text.y = element_blank(),
axis.title.y = element_blank()
)+
labs(title = "Cases by age")
# извлечение легенды из гр1 (из гр1 + легенда)
leg_p1 <- cowplot::get_legend(p1 +
theme(legend.position = "right", # извлечение вертикальной легенды
legend.justification = c(0,0.5))+ # легенды должны быть выровнены
labs(fill = "Outcome")) # заголовок легенды
# извлечение легенды из гр2 (из гр2 + легенда)
leg_p2 <- cowplot::get_legend(p2 +
theme(legend.position = "right", # извлечение вертикальной легенды
legend.justification = c(0,0.5))+ # легенды должны быть выровнены
labs(fill = "Age Category")) # заголовок легенды
# создание пустого графика для выравнивания легенды
#blank_p <- patchwork::plot_spacer() + theme_void()
# создание панелей легенд, которые могут располагаться одна на другой (или использовать разделитель, о котором говорилось выше)
legends <- cowplot::plot_grid(leg_p1, leg_p2, nrow = 2, rel_heights = c(.3, .7))
# объединение двух графиков и панели объединенных легенд
combined <- cowplot::plot_grid(p1, p2, legends, ncol = 3, rel_widths = c(.4, .4, .2))
combined # печатьЭто решение было взято из этого сообщения с небольшим исправлением выравнивания легенд из этого сообщения.
СОВЕТ: Интересный факт - ““cow”” в cowplot происходит от имени создателя - Claus O. Wilke.
Вставные графики
С помощью cowplot можно вставить один график в другой. При этом следует обратить внимание на следующие моменты:
- Определите основной график с помощью
theme_half_open()из cowplot; возможно, будет лучше, если легенда будет располагаться сверху или снизу
- Определить вставной график. Лучше всего иметь график, где легенда не нужна. Элементы темы графика можно удалить с помощью
element_blank(), как показано ниже.
- Объедините их, применив функцию
ggdraw()к основному графику, затем добавьтеdraw_plot()на вставной график и укажите координаты (x и y левого нижнего угла), высоту и ширину в пропорции ко всему основному графику.
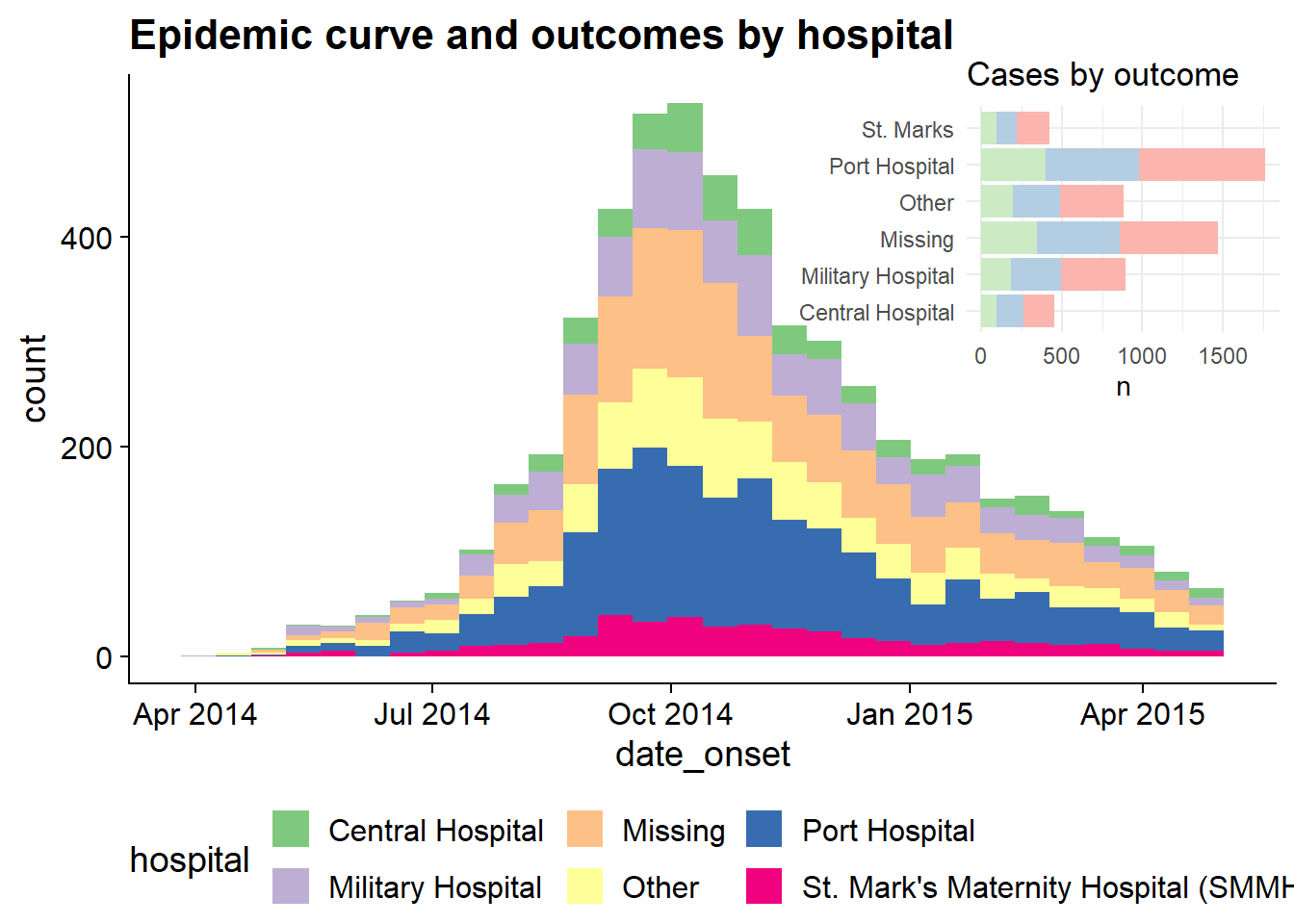
# Определить основной график
main_plot <- ggplot(data = linelist)+
geom_histogram(aes(x = date_onset, fill = hospital))+
scale_fill_brewer(type = "qual", palette = 1, na.value = "grey")+
theme_half_open()+
theme(legend.position = "bottom")+
labs(title = "Epidemic curve and outcomes by hospital")
# Определить вставной график
inset_plot <- linelist %>%
mutate(hospital = recode(hospital, "St. Mark's Maternity Hospital (SMMH)" = "St. Marks")) %>%
count(hospital, outcome) %>%
ggplot()+
geom_col(mapping = aes(x = hospital, y = n, fill = outcome))+
scale_fill_brewer(type = "qual", palette = 4, na.value = "grey")+
coord_flip()+
theme_minimal()+
theme(legend.position = "none",
axis.title.y = element_blank())+
labs(title = "Cases by outcome")
# Объединить основной график и вставной
cowplot::ggdraw(main_plot)+
draw_plot(inset_plot,
x = .6, y = .55, #x = .07, y = .65,
width = .4, height = .4)Более подробно этот метод объясняется в этих двух виньетках:
31.11 Двойные оси
Вторичная ось y часто является необходимым дополнением к графику ggplot2. Хотя в сообществе специалистов по визуализации данных ведутся активные споры о целесообразности таких графиков, и зачастую их не рекомендуют использовать, тем не менее они могут вам понадобиться. Ниже мы приводим один из способов их получения: использование пакета cowplot для объединения двух отдельных графиков.
Этот подход предполагает создание двух отдельных графиков - одного с осью y слева и другого с осью y справа. Оба графика будут использовать определенную theme_cowplot() и должны иметь одинаковую ось x. Затем в третьей команде оба графика выравниваются и накладываются друг на друга. Функциональные возможности cowplot, из которых это только одна, подробно описаны на этом сайте.
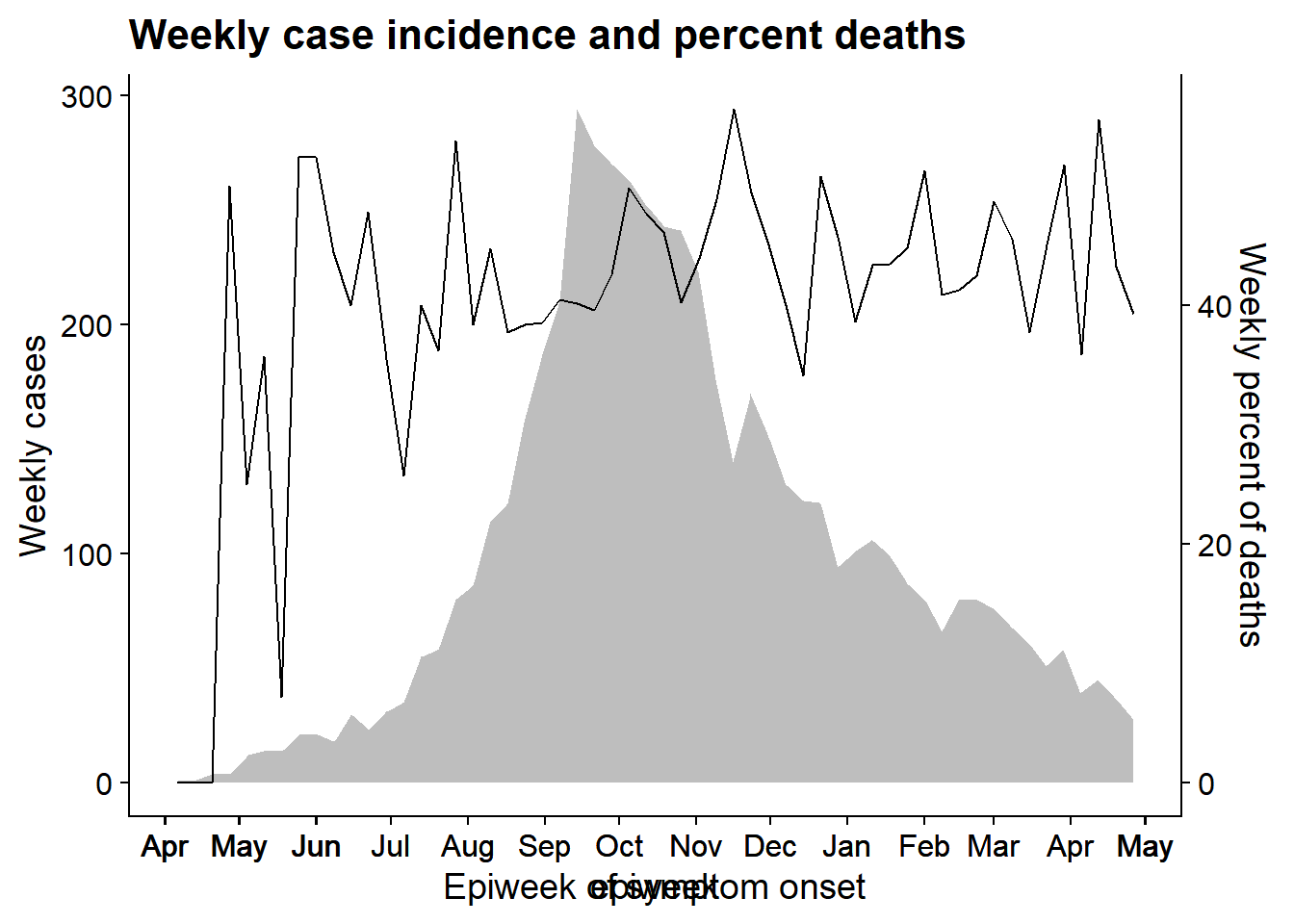
Для демонстрации этого метода мы наложим эпидемическую кривую на линию недельного процента умерших пациентов. Мы используем этот пример, поскольку выравнивание дат по оси x является более сложной задачей, чем, например, выравнивание гистограммы с другим графиком. Следует отметить следующие моменты:
- Перед построением эпикривая и линия агрегируются по неделям * и *
date_breaksиdate_labelsидентичны - это делается для того, чтобы оси x двух графиков совпадали при наложении.
- Для графика 2 ось y сдвигается вправо с помощью аргумента
position =argument ofscale_y_continuous().
- В обоих графиках используется
theme_cowplot().
Обратите внимание, что на странице [Эпидемические кривые] есть еще один пример использования этого метода - наложение кумулятивной заболеваемости на эпикривую.
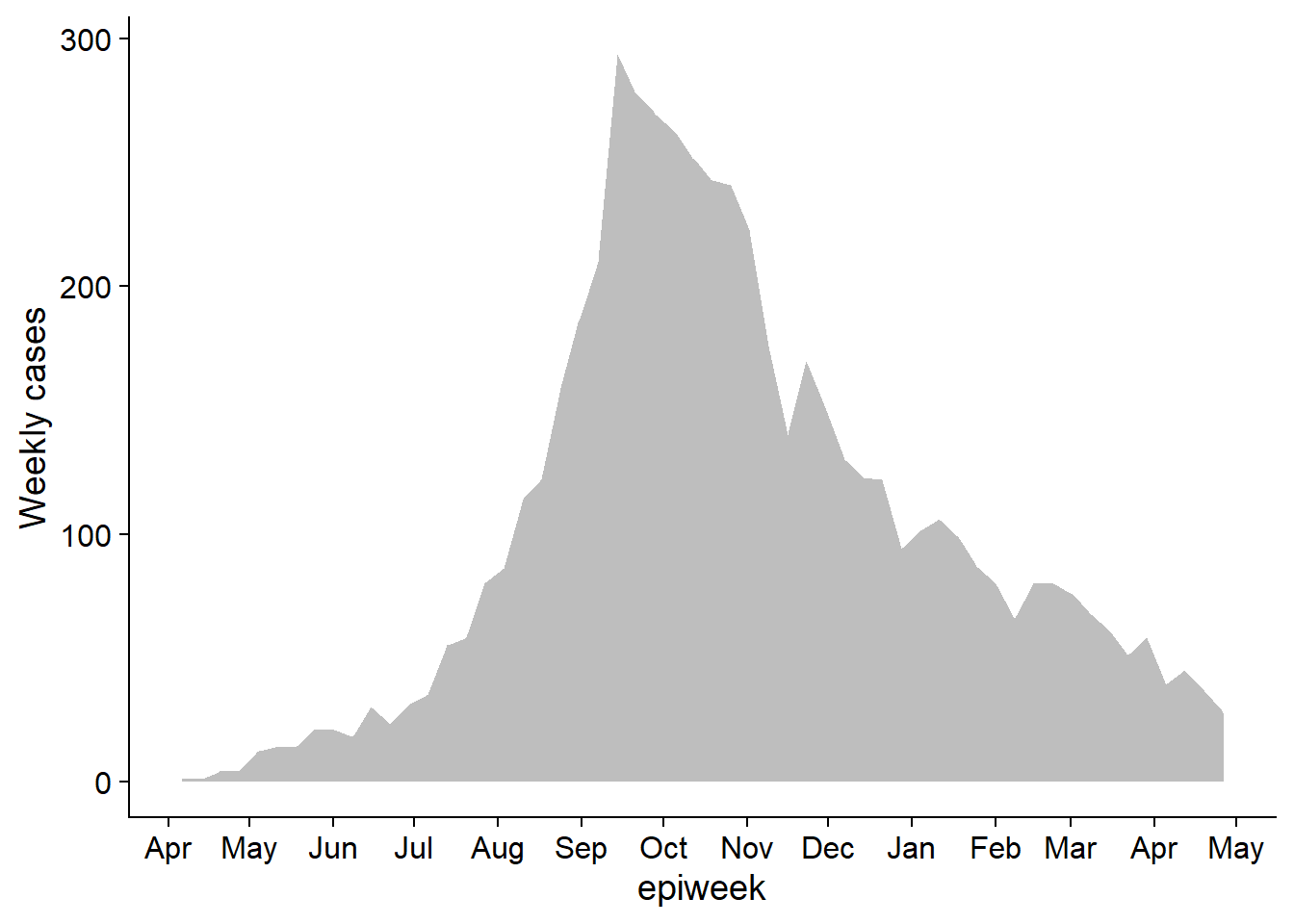
Построить график 1
По сути, это и есть эпикривая. Мы используем geom_area() только для того, чтобы продемонстрировать ее применение ( область под линией, по умолчанию)
pacman::p_load(cowplot) # загрузка/установка cowplot
p1 <- linelist %>% # сохранить график как объект
count(
epiweek = lubridate::floor_date(date_onset, "week")) %>%
ggplot()+
geom_area(aes(x = epiweek, y = n), fill = "grey")+
scale_x_date(
date_breaks = "month",
date_labels = "%b")+
theme_cowplot()+
labs(
y = "Weekly cases"
)
p1 # просмотр графика Построить график 2
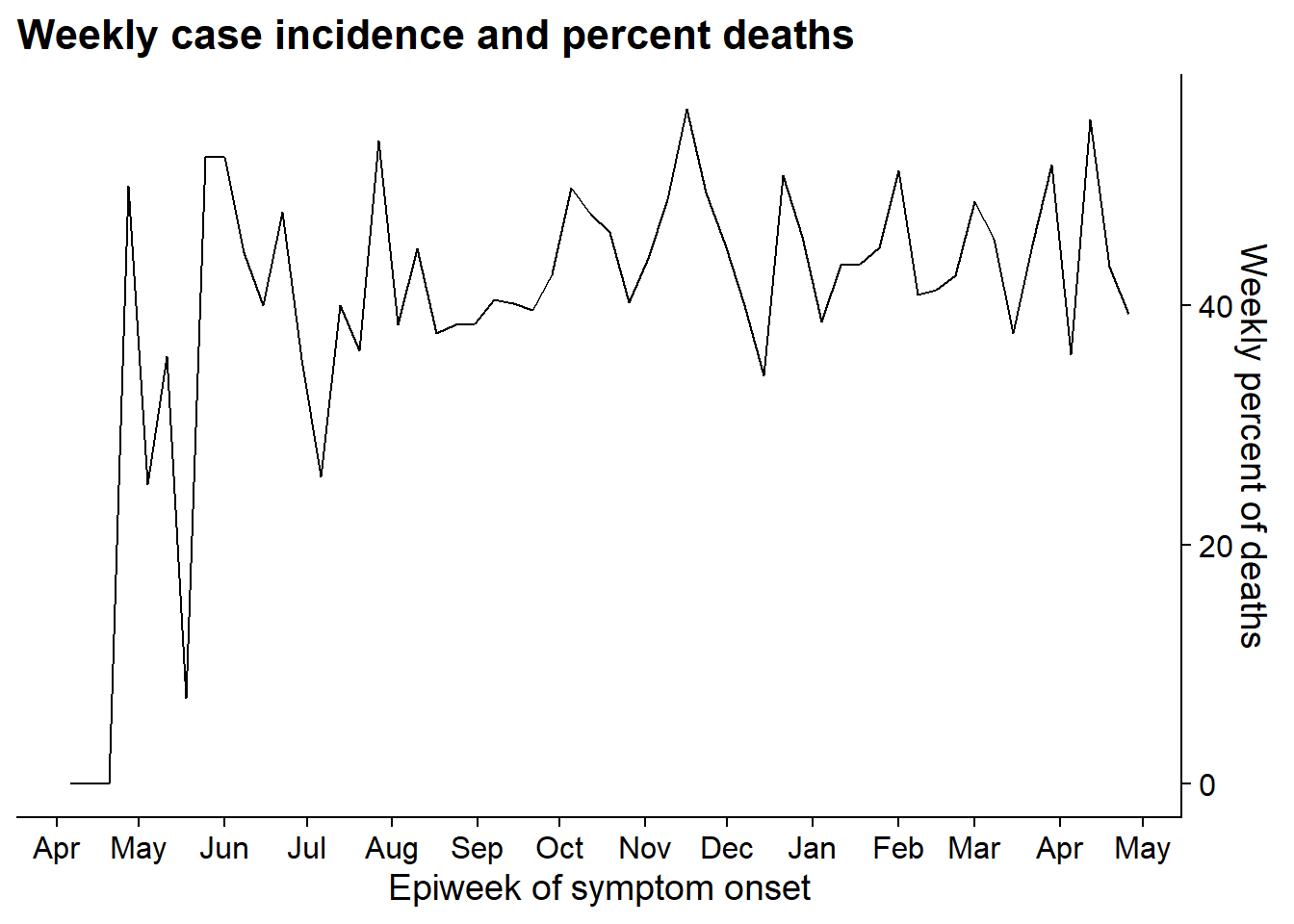
Постройте второй график, на котором изобразите линию еженедельного процента случаев смерти.
p2 <- linelist %>% # сохранить график как объект
group_by(
epiweek = lubridate::floor_date(date_onset, "week")) %>%
summarise(
n = n(),
pct_death = 100*sum(outcome == "Death", na.rm=T) / n) %>%
ggplot(aes(x = epiweek, y = pct_death))+
geom_line()+
scale_x_date(
date_breaks = "month",
date_labels = "%b")+
scale_y_continuous(
position = "right")+
theme_cowplot()+
labs(
x = "Epiweek of symptom onset",
y = "Weekly percent of deaths",
title = "Weekly case incidence and percent deaths"
)
p2 # просмотр графика Теперь выравниваем график с помощью функции align_plots(), задавая выравнивание по горизонтали и вертикали (“hv”, также может быть “h”, “v”, “,без выравнивания”). Выравнивание по всем осям (сверху, снизу, слева и справа) также задается функцией “tblr”. На выходе получаем список класса (2 элемента).
Затем мы рисуем два графика вместе, используя ggdraw() (из cowplot) и ссылаясь на две части объекта aligned_plots.
aligned_plots <- cowplot::align_plots(p1, p2, align="hv", axis="tblr") # выровнять два графика и сохранить их в виде списка
aligned_plotted <- ggdraw(aligned_plots[[1]]) + draw_plot(aligned_plots[[2]]) # наложить их друг на друга и сохранить визуальный график
aligned_plotted # печать наложенных друг на друга графиков31.12 Пакеты, которые вам помогут
Существует несколько очень удобных пакетов R, специально разработанных для того, чтобы помочь вам ориентироваться в ggplot2:
Наведите и щелкните ggplot2 с помощью equisse.
“Это дополнение позволяет интерактивно исследовать данные, визуализируя их с помощью пакета ggplot2. Оно позволяет строить столбчатые диаграммы, кривые, диаграммы рассеяния, гистограммы, коробчатые диаграммы и sf-объекты, а затем экспортировать график или получить код для его воспроизведения.”
Установите и запустите дополнение через меню RStudio или с помощью esquisse::esquisser().
См. Страница Github
31.13 Разное
Числовое отображение
Вы можете отключить научную нотацию, выполнив эту команду перед построением графика.
options(scipen=999)Или примените number_format() из пакета scales к конкретному значению или столбцу, как показано ниже.
С помощью функций из пакета scales можно легко настроить отображение чисел. Они могут быть применены к столбцам в датафрейме, но для примера показаны для отдельных чисел.
scales::number(6.2e5)[1] "620 000"scales::number(1506800.62, accuracy = 0.1,)[1] "1 506 800.6"scales::comma(1506800.62, accuracy = 0.01)[1] "1,506,800.62"scales::comma(1506800.62, accuracy = 0.01, big.mark = "." , decimal.mark = ",")[1] "1.506.800,62"scales::percent(0.1)[1] "10%"scales::dollar(56)[1] "$56"scales::scientific(100000)[1] "1e+05"31.14 Ресурсы
Для вдохновления Галерея графиков ggplot
Представление данных Европейский центр по профилактике и контролю заболеваний Руководство по представлению данных эпиднадзора
Фасеты и наложение меток Использование меток для последовательности фасет Наложение меток
Корректировка порядка с помощью факторов fct_reorder
fct_inorder
Как изменить порядок коробчатой диаграммы
Упорядочивание переменных в ggplot2
R для науки о данных - Факторы
Легенды
Настроить порядок легенд
Подписи Выравнивание подписей
Метки
ggrepel
Шпаргалки
Красивое построение графиков с помощью ggplot2
.
.