# устанавливаем последнюю версию пакета по Руководству для R для эпидемиологов
pacman::p_install_gh("appliedepi/epirhandbook")2 Скачивание руководства и данных
2.1 Download offline handbook
Вы можете загрузить автономную версию этого справочника в виде HTML-файла, чтобы просмотреть его в веб-браузере, даже если у вас больше нет доступа к Интернету. Если вы рассматриваете возможность использования справочника Epi R в автономном режиме, то следует обратить внимание на следующие моменты:
- При открытии файла может потребоваться минута или две для загрузки изображений и оглавления
- В автономном режиме руководство имеет несколько иной вид - одна очень длинная страница с оглавлением слева. Для поиска конкретных терминов используйте Ctrl+f (Cmd-f)
- См. страницу Рекомендованные пакеты, которая поможет вам установить соответствующие пакеты R до потери подключения к Интернету
- Установите наш пакет R epirhandbook, который содержит все примеры данных (процесс установки описан ниже)
Существует два способа скачивания руководства:
Использовать ссылку для скачивания
Ждя быстрого доступа правый щелчок по этой ссылке и выберите “Save link as - сохранить ссылку как”.
Если вы работаете на компьютере Mac, используйте Cmd+щелчок. Если вы используете мобильный телефон, нажмите и удерживайте ссылку, затем выберите “Сохранить ссылку”. Руководство будет загружено на ваше устройство. Если появится окно с необработанным HTML-кодом, убедитесь, что вы выполнили приведенные выше инструкции, или попробуйте вариант 2.
Использовать наш пакет R
Мы предлагаем пакет R под названием epirhandbook. Он включает функцию download_book(), которая скачивает файл с руководством с нашего репозитория Github на компьютер.
Этот пакет также содержит функцию get_data(), которая скачивает все примеры данных на ваш компьютер.
Выполните следующий код, чтобы установить наш пакет R epirhandbook с репозитория Github appliedepi. Этого пакета нет в CRAN, поэтому используйте специальную функцию p_install_gh(), чтобы установить его с Github.
Теперь загрузите пакет для использования в текущей сессии R:
# загружаем пакет для использования
pacman::p_load(epirhandbook)Далее, выполните функцию пакета download_book() (с пустыми скобками), чтобы скачать руководство на ваш компьютер. Если вы находитесь в RStudio, появится окно, позволяющее вам выбрать место для сохранения.
# скачиваем оффлайн руководство на компьютер
download_book()2.2 Скачивание данных для параллельной работы
Чторбы “работать параллельно” при просмотре страниц руководства, вы можете скачать пример данных и выходных результатов.
Используем наш пакет R
Самый простой подход для скачивания всех данных - установить наш пакет R epirhandbook. В нем есть функция get_data(), которая сохраняет все примеры данных в выбранную папку на компьютере.
Чтобы установить наш пакет R epirhandbook, выполните следующий код. Этого пакета нет в CRAN, поэтому используйте функцию p_install_gh(), чтобы его установить. Входные параметры ссылаются на нашу организацию в Github (“appliedepi”) и пакет epirhandbook.
# устанавливаем последню версию пакета для руководства по R
pacman::p_install_gh("appliedepi/epirhandbook")Теперь загрузите пакет для использования в текущей сессии R:
# загружаем пакет для использования
pacman::p_load(epirhandbook)Далее используем функцию пакета get_data(), чтобы скачать пример данных на ваш компьютер. Выполните get_data("all"), чтобы получить все примеры данных, либо укажите конкретное имя файлы и расширение внутри кавычек, чтобы получитьт только этот файл.
Данные уже загружены вместе с пакетом, и их необходимо просто перенести в папку на компьютере. Появится всплывающее окно, в котором можно выбрать местоположение папки для сохранения. Мы рекомендуем создать новую папку “data”, поскольку в ней находится около 30 файлов (включая примеры данных и примеры выходных данных).
# скачиваем все примеры данных в папку на компьютере
get_data("all")
# скачиваем только пример данных построчного списка в папку на компьютере
get_data(file = "linelist_cleaned.rds")# скачиваем конкретный файл в папку на вашем компьютере
get_data("linelist_cleaned.rds")Как только вы использовали get_data(), чтобы сохранить файл на компьютер, вам все еще нужно будет импортировать его в R. См. детали на странице Импорт и экспорт.
Если хотите, вы можете рассмотреть все данные, используемые в этом руководстве, в папке “data” в нашем репозитории Github.
Скачивание по одному
Этот вариант предполагает загрузку данных пофайлово из нашего репозитория Github либо по ссылке, либо с помощью команды R, специфичной для данного файла. Для некоторых типов файлов предусмотрена кнопка загрузки, в то время как другие можно загрузить с помощью команды R.
Построчный список случаев
Это вымышленная вспышка Эболы, расширенная командой, готовившей руководство, из набора данных для практики ebola_sim в пакете outbreaks.
Кликните, чтобы скачать the “сырой” построчный список (.xlsx). “Сырой” построчный список случаев - таблица Excel с хаотичными данными. Используйте ее, чтобы выполнять шаги на странице Вычистка данных и ключевые функции.
Кликните, чтобы скачать “чистый” построчный список (.rds). Используйте этот файл для всех других страниц данного руководства, где используется построчный список. Файл .rds - это конкретный тип файла для R, который сохраняет классы столбцов. Это позволит вам выполнить лишь минимальную вычистку после импорта данных в R.
Другие связанные файлы:
Кликните, чтобы скачать the “чистый” построчный список в виде файла Excel
Часть страницы вычистки использует “словарь вычистки” (.csv файл). Вы можете загрузить его напрямую в R, выполнив следующие команды:
pacman::p_load(rio) # установка/загрузка пакета rio
# импорт файла напрямую из Github
cleaning_dict <- import("https://github.com/appliedepi/epirhandbook_eng/raw/master/data/case_linelists/cleaning_dict.csv")Данные о количестве случаев малярии
Эти данные представляют собой фиктивное количество случаев заболевания малярией по возрастным группам, учреждениям и дням. Файл .rds - это специфический для R тип файла, сохраняющий классы столбцов. Благодаря этому после импорта данных в R вам придется выполнить лишь минимальную вычистку.
Кликните, чтобы скачать данные о количестве случаев малярии (.rds файл)
Данные для шкалы Лайкерта
Это вымышленные данные из опроса Лайкерта, используемые на странице Демографические пирамиды и шкалы Лайкерта. Вы можете загрузить эти данные напрямую в R, выполнив следующие команды:
pacman::p_load(rio) # установка/загрузка пакета rio
# импорт файла напрямую из Github
likert_data <- import("https://raw.githubusercontent.com/appliedepi/epirhandbook_eng/master/data/likert_data.csv")Flexdashboard
Ниже есть ссылки на файлы для страницы Информационные панели c R Markdown:
Отслеживание контактов
Страница Отслеживание контактов демонстрирует анализ данных по отслеживанию контактов, используя пример данных из Go.Data. Данные, используемые на этой странице, могут быть скачены как файлы .rds, кликнув на следующие ссылки:
Кликните, чтобы скачать данные по расследованию случая (.rds файл)
Кликните, чтобы скачать данные по отслеживанию контактов (.rds файл)
Кликните, чтобы скачать данные по мониторингу контактов (.rds файл)
ПРИМЕЧАНИЕ: Структурированные данные по отслеживанию контактов из других программ (например, KoBo, DHIS2 Tracker, CommCare) могут выглядеть по-другому. Если вы хотите использовать альтернативные данные лоя рьпащца иои содержания этой страницы, свяжитесь с нами.
СОВЕТ: Если вы используете данные Go.Data и хотите связать это с вашим API, см. страницу Импорта и экспорта (раздел по API) и Сообщество практиков Go.Data.
ГИС
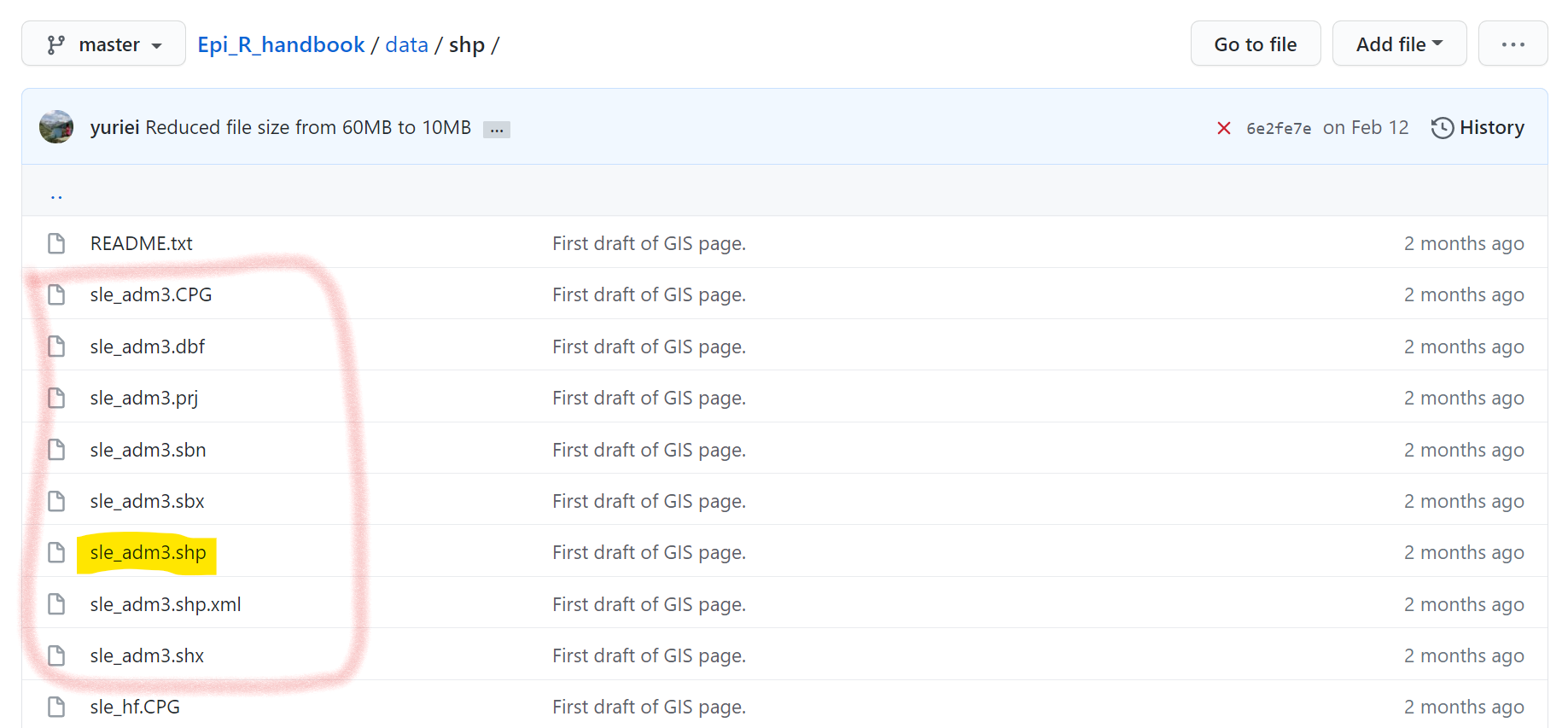
У шейп-файлов есть много файлов суб-компонентов, каждый со своим расширением файла. У одного файла будет расширение “.shp”, а у других могут быть “.dbf”, “.prj”, и т.п.
На странице Основы ГИС даны ссылки на веб-сайт Humanitarian Data Exchange, где вы можете скачать шейп-файлы напрямую в архивах.
Например, можно скачать точечные данные по медицинским организациям тут. Скачайте “hotosm_sierra_leone_health_facilities_points_shp.zip”. После сохранения на компьютер, разархивируйте папку. Вы увидите несколько файлов с разными расширениями (например, “.shp”, “.prj”, “.shx”) - их все нужно сохранять в одну папку на компьютере. Затем импортируйте в R, задайте путь к файлу и имя файла “.shp” в st_read() из пакетаsf (как описано на странице Основы ГИС).
Если вы используете Вариант 1 для скачивания всех примеров данных (через наш пакет R epirhandbook), все шейп-файлы уже включены.
В качестве альтернативы можно загрузить шейп-файлы из папки “data” справочника R на Github (см. вложенную папку “gis”). Однако учтите, что вам придется загружать каждый подфайл по отдельности на свой компьютер. В Github щелкните на каждом файле по отдельности и загрузите их, нажав на кнопку “Download”. Ниже показано, что шейпфайл “sle_adm3” состоит из множества файлов, каждый из которых необходимо загрузить с Github.
Филогенетические деревья
См. страницу Филогенетические деревья. Файл Newick филогенетического дерева, построенного на основе полногеномного секвенирования 299 образцов Shigella sonnei, и соответствующие данные по образцам (преобразованы в текстовый файл). Бельгийские образцы и полученные данные любезно предоставлены Бельгийским центром по изучению сальмонелл и шигелл в рамках проекта, осуществляемого стипендиатом EUPHEM ECDC, и также будут опубликованы в рукописи. Международные данные находятся в открытом доступе в общедоступных базах данных (ncbi) и были опубликованы ранее.
- Чтобы скачать файл с филогенетическим деревом “Shigella_tree.txt”, кликните правой кнопкой по этой ссылке (Cmd+click for Mac) и выберите “Сохранить ссылку как”.
- Чтобы скачать “sample_data_Shigella_tree.csv” с дополнительной информацией по каждому образцу, кликните правой кнопкой по этой ссылке (Cmd+click для Mac) и выберите “Сохранить ссылку как”.
- Чтобы увидеть новое, созданное дерево с подмножеством, кликните правой кнопкой по этой ссылке (Cmd+click для Mac) и выберите “Сохранить ссылку как”. Файл .txt скачается на ваш компьютер.
Вы можете затем импортировать файлы .txt с помощью read.tree() из пакета ape, как объяснялось на странице.
ape::read.tree("Shigella_tree.txt")Стандартизация
См. страницу Стандартизированные коэффициенты. Вы можете загрузить данные напрямую из нашего репозитория Github в интернете в свою сессию R с помощью следующих команд:
# установка/загрузка пакета rio
pacman::p_load(rio)
##############
# Страна A
##############
# импорт демографических данных по Стране A напрямую из Github
A_demo <- import("https://github.com/appliedepi/epirhandbook_eng/raw/master/data/standardization/Страна_demographics.csv")
# импорт смертей по Стране A напрямую из Github
A_deaths <- import("https://github.com/appliedepi/epirhandbook_eng/raw/master/data/standardization/deaths_СтранаA.csv")
##############
# Страна B
##############
# импорт демографических данных по Стране B напрямую из Github
B_demo <- import("https://github.com/appliedepi/epirhandbook_eng/raw/master/data/standardization/Страна_demographics_2.csv")
# импорт смертей по Стране B напрямую из Github
B_deaths <- import("https://github.com/appliedepi/epirhandbook_eng/raw/master/data/standardization/deaths_СтранаB.csv")
###############
# Референтная популяция
###############
# импорт демографических данных по Стране B напрямую из Github
standard_pop_data <- import("https://github.com/appliedepi/epirhandbook_eng/raw/master/data/standardization/world_standard_population_by_sex.csv")Временные ряды и обнаружение вспышек
См. страницу Временные ряды и обнаружение вспышек. Мы используем случаи кампилобактера, зарегистрированные в Германии в 2002-2011, доступные в пакете R surveillance. (примечание этот набор данных был адаптирован из оригинального в той части, что 3 месяца данных конца 2011 года были удалены в целях демонстрации)
Кликните, чтобы скачать Campylobacter in Germany (.xlsx)
Мы также используем климатические данные по Германии за 2002-2011 гг. (температура в градусах Цельсия и количество осадков в миллиметрах). Они были загружены из набора данных спутникового реанализа ЕС Copernicus с помощью пакета ecmwfr. Вам нужно будет скачать их все и импортировать с помощью stars::read_stars(), как объясняется на странице по временным рядам.
Кликните, чтобы скачать Погоду в Германии за 2002 (.nc файл)
Кликните, чтобы скачать Погоду в Германии за 2003 (.nc файл)
Кликните, чтобы скачать Погоду в Германии за 2004 (.nc файл)
Кликните, чтобы скачать Погоду в Германии за 2005 (.nc файл)
Кликните, чтобы скачать Погоду в Германии за 2006 (.nc файл)
Кликните, чтобы скачать Погоду в Германии за 2007 (.nc файл)
Кликните, чтобы скачать Погоду в Германии за 2008 (.nc файл)
Кликните, чтобы скачать Погоду в Германии за 2009 (.nc файл)
Кликните, чтобы скачать Погоду в Германии за 2010 (.nc файл)
Кликните, чтобы скачать Погоду в Германии за 2011 (.nc файл)
Анализ опросов
Для страницы Анализ опросов мы используем выдуманные данные исследования смертности на основе шаблонов опросов MSF OCA. Этот выдуманный набор данных был сгенерирован в рамках проекта “R4Epis”.
Кликните, чтобы скачать Вымышленные данные опроса (.xlsx)
Кликните, чтобы скачать Словарь данных вымышленного опроса (.xlsx)
Кликните, чтобы скачать Популяционные данные вымышленного опроса (.xlsx)
Shiny
Страница Информационные панели с Shiny демонстрирует создание простого приложения для отображения данных по малярии.
Чтобы скачать файлы R, которые создают приложение Shiny:
You can кликните, чтобы скачать файл facility_count_data.rds, который содержит данные по малярии для приложения Shiny. Обратите внимание, что вам нужно их сохранить в папке “data”, чтобы работал путь к файлу через here().
You can кликните, чтобы скачать файл global.R, который должен быть выполнен до открытия приложения, как объясняется на странице.
You can кликните, чтобы скачать файл plot_epicurve.R, к которому обращается global.R. Обратите внимание, что его нужно сохранить в папке “funcs”, чтобы работал путь к файлу через here().