pacman::p_load(
rio, # Importa arquivos
here, # Localiza arquivos
tidyverse, # gestão dos dados + gráficos no ggplot2
stringr, # manipulação de textos em formato string
purrr, # explore os objetos de forma organizada
gtsummary, # resumos estatísticos e testes
broom, # organize os resultados das regressões
lmtest, # testes de relação de verosimilhança
parameters, # alternativa para organizar os resultados das regressões
see # alternativa para visualizar os gráficos em floresta
)19 Regressão simples e múltipla
Esta página demonstra o uso das funções de regressão do pacote R base , como glm(), e o pacote gtsummary para verificar as associações entre as variáveis (ex.: riscos relativos, relações de risco e hazard ratio). Também serão mostradads funções como tidy(), do pacote broom, para limpar os resultados da regressão.
- Univariado: tabelas 2 x 2
- Estratificado: método de Mantel-Haenszel
- Multivariada: seleção variada, seleção de modelo, tabela final
- Gráfico em floresta
Para realizar a regressão proporcional de riscos (regressão Cox), veja a página Análise de sobrevivência.
NOTE: O termo multivariável é utilizado aqui ao se referir à uma regressão com múltiplas variáveis explicativas. Assim, um modelo multivariado seria uma regressão com diferentes resultados - veja esse editorial para detalhes
19.1 Preparação
Carregue os pacotes
Este código realiza o carregamento dos pacotes necessários para as análises. Neste manual, nós enfatizamos o uso da função p_load(), do pacote pacman, que instala os pacotes, caso necessário, e os carrega para utilização. Você também pode utilizar a função library(), do pacote R base , para carregar pacotes instalados. Veja a página sobre o Introdução ao R](#basics) para mais informações sobre os pacotes R.
Importe os dados
Nós iremos importar o banco de dados dos casos de uma simulação de epidemia de Ebola. Se você quiser acompanhar os passos abaixo, clique aqui para fazer o download do banco de dados ‘limpo’ (como arquivo .rds). Importe seus dados utilizando a função import(), do pacote rio (esta função importa muitos tipos de arquivos, como .xlsx, .rds, .csv - veja a página Importar e exportar para detalhes).
# importe os dados no R no objeto linelist
linelist <- import("linelist_cleaned.rds")As primeiras 50 linhas do linelist são mostradas abaixo.
Limpando os dados
Armazene as variáveis explicativas
No código abaixo, os nomes das colunas explicativas são salvos como um vetor de caracteres. Eles serão utilizados posteriormente.
## escolha as variáveis de interesse
explanatory_vars <- c("gender", "fever", "chills", "cough", "aches", "vomit")Converta para 1s e 0s
Abaixo, as colunas de variáveis explicativas com as opções binárias “yes”/“no”, “m”/“f”, e “dead”/“alive” são convertidas para 1 / 0, visando serem utilizadas nos modelos de regressão. Para fazer isso de forma eficiente, utilize a função across(), do dplyr, para transformar múltiplas colunas de uma vez. A função case_when() (também do dplyr) utiliza argumentos lógicos para converter valores específicos para 1s ou 0s. Veja as seções das funções across() e case_when() na página de limpando dados e funções essenciais).
Nota: o “.” abaixo representa a coluna que está sendo processada pela função across() no momento.
## converte variáveis dicotômicas para 0/1
linelist <- linelist %>%
mutate(across(
.cols = all_of(c(explanatory_vars, "outcome")), ## para cada coluna listada e "outcome"
.fns = ~case_when(
. %in% c("m", "yes", "Death") ~ 1, ## transforma male/yes/death em 1
. %in% c("f", "no", "Recover") ~ 0, ## transforma female/no/recover em 0
TRUE ~ NA_real_) ## do contrário, transforma em 'missing'
)
)Exclua linhas com valores em branco
Para excluir linhas com valores em branco, é possível utilizar a função drop_na(), do pacote tidyr. Entretanto, nós queremos que isso aconteça apenas nas linhas com campos em branco nas colunas de interesse.
A primeira coisa a fazer é garantir que o nosso vetor explanatory_vars contenha a coluna age (age deve ter gerado um erro na operação anterior utilizando case_when(), que era apenas para variáveis dicotômicas). Então, nós utilizamos o objeto linelist na função drop_na() para remover qualquer linha com campos em branco na coluna outcome ou em qualquer uma das colunas salvas em explanatory_vars.
Antes de executar o código, o número de linhas no objeto linelist é obtido por nrow(linelist).
## adiciona a coluna age_category no vetor explanatory_vars
explanatory_vars <- c(explanatory_vars, "age_cat")
## exclua linhas com campos em branco nas variáveis de interesse
linelist <- linelist %>%
drop_na(any_of(c("outcome", explanatory_vars)))O número de linhas restante no linelist é nrow(linelist).
19.2 Univariado
Assim como na página sobre Tabelas descritivas, os seus objetivos irão determinar quais pacotes R utilizar. Aqui, nós apresentamos duas opções para realizar análises univariadas:
- Utilize as funções disponíveis no pacote R base para rapidamente obter os resultados no terminal. Utilize o pacote broom para organizar os resultados.
- Utilize o pacote gtsummary para modelar e obter resultados prontos para publicação
Pacote R base
Regressão linear
A função lm(), do R base, executa regressões lineares, avaliando a relação entre respostas numéricas e variáveis explanatórias (independentes), que se presume terem uma relação linear.
Forneça a equação como uma fórmula, com os nomes das colunas contendo as respostas numéricas e as variáveis explanatórias separadas por um til ~. Adicionalmente, especifique qual o banco de dados em data =. Para utiliza-los posteriormente, atribua os resultados da modelagem a um objeto R.
lm_results <- lm(ht_cm ~ age, data = linelist)Você pode executar a função summary() nos resultados obtidos para visualizar os coeficientes (Estimates), p-valor, resíduos, e outras medições.
summary(lm_results)
Call:
lm(formula = ht_cm ~ age, data = linelist)
Residuals:
Min 1Q Median 3Q Max
-128.579 -15.854 1.177 15.887 175.483
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 69.9051 0.5979 116.9 <2e-16 ***
age 3.4354 0.0293 117.2 <2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 23.75 on 4165 degrees of freedom
Multiple R-squared: 0.7675, Adjusted R-squared: 0.7674
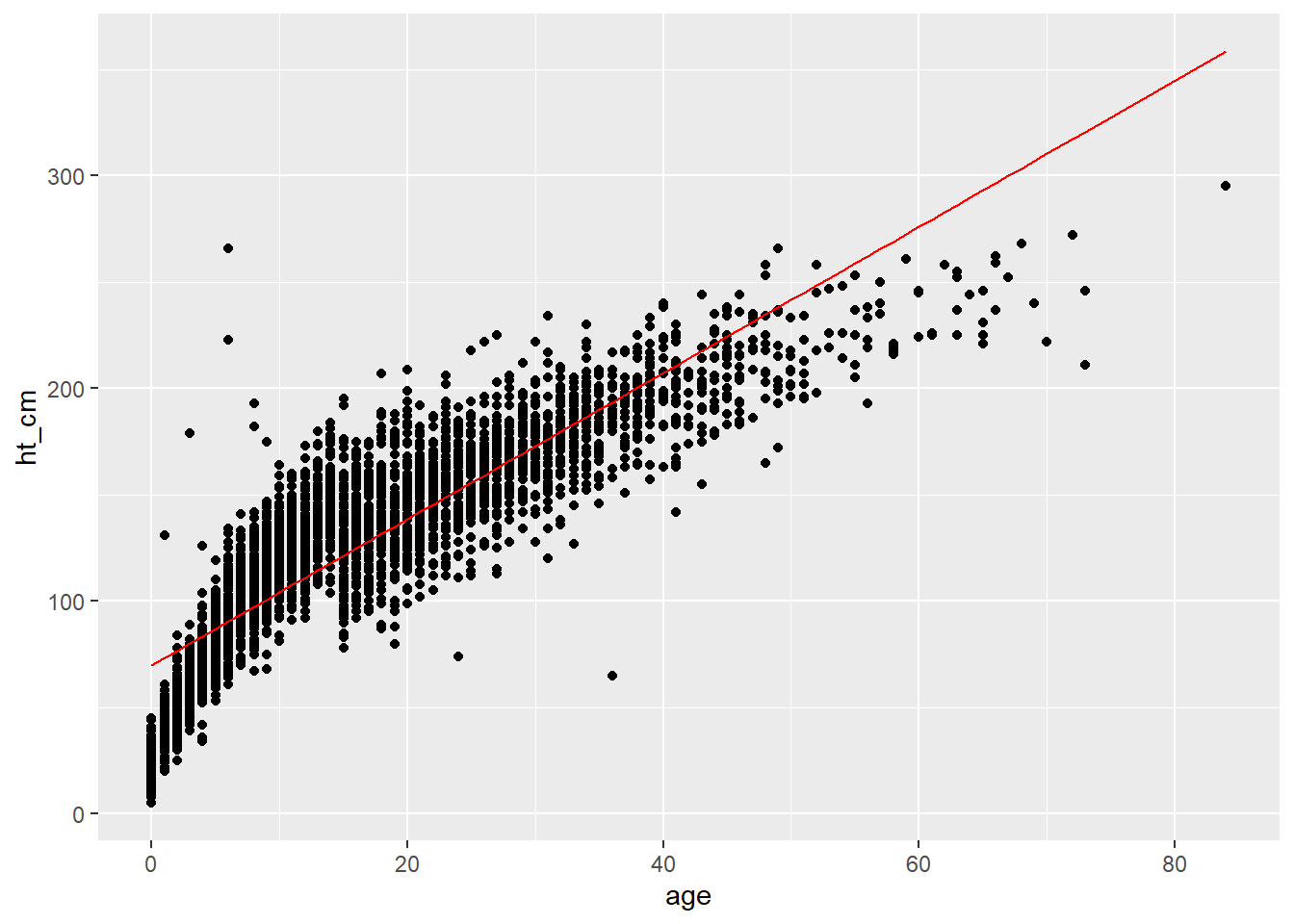
F-statistic: 1.375e+04 on 1 and 4165 DF, p-value: < 2.2e-16Alternativamente, é possível utilizar a função tidy(), do pacote broom, para organizar os resultados em uma tabela. O que os resultados nos mostram é que, a cada ano, a altura aumenta em 3.5 cm, e isto é estatisticamente significativo.
tidy(lm_results)# A tibble: 2 × 5
term estimate std.error statistic p.value
<chr> <dbl> <dbl> <dbl> <dbl>
1 (Intercept) 69.9 0.598 117. 0
2 age 3.44 0.0293 117. 0Você também pode adicionar essa regressão na função ggplot. Para tanto, primeiro plotamos os dados observados e a linha de tendência da regressão linear em um quadro de dados utilizando a função augment(), do pacote broom.
## coloque os pontos da regressão e os dados observados em um banco de dados
points <- augment(lm_results)
## trace um gráfico dos dados utilizando a variável 'age' no eixo x
ggplot(points, aes(x = age)) +
## inclua os pontos para a altura
geom_point(aes(y = ht_cm)) +
## inclua a linha de tendência da regressão linear
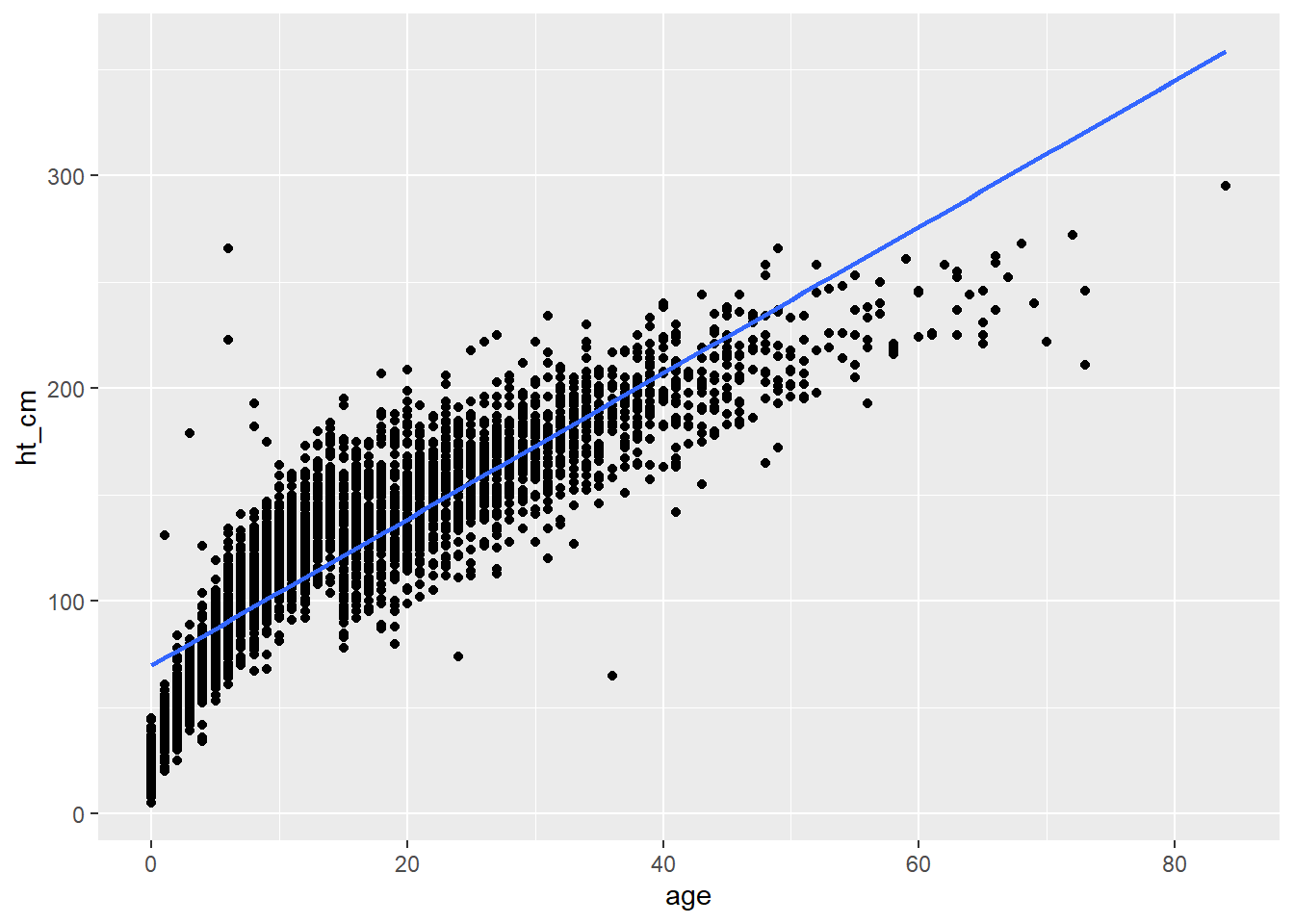
geom_line(aes(y = .fitted), colour = "red")Também é possível adicionar uma linha simples de regressão linear diretamente no ggplot, utilizando a função geom_smooth().
## coloque seus dados em um gráfico
ggplot(linelist, aes(x = age, y = ht_cm)) +
## mostre os pontos
geom_point() +
## inclua uma regressão linear
geom_smooth(method = "lm", se = FALSE)`geom_smooth()` using formula = 'y ~ x'Veja a seção sobre Recursos extras no final deste capítulo para mais tutoriais detalhados.
Regressão logística
A função glm(), do pacote stats (parte do pacote R base), é utilizada para ajustar Modelos Lineares Generalizados (GLM).
glm() pode ser utilizada para regressões logísticas univariadas e multivariadas (ex.: para obter probabilidades). Aqui estão as partes principais:
# argumentos utilizados na função glm()
glm(formula, family, data, weights, subset, ...)-
formula =o modelo é fornecido aoglm()como uma equação, com o resultado no lado esquerdo e as variáveis explicativas no lado direito de um til~. -
family =Isto determina o tipo de modelo a ser executado. Para regressão logística, utilizefamily = "binomial", para o modelo log-linear de poisson utilizefamily = "poisson". Outros exemplos estão na tabela abaixo. -
data =Especifique sua fonte de dados
Se necessário, você pode especificar o link da função utilizando a sintaxe family = familytype(link = "linkfunction")). Você pode obter mais informações sobre outras famílias e argumentos opcionais, como weights = e subset = (?glm), na documentação.
| Família | Link padrão da função |
|---|---|
"binomial" |
(link = "logit") |
"gaussian" |
(link = "identity") |
"Gamma" |
(link = "inverse") |
"inverse.gaussian" |
(link = "1/mu^2") |
"poisson" |
(link = "log") |
"quasi" |
(link = "identity", variance = "constant") |
"quasibinomial" |
(link = "logit") |
"quasipoisson" |
(link = "log") |
Ao executar a funçaõ glm(), é comum salvar os resultados em um objeto R. Assim, você pode visualizar os resultados em seu terminal utilizando a função summary(), como mostrado abaixo, ou realizar outras operações com os resultados (ex.: potenciação).
Se você precisa executar uma regressão binominal negativa, é possível utilizar o pacote MASS; a função glm.nb() utiliza a mesma sintaxe que glm(). Para um passo a passo sobre os diferentes modelos de regressão, acesse a página sobre estatística da UCLA.
Função glm() univariada
Neste exemplo, nós iremos avaliar a associação entre diferentes categorias de idades e a evolução para óbito (codificado como 1 na seção de Preparação). Abaixo está um modelo univariado de outcome por age_cat. Os resultados do modelo são savos como model, e mostrados no terminal com a função summary(). Observe que as estimativas fornecidas são as probabilidades em log, onde o nível base é o primeiro Factor (classe Factor) da variável age_cat(“0-4”).
model <- glm(outcome ~ age_cat, family = "binomial", data = linelist)
summary(model)
Call:
glm(formula = outcome ~ age_cat, family = "binomial", data = linelist)
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) 0.233738 0.072805 3.210 0.00133 **
age_cat5-9 -0.062898 0.101733 -0.618 0.53640
age_cat10-14 0.138204 0.107186 1.289 0.19726
age_cat15-19 -0.005565 0.113343 -0.049 0.96084
age_cat20-29 0.027511 0.102133 0.269 0.78765
age_cat30-49 0.063764 0.113771 0.560 0.57517
age_cat50-69 -0.387889 0.259240 -1.496 0.13459
age_cat70+ -0.639203 0.915770 -0.698 0.48518
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
(Dispersion parameter for binomial family taken to be 1)
Null deviance: 5712.4 on 4166 degrees of freedom
Residual deviance: 5705.1 on 4159 degrees of freedom
AIC: 5721.1
Number of Fisher Scoring iterations: 4Para alterar o nível base de comparação de dada variável, garanta que a coluna é da classe Factor e altere a primeira posição ao nível desejado utilizando a função fct_relevel() (veja a página sobre Fatores). Por exemplo, abaixo nós adaptamos a coluna age_cat e escolhemos “20-29” como nível base antes de aplicar estes dados na função glm().
linelist %>%
mutate(age_cat = fct_relevel(age_cat, "20-29", after = 0)) %>%
glm(formula = outcome ~ age_cat, family = "binomial") %>%
summary()
Call:
glm(formula = outcome ~ age_cat, family = "binomial", data = .)
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) 0.26125 0.07163 3.647 0.000265 ***
age_cat0-4 -0.02751 0.10213 -0.269 0.787652
age_cat5-9 -0.09041 0.10090 -0.896 0.370220
age_cat10-14 0.11069 0.10639 1.040 0.298133
age_cat15-19 -0.03308 0.11259 -0.294 0.768934
age_cat30-49 0.03625 0.11302 0.321 0.748390
age_cat50-69 -0.41540 0.25891 -1.604 0.108625
age_cat70+ -0.66671 0.91568 -0.728 0.466546
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
(Dispersion parameter for binomial family taken to be 1)
Null deviance: 5712.4 on 4166 degrees of freedom
Residual deviance: 5705.1 on 4159 degrees of freedom
AIC: 5721.1
Number of Fisher Scoring iterations: 4Vizualizando os resultados
Na maioria das vezes, diferentes modificações precisam ser feitas nos resultados acima. A função tidy(), do pacote broom, é conveniente para transformar os resultados em um formato apresentável.
Aqui, nós demonstramos como combinar o resultado da modelagem com uma tabela de contagens.
- Obtenha as probabilidades em log na forma exponencial e os intervalos de confiança ao aplicar o modelo na função
tidy(), e ajustar os atributosexponentiate = TRUEeconf.int = TRUE.
model <- glm(outcome ~ age_cat, family = "binomial", data = linelist) %>%
tidy(exponentiate = TRUE, conf.int = TRUE) %>% # realize a potenciação e produza intervalos de confiança
mutate(across(where(is.numeric), round, digits = 2)) # arredonde todas as colunas numéricasWarning: There was 1 warning in `mutate()`.
ℹ In argument: `across(where(is.numeric), round, digits = 2)`.
Caused by warning:
! The `...` argument of `across()` is deprecated as of dplyr 1.1.0.
Supply arguments directly to `.fns` through an anonymous function instead.
# Previously
across(a:b, mean, na.rm = TRUE)
# Now
across(a:b, \(x) mean(x, na.rm = TRUE))Abaixo é o objeto model mostrado de forma organizada:
- Combine os resultados dessa modelagem com uma tabela de contagens. Abaixo, nós criamos uma tabela cruzada de contagens com a função
tabyl()do pacote janitor, como descrito na página sobre Tabelas descritivas:
counts_table <- linelist %>%
janitor::tabyl(age_cat, outcome)Aqui está como os dados em counts_table ficam quando tabelados:
Agora nós podemos ligar os resultados dos objetos counts_table e model horizontalmente com a função bind_cols() (dplyr). Lembre que, com a função bind_cols(), as linhas dos dois objetos precisam estar alinhadas perfeitamente. Neste código, porque estamos ligando uma cadeia de comandos, nós utilizamos o . para representar o objeto de counts_table enquanto o ligamos ao model. Para finalizar o processo, a função select() é utilizada para selecionar colunas de interesse e sua ordem, e finalmente aplicar a função round(), do R base, em todas as colunas numéricas para até duas casas decimais.
combined <- counts_table %>% # inicie com uma tabela de contagens
bind_cols(., model) %>% # combine ela com os resultados da regressão
select(term, 2:3, estimate, # selecione e organize as colunas
conf.low, conf.high, p.value) %>%
mutate(across(where(is.numeric), round, digits = 2)) # arredonde para 2 casas decimaisAqui está o resultado da combinação de duas tabelas, exportado como um bela imagem utilizando uma função do flextable. A página Tabelas para apresentação explica como customizar tais tabelas com o flextable, mas você pode utilizar outros inúmeros pacotes do R, como knitr ou GT.
combined <- combined %>%
flextable::qflextable()Rodando múltiplos modelos univariados
Abaixo, nós mostramos um método usando glm()e tidy(). Para uma abordagem mais simples, veja a seção sobre o gtsummary.
Para rodar os modelos com diferentes variáveis e produzir probabilidades univariadas (ex.: sem dependência entre elas), você pode utilizar a abordagem abaixo. Ela utiliza a função str_c(), do pacote stringr, para criar fórmulas univaridas (veja a página caracteres e strings), e rodar a regressão glm()com cada fórmula, aplicando cada resultado glm() no tidy(), e, finalmente, unindo todos os resultados dos modelos com a função bind_rows(), do pacote tidyr. Esta abordagem utiliza a função map(), do pacote purrr, para repetir as funções - veja a página sobre Iteração, loops, e listas para mais informações sobre essa ferramenta.
Crie um vetor com o nome das colunas com as variáveis explicativas. Nós já criamos este vetor como
explanatory_varsna seção Preparação desta página.Utilize a função
str_c()para criar múltiplas fórmulas em texto, com ooutcomeno lado esquerdo, e o nome de uma coluna doexplanatory_varsno lado direito. O ponto final ‘.’ é substituído pelo nome da coluna emexplanatory_vars.
explanatory_vars %>% str_c("outcome ~ ", .)[1] "outcome ~ gender" "outcome ~ fever" "outcome ~ chills"
[4] "outcome ~ cough" "outcome ~ aches" "outcome ~ vomit"
[7] "outcome ~ age_cat"Utilize essas fórmulas na função
map()e ajuste~glm()como a função a ser utilizada com cada entrada. Dentro da funçãoglm(), ajuste a fórmula de regressão paraas.formula(.x), onde.xserá substituído pela fórmula definida na etapa acima. A funçãomap()irá rodar com cada uma das fórmulas, executando regressões para cada uma.Os resultados deste primeiro
map()são utilizados em um segundo comandomap(), que aplicatidy()nos resultados das regressões.Finalmente, o resultado do segundo
map()(uma lista de quadros de dados organizados) é condensado com a funçãobind_rows(), resultando em um quadro de dados com todos os resultados univariados.
models <- explanatory_vars %>% # inicie com as variáveis de interesse
str_c("outcome ~ ", .) %>% # combine cada variável na fórmula ("outcome ~ variável de interesse")
# repita as etapas para cada fórmula univariável
map(
.f = ~glm( # utilize as fórmulas uma por uma no glm()
formula = as.formula(.x), # dentro do glm(), a formula é representada por .x
family = "binomial", # especifique o tipo do glm (logístico)
data = linelist)) %>% # indique o banco de dados
# organize cada um dos resultados das regressões acima
map(
.f = ~tidy(
.x,
exponentiate = TRUE, # realize a exponenciação
conf.int = TRUE)) %>% # obtenha os intervalos de confiança
# condensa a lista dos resultados das regressões em um único quadro de dados
bind_rows() %>%
# arredonde todas as colunas numéricas
mutate(across(where(is.numeric), round, digits = 2))Desta vez, o objeto final models é maior porque agora representa os resultados combinados de diferentes regressões univariadas. Clique na tabela para visualizar todas as linhas do model.
Como antes, nós podemos criar uma tabela de contagem do objeto linelist para cada variável explicativa, ligar ela no objeto models, e fazer uma bela tabela. Nós podemos começar com as variáveis, e repetir os processos com a função map(). Nós repetimos a execução de uma função definida pelo usuário, que envolve a criação de uma tabela de contagem utilizando as funções do dplyr. Então, os resultados são combinados e ligados com os resultados de models.
## para cada variável explanatória
univ_tab_base <- explanatory_vars %>%
map(.f =
~{linelist %>% ## inicie com o linelist
group_by(outcome) %>% ## agrupe os dados por outcome
count(.data[[.x]]) %>% ## produza contagens das variáveis de interesse
pivot_wider( ## transforme para o formato amplo (wide), como em uma tabulação cruzada
names_from = outcome,
values_from = n) %>%
drop_na(.data[[.x]]) %>% ## exclua as linhas com campos em branco
rename("variable" = .x) %>% ## altere a coluna com a variável de interesse para "variable"
mutate(variable = as.character(variable))} ## converta para caractéres, do contrário as variáveis não dicotômicas (categóricas) geram a classe factor e não podem ser unidas
) %>%
## condensa a lista com o resultado das contagens em um único quadro de dados
bind_rows() %>%
## une com os resultados da regressão
bind_cols(., models) %>%
## mantenha apenas as colunas de interesse
select(term, 2:3, estimate, conf.low, conf.high, p.value) %>%
## arredonde as casas decimais
mutate(across(where(is.numeric), round, digits = 2))Abaixo é como o quadro de dados fica após a execução do código. Veja a página sobre Tabelas para apresentação para ideias de como converter essa tabela para um formato bonito em HTML (ex.: com flextable).
Pacote gtsummary
Abaixo, nós apresentamos o uso da função tbl_uvregression() do pacote gtsummary. Assim como na página sobre Tabelas descritivas, as funções do gtsummary fazem um bom trabalho executando estatísticas e produzindo resultados com aparência profissional. A função tbl_uvregression() produz uma tabela com os resultados de uma regressão univariada.
Primeiro, nós selecionamos apenas as colunas de interesse do objeto linelist (variáveis explanatórias e a variável de evolução clínica [outcome]), e as aplicamos na função tbl_uvregression(). Então, iremos executar uma regressão univariada em cada uma das colunas definidas no vetor explanatory_vars, previamente criado na seção sobre Preparação dos dados (colunas gender, fever, chills, cough, aches, vomit, e age_cat).
Dentro da função, os atributos serão modificados, como em method = ao glm (sem aspas), o y = com a coluna de evolução dos casos (outcome), especificar para o method.args = que queremos rodar uma regressão logística através do atributo family = binomial, e então finalizamos com um comando para realizar a exponenciação dos resultados.
O resultado é gerado no formado HTML, e contém as contagens
univ_tab <- linelist %>%
dplyr::select(explanatory_vars, outcome) %>% ## selecione as variáveis de interesse
tbl_uvregression( ## produz uma tabela univariável
method = glm, ## define qual regressão será rodada (glm)
y = outcome, ## define a variável da evolução clínica (outcome)
method.args = list(family = binomial), ## define qual tipo de glm será rodador (logístico)
exponentiate = TRUE ## realiza a exponenciação para produzir as probabilidades (em vez de probabilidaes em log)
)
## visualize a tabela com os resultados da análise univariada
univ_tabCharacteristic |
N |
OR 1 |
95% CI 1 |
p-value |
|---|---|---|---|---|
| gender | 4,167 | 1.00 | 0.88, 1.13 | >0.9 |
| fever | 4,167 | 1.00 | 0.85, 1.17 | >0.9 |
| chills | 4,167 | 1.03 | 0.89, 1.21 | 0.7 |
| cough | 4,167 | 1.15 | 0.97, 1.37 | 0.11 |
| aches | 4,167 | 0.93 | 0.76, 1.14 | 0.5 |
| vomit | 4,167 | 1.09 | 0.96, 1.23 | 0.2 |
| age_cat | 4,167 | |||
| 0-4 | — | — | ||
| 5-9 | 0.94 | 0.77, 1.15 | 0.5 | |
| 10-14 | 1.15 | 0.93, 1.42 | 0.2 | |
| 15-19 | 0.99 | 0.80, 1.24 | >0.9 | |
| 20-29 | 1.03 | 0.84, 1.26 | 0.8 | |
| 30-49 | 1.07 | 0.85, 1.33 | 0.6 | |
| 50-69 | 0.68 | 0.41, 1.13 | 0.13 | |
| 70+ | 0.53 | 0.07, 3.20 | 0.5 | |
|
1
OR = Odds Ratio, CI = Confidence Interval |
||||
Exitem muitas modificações que podem ser feitas com a tabela gerada, como ajustar os rótulos em texto, destacar linhas pelo seu valor de p, etc. Veja tutoriais aqui e em outras fontes online.
19.3 Análise estratificada
A seção sobre análise estratificada ainda está sendo trabalhada no gtsummary. Esta página será atualizada quando possível.
19.4 Multivariada
Para a análise multivariada, novamente apresentamos duas abordagens:
-
glm()etidy()
- pacote gtsummary
O fluxo de trabalho é similar para cada uma das abordagens, sendo apenas a última diferente quando a tabela final é obtida.
Conduza a análise multivariada
Aqui nós utilizamos a função glm(), mas adicionaremos mais variáveis no lado direito da equação, separadas pelos símbolos de mais (+).
Para rodar o modelo com todas as nossas variáveis exploratórias, nós executamos o seguinte código:
mv_reg <- glm(outcome ~ gender + fever + chills + cough + aches + vomit + age_cat, family = "binomial", data = linelist)
summary(mv_reg)
Call:
glm(formula = outcome ~ gender + fever + chills + cough + aches +
vomit + age_cat, family = "binomial", data = linelist)
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) 0.069054 0.131726 0.524 0.600
gender 0.002448 0.065133 0.038 0.970
fever 0.004309 0.080522 0.054 0.957
chills 0.034112 0.078924 0.432 0.666
cough 0.138584 0.089909 1.541 0.123
aches -0.070705 0.104078 -0.679 0.497
vomit 0.086098 0.062618 1.375 0.169
age_cat5-9 -0.063562 0.101851 -0.624 0.533
age_cat10-14 0.136372 0.107275 1.271 0.204
age_cat15-19 -0.011074 0.113640 -0.097 0.922
age_cat20-29 0.026552 0.102780 0.258 0.796
age_cat30-49 0.059569 0.116402 0.512 0.609
age_cat50-69 -0.388964 0.262384 -1.482 0.138
age_cat70+ -0.647443 0.917375 -0.706 0.480
(Dispersion parameter for binomial family taken to be 1)
Null deviance: 5712.4 on 4166 degrees of freedom
Residual deviance: 5700.2 on 4153 degrees of freedom
AIC: 5728.2
Number of Fisher Scoring iterations: 4Se você quiser incluir duas variáveis e uma interação entre elas, é possível separá-las com um asterisco * em vez do +. Separe eles com dois pontos : se você está especificando apenas a interação. Por exemplo:
glm(outcome ~ gender + age_cat * fever, family = "binomial", data = linelist)Opcionalmente, você pode utilizar este código para nivelar o vetor pré-definido com os nomes das colunas, e re-criar o comando acima utilizando a função str_c(). Isto pode ser útil caso os nomes das suas variáveis explicativas estiverem mudando, ou se você não quiser digitar todas elas novamente.
## rode uma regressão com todas as variáveis de interesse
mv_reg <- explanatory_vars %>% ## inicie com um vetor contendo o nome das colunas explicativas
str_c(collapse = "+") %>% ## combine todos os nomes das variáveis de interesse separados por um 'mais'
str_c("outcome ~ ", .) %>% ## combine os nomes das variáveis de interesse com o 'outcome' no estilo de fórmula
glm(family = "binomial", ## defina o tipo de glm como logístico,
data = linelist) ## defina seu banco de dadosConstruíndo o modelo
É possível construir seu modelo passo a passo, salvando diferentes modelos que incluem certas variáveis explicativas. Esses modelos podem ser comparados com os testes de probabilidade utilizando a função lrtest(), do pacote lmtest, como mostrado abaixo:
NOTA: Utilizar o teste anova(model1, model2, test = "Chisq) do R base produz os mesmos resultados
model1 <- glm(outcome ~ age_cat, family = "binomial", data = linelist)
model2 <- glm(outcome ~ age_cat + gender, family = "binomial", data = linelist)
lmtest::lrtest(model1, model2)Likelihood ratio test
Model 1: outcome ~ age_cat
Model 2: outcome ~ age_cat + gender
#Df LogLik Df Chisq Pr(>Chisq)
1 8 -2852.6
2 9 -2852.6 1 2e-04 0.9883Outra opção é utilizar o objeto modelado diretamente na função step(), do pacote stats. Especifique qual a direção da seleção das variáveis que quer utilizar quando for construir o modelo.
## escolha um modelo utilizando a seleção 'foward' baseada no AIC
## você também pode escolher "backward" ou "both" ao ajustar a direção
final_mv_reg <- mv_reg %>%
step(direction = "forward", trace = FALSE)Para facilitar a visualização, é possível desativar a notação científica na sua sessão do R:
options(scipen=999)Como descrito na seção sobre análise univariada, aplique o resultado da modelagem na função tidy() para potencializar as probabilidades em log e os intervalos de confiança. Finalmente, todas as colunas numéricas são arredondadads para duas casas decimais. Role o cursor para visualizar todas as linhas.
mv_tab_base <- final_mv_reg %>%
broom::tidy(exponentiate = TRUE, conf.int = TRUE) %>% ## obtenha um quadro de dados organizado das estimativas
mutate(across(where(is.numeric), round, digits = 2)) ## arredonde Aqui está o quadro de dados final:
Combine as análises univariadas e multivariadas
Combine com o gtsummary
O pacote gtsummary possui a função tbl_regression(), que utiliza os resultados de uma regressão (glm() neste caso) e produz uma linda tabela resumo.
## mostra a tabela de resultados de uma regressão
mv_tab <- tbl_regression(final_mv_reg, exponentiate = TRUE)Vamos visualizar a tabela:
mv_tabCharacteristic |
OR 1 |
95% CI 1 |
p-value |
|---|---|---|---|
| gender | 1.00 | 0.88, 1.14 | >0.9 |
| fever | 1.00 | 0.86, 1.18 | >0.9 |
| chills | 1.03 | 0.89, 1.21 | 0.7 |
| cough | 1.15 | 0.96, 1.37 | 0.12 |
| aches | 0.93 | 0.76, 1.14 | 0.5 |
| vomit | 1.09 | 0.96, 1.23 | 0.2 |
| age_cat | |||
| 0-4 | — | — | |
| 5-9 | 0.94 | 0.77, 1.15 | 0.5 |
| 10-14 | 1.15 | 0.93, 1.41 | 0.2 |
| 15-19 | 0.99 | 0.79, 1.24 | >0.9 |
| 20-29 | 1.03 | 0.84, 1.26 | 0.8 |
| 30-49 | 1.06 | 0.85, 1.33 | 0.6 |
| 50-69 | 0.68 | 0.40, 1.13 | 0.14 |
| 70+ | 0.52 | 0.07, 3.19 | 0.5 |
|
1
OR = Odds Ratio, CI = Confidence Interval |
|||
Também é possível combinar diferentes tabelas de resultados produzidas pelo gtsummary com a função tbl_merge(). Assim, podemos combinar os resultados multivariados com os resultados univariados do gtsummary que criamos acima:
## combine com os resultados univariados
tbl_merge(
tbls = list(univ_tab, mv_tab), # combine as tabelas
tab_spanner = c("**Univariate**", "**Multivariable**")) # escolha o nome dos cabeçalhosCharacteristic |
Univariate |
Multivariable |
|||||
|---|---|---|---|---|---|---|---|
N |
OR 1 |
95% CI 1 |
p-value |
OR 1 |
95% CI 1 |
p-value |
|
| gender | 4,167 | 1.00 | 0.88, 1.13 | >0.9 | 1.00 | 0.88, 1.14 | >0.9 |
| fever | 4,167 | 1.00 | 0.85, 1.17 | >0.9 | 1.00 | 0.86, 1.18 | >0.9 |
| chills | 4,167 | 1.03 | 0.89, 1.21 | 0.7 | 1.03 | 0.89, 1.21 | 0.7 |
| cough | 4,167 | 1.15 | 0.97, 1.37 | 0.11 | 1.15 | 0.96, 1.37 | 0.12 |
| aches | 4,167 | 0.93 | 0.76, 1.14 | 0.5 | 0.93 | 0.76, 1.14 | 0.5 |
| vomit | 4,167 | 1.09 | 0.96, 1.23 | 0.2 | 1.09 | 0.96, 1.23 | 0.2 |
| age_cat | 4,167 | ||||||
| 0-4 | — | — | — | — | |||
| 5-9 | 0.94 | 0.77, 1.15 | 0.5 | 0.94 | 0.77, 1.15 | 0.5 | |
| 10-14 | 1.15 | 0.93, 1.42 | 0.2 | 1.15 | 0.93, 1.41 | 0.2 | |
| 15-19 | 0.99 | 0.80, 1.24 | >0.9 | 0.99 | 0.79, 1.24 | >0.9 | |
| 20-29 | 1.03 | 0.84, 1.26 | 0.8 | 1.03 | 0.84, 1.26 | 0.8 | |
| 30-49 | 1.07 | 0.85, 1.33 | 0.6 | 1.06 | 0.85, 1.33 | 0.6 | |
| 50-69 | 0.68 | 0.41, 1.13 | 0.13 | 0.68 | 0.40, 1.13 | 0.14 | |
| 70+ | 0.53 | 0.07, 3.20 | 0.5 | 0.52 | 0.07, 3.19 | 0.5 | |
|
1
OR = Odds Ratio, CI = Confidence Interval |
|||||||
Combine com o pacote dplyr
Uma alternativa para combinar os resultados univariados e multivariados do glm()/tidy() é com as funções de união do dplyr.
- Una os resultados univariados anteriores (
univ_tab_base, com contagens) com os resultados multivariados organizadosmv_tab_base
- Use a função
select()para manter apenas as colunas de interesse, especificar a sua ordem, e renomear elas - Use a função
round()com duas casas decimais em todas as colunas da classe Double
## combine tabelas univariadas e multivariadas
left_join(univ_tab_base, mv_tab_base, by = "term") %>%
## escolha as colunas e as renomeie
select( # new name = old name
"characteristic" = term,
"recovered" = "0",
"dead" = "1",
"univ_or" = estimate.x,
"univ_ci_low" = conf.low.x,
"univ_ci_high" = conf.high.x,
"univ_pval" = p.value.x,
"mv_or" = estimate.y,
"mvv_ci_low" = conf.low.y,
"mv_ci_high" = conf.high.y,
"mv_pval" = p.value.y
) %>%
mutate(across(where(is.double), round, 2)) # A tibble: 20 × 11
characteristic recovered dead univ_or univ_ci_low univ_ci_high univ_pval
<chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 (Intercept) 909 1168 1.28 1.18 1.4 0
2 gender 916 1174 1 0.88 1.13 0.97
3 (Intercept) 340 436 1.28 1.11 1.48 0
4 fever 1485 1906 1 0.85 1.17 0.99
5 (Intercept) 1472 1877 1.28 1.19 1.37 0
6 chills 353 465 1.03 0.89 1.21 0.68
7 (Intercept) 272 309 1.14 0.97 1.34 0.13
8 cough 1553 2033 1.15 0.97 1.37 0.11
9 (Intercept) 1636 2114 1.29 1.21 1.38 0
10 aches 189 228 0.93 0.76 1.14 0.51
11 (Intercept) 931 1144 1.23 1.13 1.34 0
12 vomit 894 1198 1.09 0.96 1.23 0.17
13 (Intercept) 338 427 1.26 1.1 1.46 0
14 age_cat5-9 365 433 0.94 0.77 1.15 0.54
15 age_cat10-14 273 396 1.15 0.93 1.42 0.2
16 age_cat15-19 238 299 0.99 0.8 1.24 0.96
17 age_cat20-29 345 448 1.03 0.84 1.26 0.79
18 age_cat30-49 228 307 1.07 0.85 1.33 0.58
19 age_cat50-69 35 30 0.68 0.41 1.13 0.13
20 age_cat70+ 3 2 0.53 0.07 3.2 0.49
# ℹ 4 more variables: mv_or <dbl>, mvv_ci_low <dbl>, mv_ci_high <dbl>,
# mv_pval <dbl>19.5 Gráfico em floresta (Forest Plot)
Esta seção mostra como produzir um gráfico com os resultados da sua regressão. Existem duas opções. Você pode construir uma plotagem utilizando o ggplot2 ou utilizando um pacote-meta (um pacote que incluí muitos pacotes) chamado easystats.
Veja a página sobre básico do ggplot se você não é familiar com o pacote de plotagem ggplot2.
Pacote ggplot2
É possível construir um gráfico em floresta com a função ggplot() ao plotar os resultados de uma regressão multivariada. Adicione as camadas das plotagens com estas funções “geoms”:
- realize estimativas com
geom_point()
- obtenha intervalos de confiança com
geom_errorbar()
- uma linha vertical em OU = 1 com
geom_vline()
Antes de traçar o gráfico, é interessante utilizar a função fct_relevel(), do pacote forcats, para escolher a ordem das variáveis/níveis no eixo y. ggplot() pode mostrar elas em uma ordem alfa-numérica que pode não funcionar bem com os valores da variável ‘age category’ (“30” apareceria antes de “5”). Veja a página sobre fatores para mais detalhes.
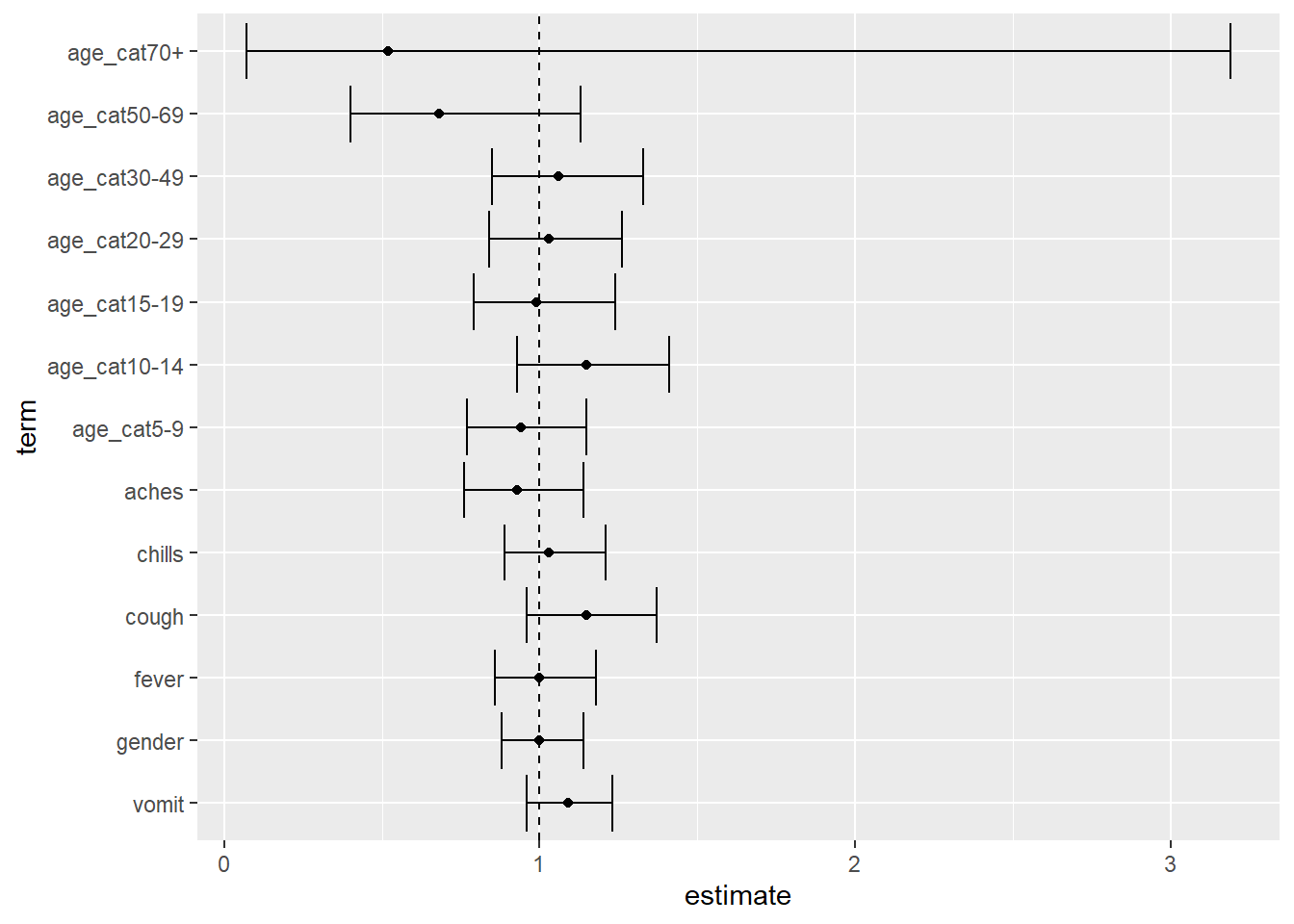
## remove o termo da intercepção dos seus resultados multivariados
mv_tab_base %>%
# escolhe a ordem dos níveis que aparecem no eixo y
mutate(term = fct_relevel(
term,
"vomit", "gender", "fever", "cough", "chills", "aches",
"age_cat5-9", "age_cat10-14", "age_cat15-19", "age_cat20-29",
"age_cat30-49", "age_cat50-69", "age_cat70+")) %>%
# remove a linha "intercept" do gráfico
filter(term != "(Intercept)") %>%
## trace um gráfico no eixo y e/ou as estimativas no eixo x
ggplot(aes(x = estimate, y = term)) +
## mostre a estimativa como um ponto
geom_point() +
## adicione uma barra de erro para os intervalos de confiança
geom_errorbar(aes(xmin = conf.low, xmax = conf.high)) +
## mostre a linha. OR = 1 é referência para uma linha tracejada
geom_vline(xintercept = 1, linetype = "dashed")Pacotes easystats
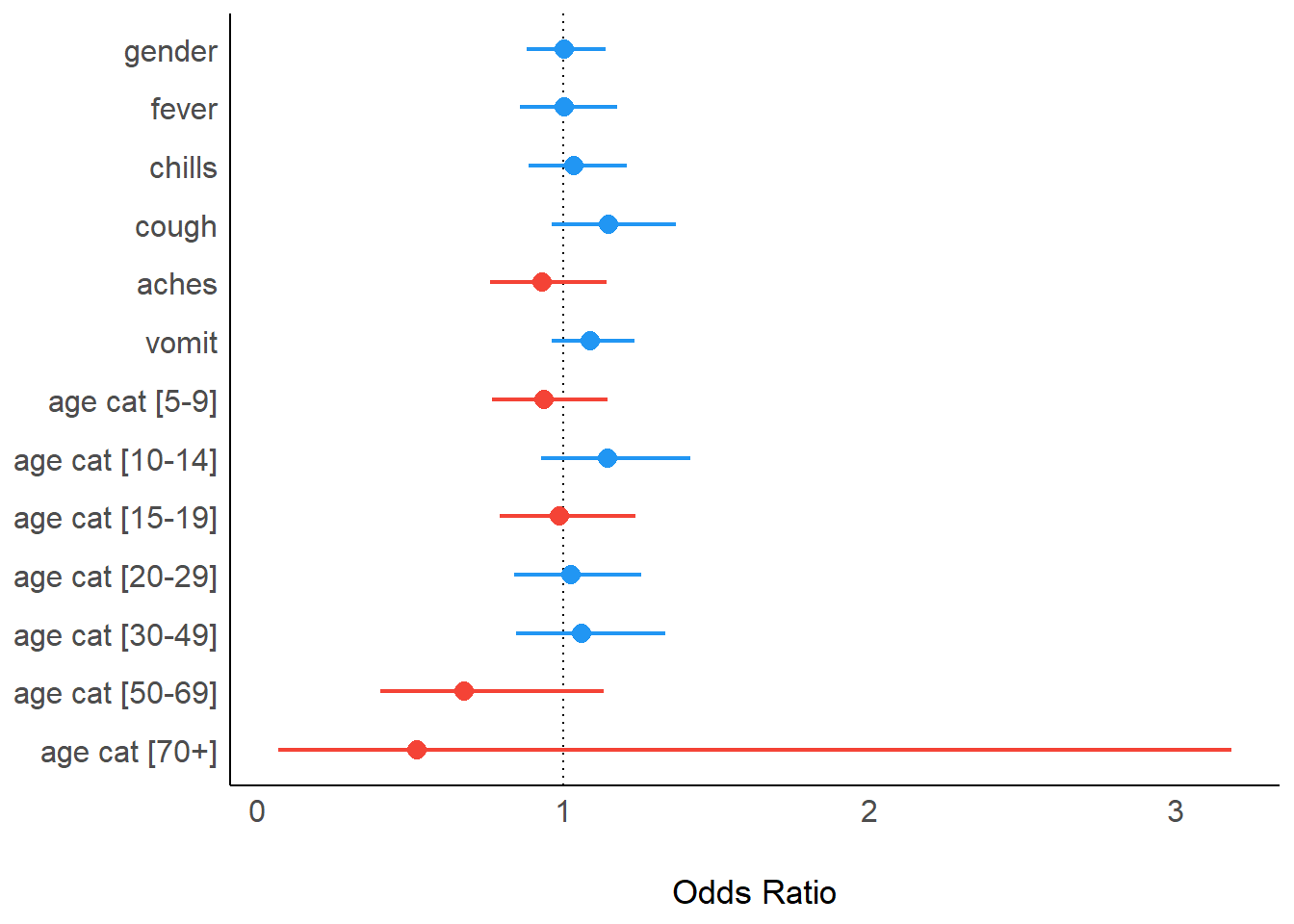
Uma alternativa, caso você não queira realizar os ajustes finos no ggplot2, é utilizar uma combinação dos pacotes do easystats.
A função model_parameters(), do pacote parameters, faz o equivalente da função tidy() do pacote broom. O pacote see aceita estes resultados e cria uma plotagem em floresta padrão, como se fosse um objeto ggplot().
19.6 Recursos
O conteúdo desta página foi adaptado destes recursos e tutoriais onlines: