pacman::p_load(
rio, # importa/exporta
here, # localizador de arquivos
purrr, # iteração
grates, # scales in ggplot
tidyverse # gerenciamento e visualização de dados
)16 Iterações, loops e listas
Os epidemiologistas muitas vezes se deparam com análises repetidas em subgrupos, como países, distritos ou faixas etárias. Estas são apenas algumas das muitas situações envolvendo iteração. Codificar suas operações iterativamente usando as abordagens abaixo ajudará você a executar essas tarefas repetitivas mais rapidamente, reduzir a chance de erro e reduzir o comprimento do código.
Esta página apresentará duas abordagens para operações iterativas - usando os loops for e usando o pacote purrr.
- loops for iteram código a partir de uma série de entradas, mas são menos comuns em R do que em outras linguagens de programação. No entanto, nós os apresentamos aqui como uma ferramenta de aprendizagem e referência
- O pacote purrr é a abordagem tidyverse para operações iterativas - ele funciona “mapeando” uma função em muitas entradas (valores, colunas, conjuntos de dados etc.)
Ao longo do caminho, mostraremos exemplos como:
- Importando e exportando vários arquivos
- Criando curvas epidemiológicas para várias jurisdições
- Executando testes T para várias colunas em um data frame
Na seção purrr também forneceremos vários exemplos de criação e manipulação de listas.
16.1 Preparação
Carregando pacotes
Este trecho de código mostra o carregamento de pacotes necessários para as análises. Neste livro nós enfatizamos o p_load() do pacman, que instala o pacote, se necessário, e o carrega para uso. Você também pode carregar pacotes instalados com o library() do R base. Veja a página sobre o [R - o básico] para mais informações sobre pacotes R.
Importando dados
Importamos o conjunto de dados de casos de uma epidemia simulada de Ebola. Se você quiser acompanhar clique aqui para fazer download da linelist “limpa” (como um arquivo .rds). Importamos os dados com a função import() do pacote rio (ela lida com muitos tipos de arquivos como .xlsx, .csv, .rds - veja a página Importar e exportar para detalhes).
# importando a linelist
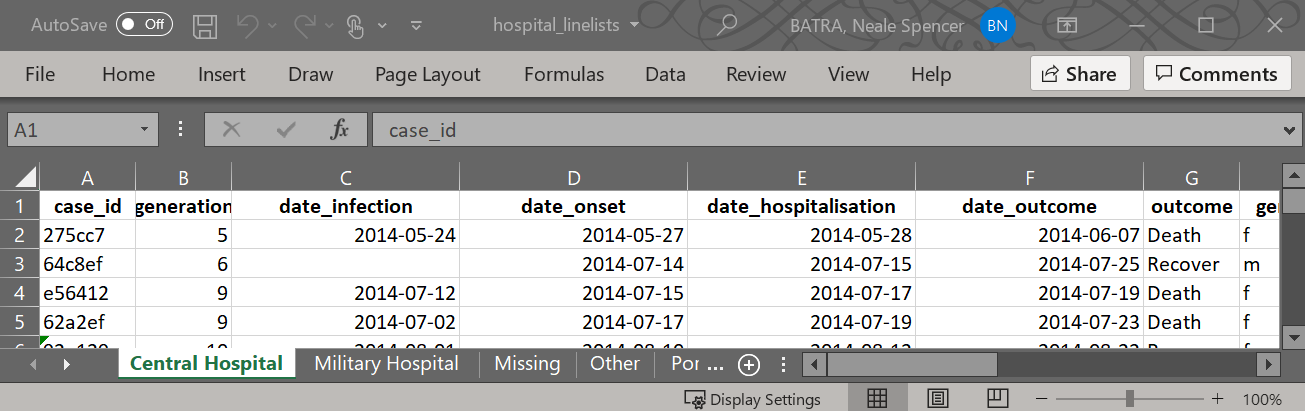
linelist <- import("linelist_cleaned.rds")As primeiras 50 linhas da linelist são exibidas abaixo.
16.2 loops for
loops for no R
loops for não são enfatizados em R, mas são comuns em outras linguagens de programação. Para iniciantes, eles podem ser úteis para aprender e praticar porque são mais fáceis de “explorar”, “depurar” e entender exatamente o que está acontecendo para cada iteração, especialmente quando você ainda não está confortável em escrever suas próprias funções .
Você pode passar rapidamente do loop for para iterar com funções mapeadas com o purrr (consulte a seção abaixo)
Principais componentes
Um loop for tem três partes principais:
- A sequência de itens para percorrer
- As operações a serem conduzidas por item na sequência
- O contêiner para armazenar os resultados (opcional)
A sintaxe básica é: for (item em sequência) {fazer operações usando item}. Observe os parênteses e os colchetes. Os resultados podem ser impressos no console ou armazenados em um objeto R (o container).
Um exemplo simples de loop for está abaixo.
for (num in c(1,2,3,4,5)) { # a SEQUÊNCIA é definida (números 1 a 5) e o loop é aberto com "{"
print(num + 2) # As OPERAÇÕES (adicione dois a cada número de sequência e imprima)
} # O loop é fechado com "}" [1] 3
[1] 4
[1] 5
[1] 6
[1] 7Sequência
Esta é a parte “for” de um loop for - as operações serão executadas “para” cada item na sequência. A sequência pode ser uma série de valores (por exemplo, nomes de jurisdições, doenças, nomes de colunas, elementos de lista, etc.), ou pode ser uma série de números consecutivos (por exemplo, 1,2,3,4,5). Cada abordagem tem suas próprias utilidades, descritas abaixo.
A estrutura básica de uma instrução de sequência é item em vetor.
- Você pode escrever qualquer caractere ou palavra no lugar de “item” (por exemplo, “i”, “num”, “hosp”, “distrito”, etc.). O valor desse “item” muda a cada iteração do loop, passando por cada valor no vetor.
- O vetor pode ser de valores do tipo caracteres, nomes de colunas ou talvez uma sequência de números - esses são os valores que mudarão a cada iteração. Você pode usá-los nas operações do loop for usando o termo “item”.
Exemplo: sequência de valores de caracteres
Neste exemplo, um loop é executado para cada valor em um vetor de caracteres predefinido de nomes de hospitais.
# criando um vetor com os nomes dos hospitais
hospital_names <- unique(linelist$hospital)
hospital_names # printe (mostre o nome do hospital)[1] "Other"
[2] "Ausente"
[3] "St. Mark's Maternity Hospital (SMMH)"
[4] "Port Hospital"
[5] "Military Hospital"
[6] "Central Hospital" Escolhemos o termo hosp para representar valores do vetor hospital_names. Para a primeira iteração do loop, o valor de hosp será hospital_names[[1]]. Para o segundo loop será hospital_names[[2]]. E assim por diante…
# um 'loop for' com sequência de caracteres
for (hosp in hospital_names){ # sequência
# OPERAÇÕES AQUI
}Exemplo: sequência de nomes de colunas
Esta é uma variação da sequência de caracteres acima, na qual os nomes de um objeto R existente são extraídos e se tornam o vetor. Por exemplo, os nomes das colunas de um data frame. Convenientemente, no código de operações do loop for, os nomes das colunas podem ser usados para indexar (subconjunto) seu data frame original.
Abaixo, a sequência é o names() (nomes das colunas) do data frame linelist. Nosso nome de “item” é col, que representará o nome de cada coluna à medida que os loops prosseguem.
Para fins de exemplo, incluímos o código de operações dentro do loop for, que é executado para cada valor na sequência. Neste código, os valores de sequência (nomes das colunas) são usados para indexar (subconjunto) linelist, um de cada vez. Conforme ensinado na página Introdução ao R, colchetes duplos [[ ]] são usados para subconjunto. A coluna resultante é passada para is.na(), então para sum() para produzir o número de valores na coluna que estão faltando. O resultado é impresso no console - um número para cada coluna.
for (col in names(linelist)){ # o loop é executado para cada coluna na linelist; nome da coluna representado por "col"
# Exemplo de código de operações - printe o número de valores ausentes na coluna
print(sum(is.na(linelist[[col]]))) # linelist é indexado pelo valor atual de "col"
}[1] 0
[1] 0
[1] 2087
[1] 256
[1] 0
[1] 936
[1] 1323
[1] 278
[1] 86
[1] 0
[1] 86
[1] 86
[1] 86
[1] 0
[1] 0
[1] 0
[1] 2088
[1] 2088
[1] 0
[1] 0
[1] 0
[1] 249
[1] 249
[1] 249
[1] 249
[1] 249
[1] 149
[1] 765
[1] 0
[1] 256Sequência de números
Nesta abordagem, a sequência é uma série de números consecutivos. Assim, o valor do “item” não é um valor de caractere (por exemplo, “Hospital Central” ou “data_onset”), mas é um número. Isso é útil para fazer loop pelos data frames, pois você pode usar o número do “item” dentro do loop for para indexar o data frame pelo número da linha.
Por exemplo, digamos que você queira percorrer cada linha em seu data frame e extrair determinadas informações. Seus “itens” seriam números de linha numéricos. Frequentemente, “itens” neste caso são escritos como i.
O processo loop for pode ser explicado em palavras como “para cada item em uma sequência de números de 1 ao número total de linhas no meu data frame, faça X”. Para a primeira iteração do loop, o valor de “item” i seria 1. Para a segunda iteração, i seria 2, etc.
Aqui está a aparência da sequência no código: for (i in 1:nrow(linelist)) {OPERATIONS CODE} onde i representa o “item” e 1:nrow(linelist) produz uma sequência de números de 1 até o número de linhas em linelist.
for (i in 1:nrow(linelist)) { # use em um data frame
# OPERAÇÕES AQUI
} Se você deseja que a sequência seja de números, mas está começando de um vetor (não de um data frame), use o atalho seq_along() para retornar uma sequência de números para cada elemento do vetor. Por exemplo, for (i in seq_along(hospital_names) {OPERATIONS CODE}.
O código abaixo na verdade retorna números, que se tornariam o valor de i em seu respectivo loop.
seq_along(hospital_names) # use em um vetor de nomes[1] 1 2 3 4 5 6Uma vantagem de usar números na sequência é que é fácil também usar o número i para indexar um contêiner que armazena as saídas do loop. Há um exemplo disso na seção Operações abaixo.
Operações
Este é o código dentro das chaves { } do loop for. Você deseja que esse código seja executado para cada “item” na sequência. Portanto, tome cuidado para que cada parte do seu código que muda pelo “item” seja codificada corretamente de forma que realmente mude! Por exemplo, lembre-se de usar [[ ]] para indexação.
No exemplo abaixo, iteramos em cada linha na linelist. Os valores gender e age de cada linha são colados e armazenados no vetor de caracteres do contêiner cases_demographics. Observe como também usamos a indexação [[i]] para salvar a saída do loop na posição correta no vetor “contêiner”.
# criando contêiner para armazenar resultados - um vetor de caractere
cases_demographics <- vector(mode = "character", length = nrow(linelist))
# o loop for
for (i in 1:nrow(linelist)){
# OPERAÇÕES
# extraindo valores da linelist para a linha i, usando colchetes para indexação
row_gender <- linelist$gender[[i]]
row_age <- linelist$age_years[[i]] # não se esqueça de indexar!
# combinando gender-age e armazenar no vetor de contêiner no local indexado
cases_demographics[[i]] <- str_c(row_gender, row_age, sep = ",")
} # finalizando o loop for
# exibindo as primeiras 10 linhas do contêiner
head(cases_demographics, 10) [1] "m,2" "f,3" "m,56" "f,18" "m,3" "f,16" "f,16" "f,0" "m,61" "f,27"Contêiner
Às vezes, os resultados do seu loop for serão impressos no console ou no painel RStudio Plots. Outras vezes, você desejará armazenar as saídas em um “contêiner” para uso posterior. Esse contêiner pode ser um vetor, um data frame ou até mesmo uma lista.
É mais eficiente criar o contêiner para os resultados antes mesmo de iniciar o loop. Na prática, isso significa criar um vetor, data frame ou lista vazio. Estes podem ser criados com as funções vector() para vetores ou listas, ou com matrix() e data.frame() para um data frame.
Vetor vazio
Use vector() e especifique o mode = com base na classe esperada dos objetos que você irá inserir - seja “double” (para armazenar números), “character” ou “logical”. Você também deve definir o length = com antecedência. Este deve ser o comprimento da sua sequência loop for.
Digamos que você queira armazenar o atraso médio até a admissão de cada hospital. Você usaria “double” e definiria o comprimento como o número de saídas esperadas (o número de hospitais exclusivos no conjunto de dados).
delays <- vector(
mode = "double", # esperamos armazenar números
length = length(unique(linelist$hospital))) # o número de hospitais únicos no conjunto de dadosData frame vazio
Você pode criar um data frame vazio especificando o número de linhas e colunas assim:
delays <- data.frame(matrix(ncol = 2, nrow = 3))Lista vazia
Você pode querer armazenar alguns gráficos criados por um loop for em uma lista. Uma lista é como um vetor, mas contém outros objetos R dentro dela que podem ser de diferentes classes. Os itens em uma lista podem ser um único número, um data frame, um vetor e até outra lista.
Você realmente inicializa uma lista vazia usando o mesmo comando vector() acima, mas com mode = "list". Especifique o comprimento como desejar.
plots <- vector(mode = "list", length = 16)Imprimindo
Observe que para “printar” (mostrar) de dentro de um loop for você provavelmente precisará envolver explicitamente a função print().
Neste exemplo abaixo, a sequência é um vetor de caracteres explícito, que é usado para acessar o subconjunto da linelist por hospital. Os resultados não são armazenados em um contêiner, mas são impressos no console com a função print().
for (hosp in hospital_names){
hospital_cases <- linelist %>% filter(hospital == hosp)
print(nrow(hospital_cases))
}[1] 885
[1] 1469
[1] 422
[1] 1762
[1] 896
[1] 454Testando o seu loop
Para testar seu loop, você pode executar um comando para fazer uma atribuição temporária do “item”, como i <- 10 ou hosp <- "Central Hospital". Faça isso fora do loop e execute apenas seu código de operações (o código entre colchetes) para ver se os resultados esperados são produzidos.
Gráficos em loop
Para juntar todos os três componentes (contêiner, sequência e operações), vamos tentar traçar uma epicurva para cada hospital (consulte a página em Curvas epidêmicas).
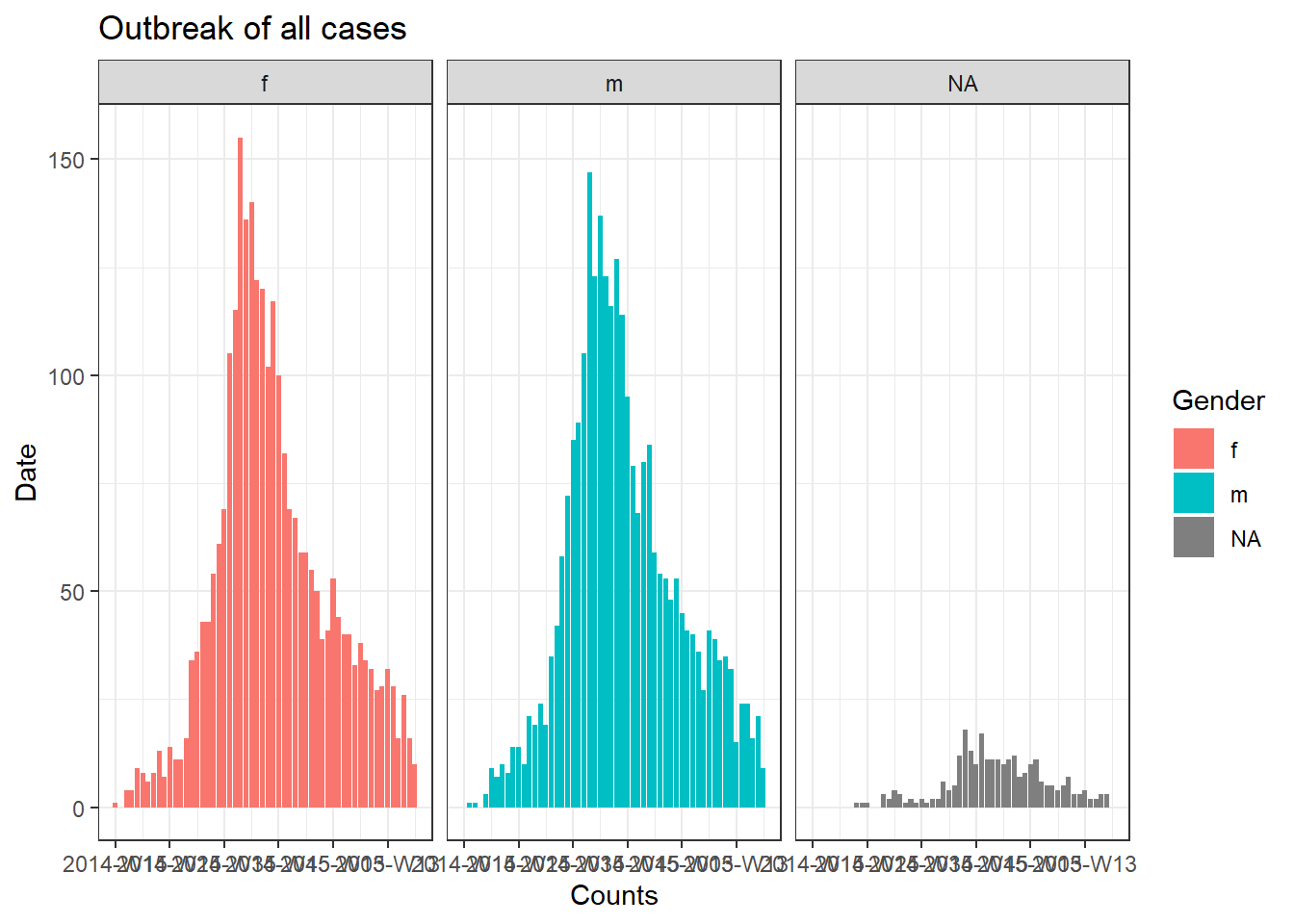
Podemos fazer uma bela epicurva de todos os casos por gênero usando o pacote incidence2 conforme abaixo:
# criando o objeto do tipo 'incidence'
outbreak <- incidence2::incidence(
x = linelist, # data frame - linelist completo
date_index = "date_onset", # coluna de data
interval = "week", # contagens agregadas semanalmente
groups = "gender") # valores de grupo por gênero
#na_as_group = TRUE) # o sexo ausente é um grupo próprio
# plotando a epicurva
ggplot(outbreak, # nom de l'objet d'incidence
aes(x = date_index, #aesthetiques et axes
y = count,
fill = gender), # Fill colour of bars by gender
color = "black" # Contour colour of bars
) +
geom_col() +
facet_wrap(~gender) +
theme_bw() +
labs(title = "Outbreak of all cases", #titre
x = "Counts",
y = "Date",
fill = "Gender",
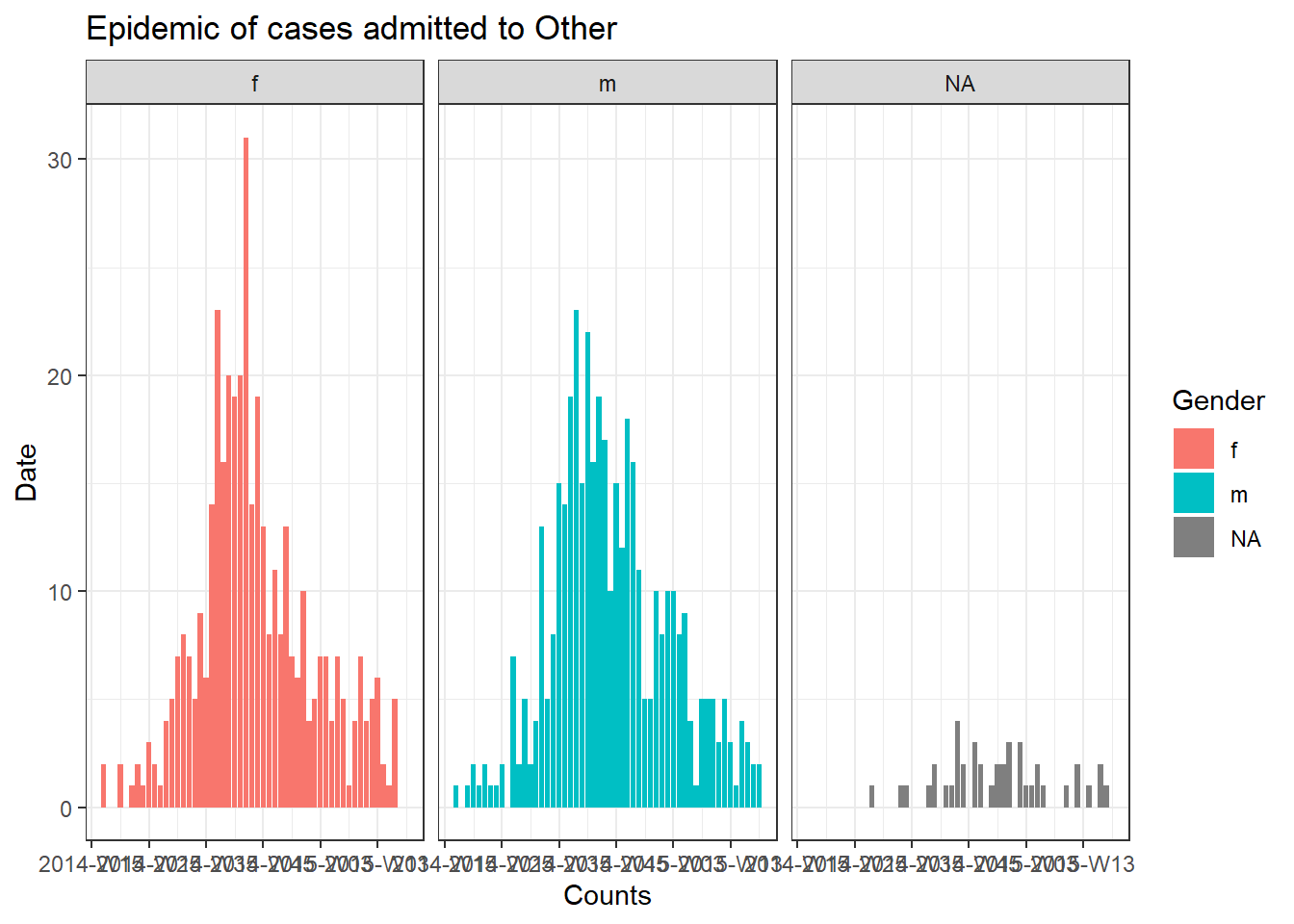
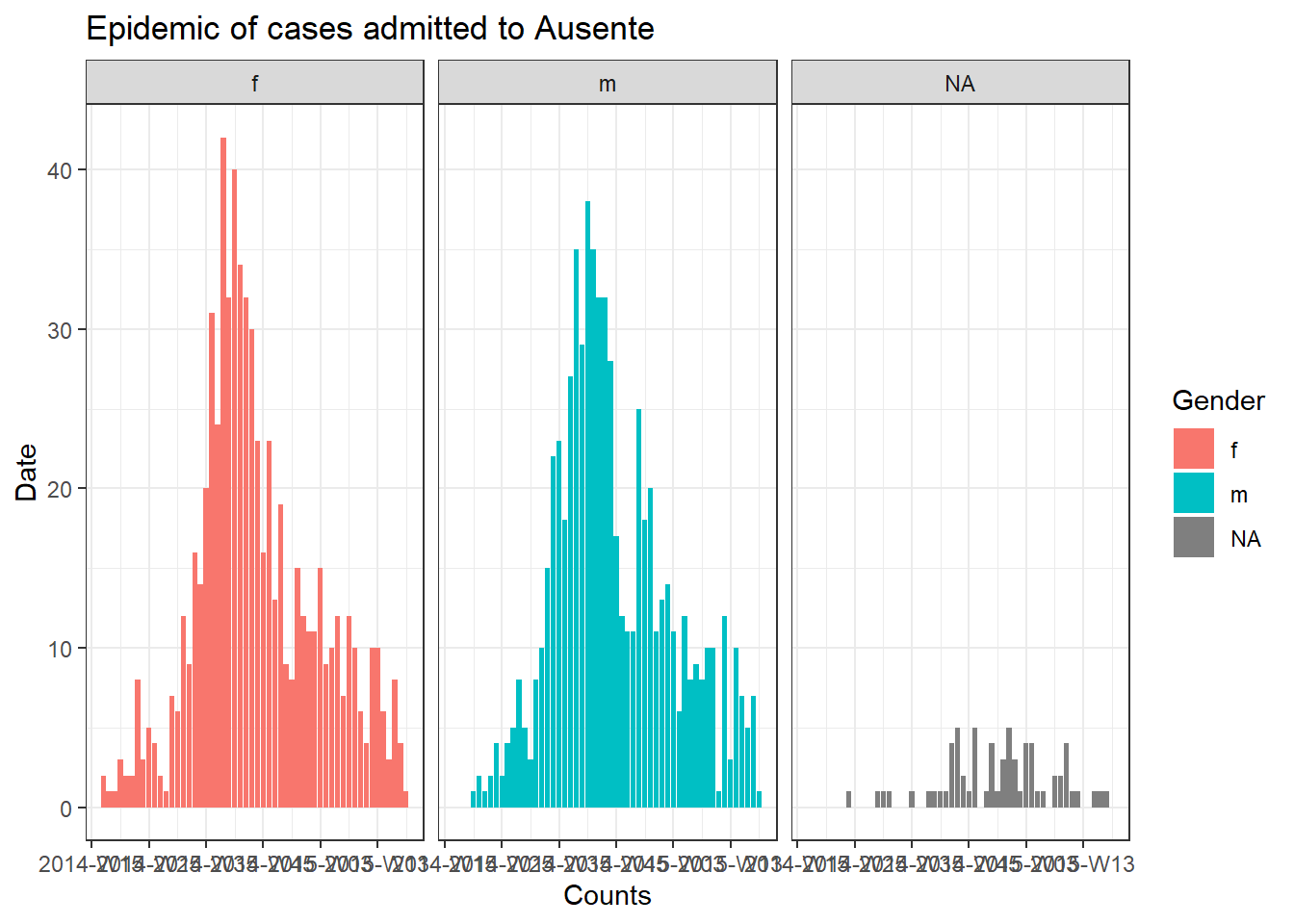
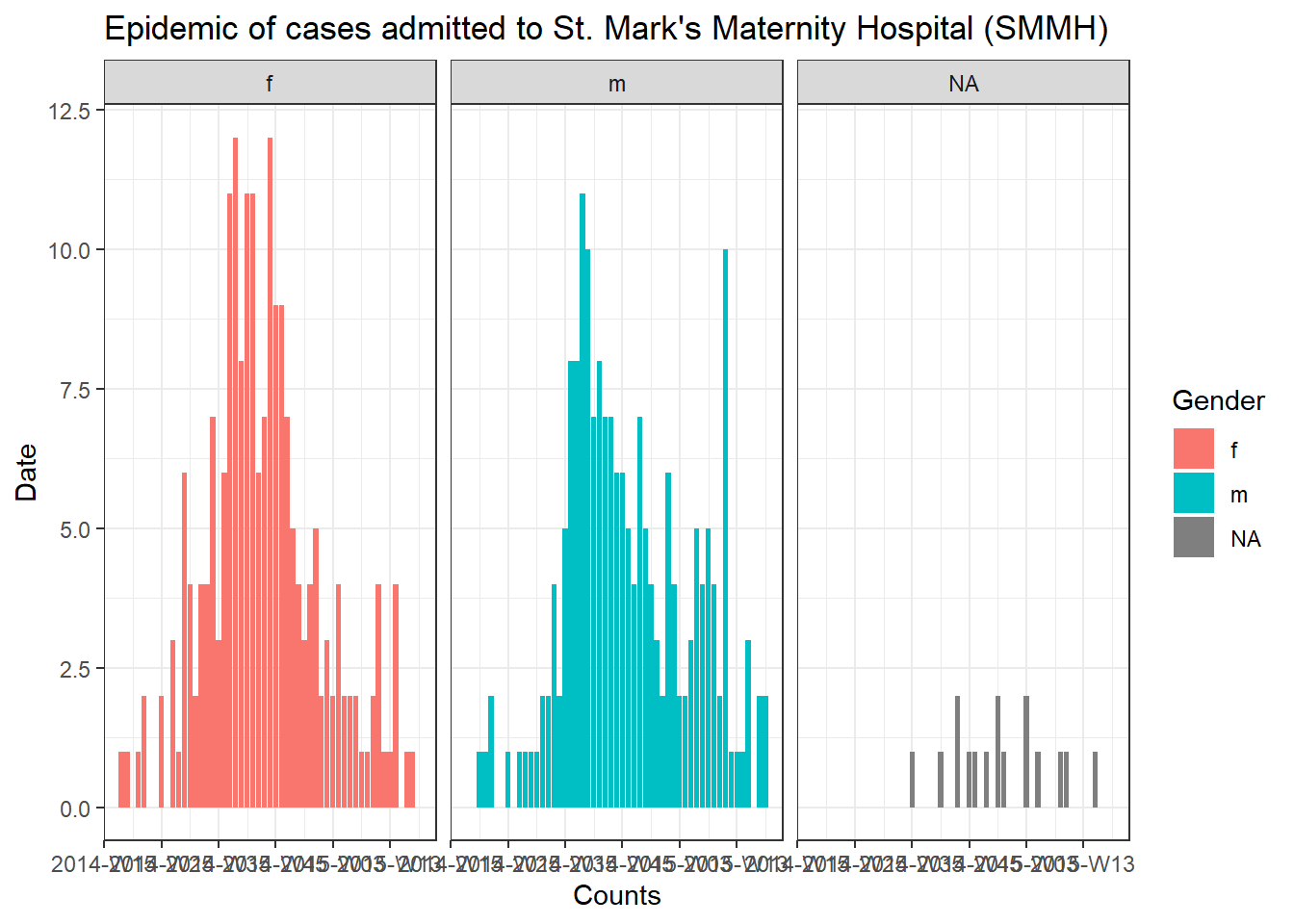
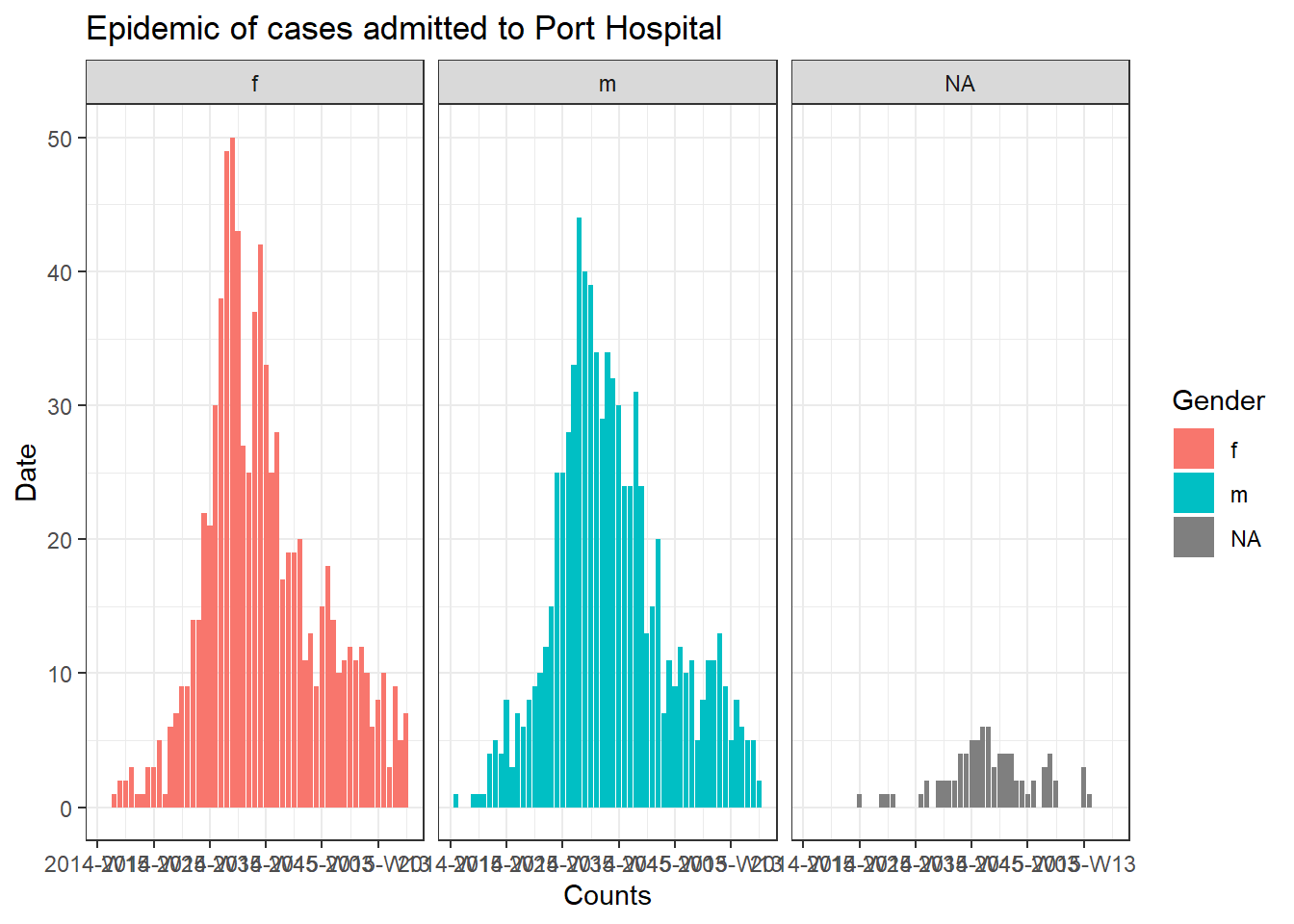
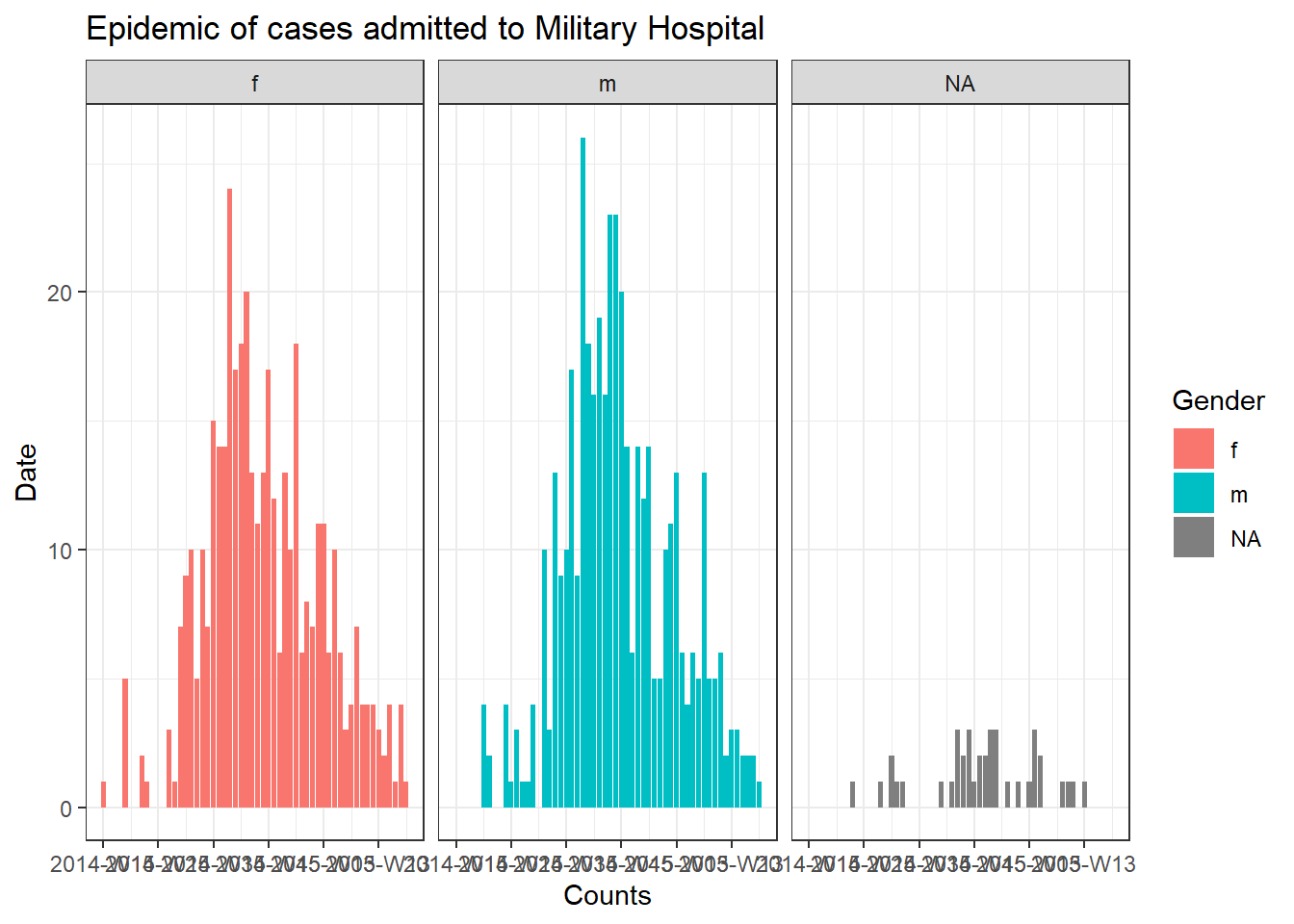
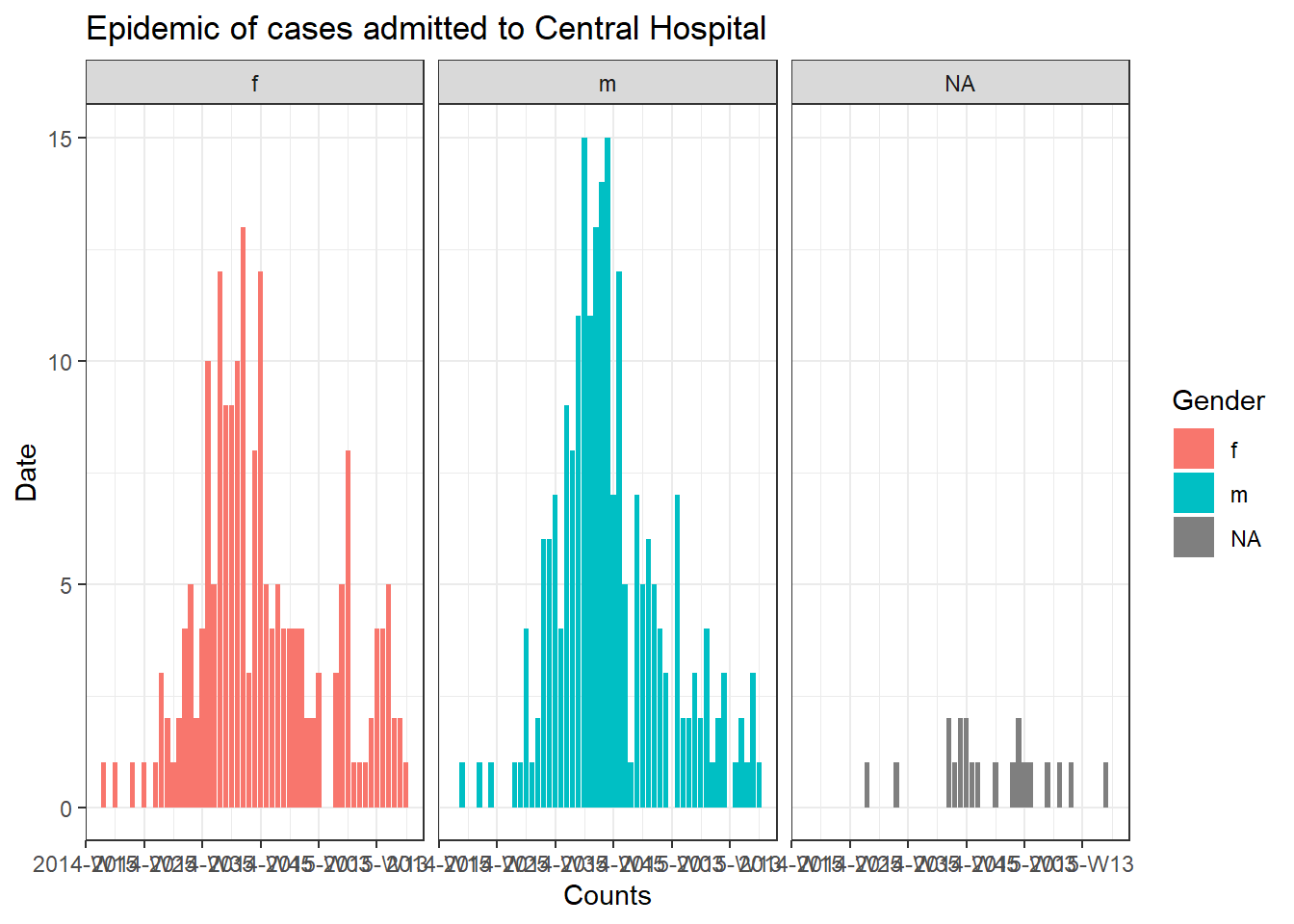
color = "Gender")Para produzir um gráfico separado para os casos de cada hospital, podemos colocar esse código da epicurva dentro de um loop for.
Primeiro, salvamos um vetor com os nomes exclusivos do hospital, hospital_names. O loop for será executado uma vez para cada um destes nomes: for (hosp in hospital_names). A cada iteração do loop for, o nome do hospital atual do vetor será representado como hosp para uso dentro do loop.
Dentro das operações de loop, você pode escrever o código R normalmente, mas use o “item” (hosp neste caso) sabendo que seu valor será alterado. Dentro deste loop:
- Um
filter()é aplicado alinelist, de modo que a colunahospitaldeve ser igual ao valor atual dehosp - O objeto do tipo incidence é criado na linelist filtrada
- O gráfico para o hospital atual é criado, com um título de ajuste automático que usa
hosp - O gráfico do hospital atual é salvo temporariamente e depois impresso
- O loop então avança para repetir com o próximo hospital em
hospital_names
# criando o vetor dos nomes dos hospitais
hospital_names <- unique(linelist$hospital)
# para cada nome ("hosp") em hospital_names, crie e imprima a epicurva
for (hosp in hospital_names) {
# criando objeto de incidência específico para o hospital atual
outbreak_hosp <- incidence2::incidence(
x = linelist %>% filter(hospital == hosp), # linelist é filtrada para o hospital atual
date_index = "date_onset",
interval = "week",
groups = "gender"#,
#na_as_group = TRUE
)
# Criando e salvando o gráfico. O título se ajusta automaticamente ao hospital atual
plot_hosp <- ggplot(outbreak_hosp, # incidence object name
aes(x = date_index, #axes
y = count,
fill = gender), # fill colour by gender
color = "black" # colour of bar contour
) +
geom_col() +
facet_wrap(~gender) +
theme_bw() +
labs(title = stringr::str_glue("Epidemic of cases admitted to {hosp}"), #title
x = "Counts",
y = "Date",
fill = "Gender",
color = "Gender")
# With older versions of R, remove the # before na_as_group and use this plot command instead.
# plot_hosp <- plot(
# outbreak_hosp,
# fill = "gender",
# color = "black",
# title = stringr::str_glue("Epidemic of cases admitted to {hosp}")
# )
# exibindo o gráfico para o hospital atual
print(plot_hosp)
} # encerrando o loop for quando ele tiver sido executado para todos os hospitais em hospital_namesAcompanhando o progresso de um loop
Um loop com muitas iterações pode ser executado por muitos minutos ou até horas. Assim, pode ser útil imprimir o progresso no console R. A instrução if (que significa “se” em inglês) abaixo pode ser colocada dentro das operações de loop para exibir a cada 100 números. Basta ajustá-lo para que i seja o “item” em seu loop.
# loop com código para exibir o progresso a cada 100 iterações
for (i in seq_len(nrow(linelist))){
# print progress
if(i %% 100==0){ # O operador %% calcula o resto da operação
print(i)
}16.3 purrr e listas
Outra abordagem para operações iterativas é o pacote purrr - essa é a abordagem tidyverse para iteração.
Se você tiver que executar a mesma tarefa várias vezes, provavelmente vale a pena criar uma solução generalizada que possa ser usada com várias entradas. Por exemplo, produzindo gráficos para várias jurisdições ou importando e combinando muitos arquivos.
Há também algumas outras vantagens para usar o purrr - você pode usá-lo com pipes %>%, ele lida com erros melhor do que o normal loop for, e a sintaxe é bastante limpa e simples! Se você estiver usando um loop for, provavelmente poderá fazê-lo de forma mais clara e sucinta com purrr!
Tenha em mente que o purrr é uma ferramenta de programação funcional. Ou seja, as operações que devem ser aplicadas iterativamente são agrupadas em funções. Consulte a página Escrevendo funções para aprender a escrever suas próprias funções.
O purrr também é quase inteiramente baseado em listas e vetores - então pense nisso como aplicar uma função a cada elemento dessa lista/vetor!
Carregando pacotes
O purrr faz parte do tidyverse, portanto, não há necessidade de instalar/carregar um pacote separado.
pacman::p_load(
rio, # importa/exporta
here, # caminhos de arquivos relativos
tidyverse, # gerenciamento de dados e visualização
writexl, # escreve arquivos Excel com várias abas
readxl # importa arquivos Excel com várias abas
)map()
A função principal do purrr é a map(), que “mapeia” (aplica) uma função para cada elemento de entrada de uma lista/vetor que você fornece.
A sintaxe básica é map(.x = SEQUENCE, .f = FUNCTION, OTHER ARGUMENTS). Com um pouco mais de detalhes:
-
.x =são as entradas nas quais a função.fserá aplicada iterativamente - ex. um vetor de nomes de jurisdição, colunas em um data frame ou uma lista de data frames -
.f =é a função a ser aplicada a cada elemento da entrada.x- pode ser uma função comoprint()que já existe, ou uma função personalizada que você define. A função geralmente é escrita após um til~(detalhes abaixo).
Mais algumas notas sobre a sintaxe:
- Se a função não precisar de mais argumentos especificados, ela pode ser escrita sem parênteses e sem til (por exemplo,
.f = mean). Para fornecer argumentos que terão o mesmo valor para cada iteração, forneça-os dentro damap()mas fora do argumento.f =, comona.rm = Temmap(.x = my_list, .f = média, na.rm=T). - Você pode usar
.x(ou simplesmente.) dentro da função.f =como um espaço reservado para o valor.xdessa iteração - Use a sintaxe til (
~) para ter maior controle sobre a função - escreva a função normalmente com parênteses, como:map(.x = my_list, .f = ~mean(., na.rm = T) ). Use esta sintaxe especialmente se o valor de um argumento mudar a cada iteração, ou se for o próprio valor.x(veja os exemplos abaixo)
O resultado da função map() é uma lista - uma lista é uma classe de objeto como um vetor, mas cujos elementos podem ser de classes diferentes. Assim, uma lista produzida pela map() pode conter muitos data frames, ou muitos vetores, muitos valores únicos, ou mesmo muitas listas! Existem versões alternativas da map() explicadas abaixo que produzem outros tipos de saídas (por exemplo, map_dfr() para produzir um data frame, map_chr() para produzir vetores de caracteres e map_dbl() para produzir vetores).
Exemplo - importar e combinar planilhas do Excel
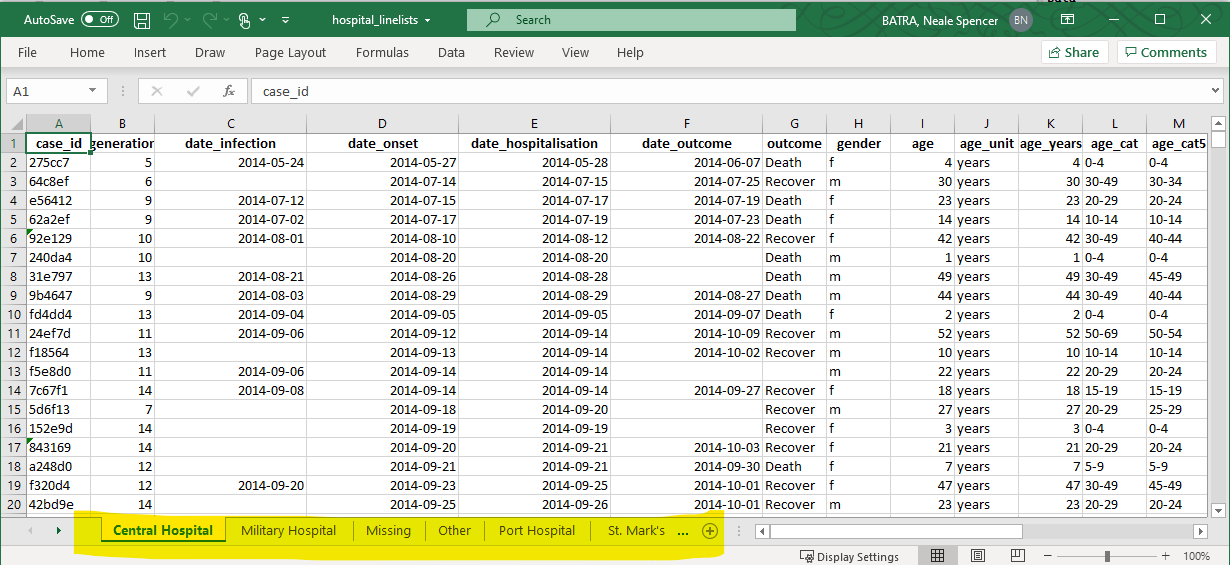
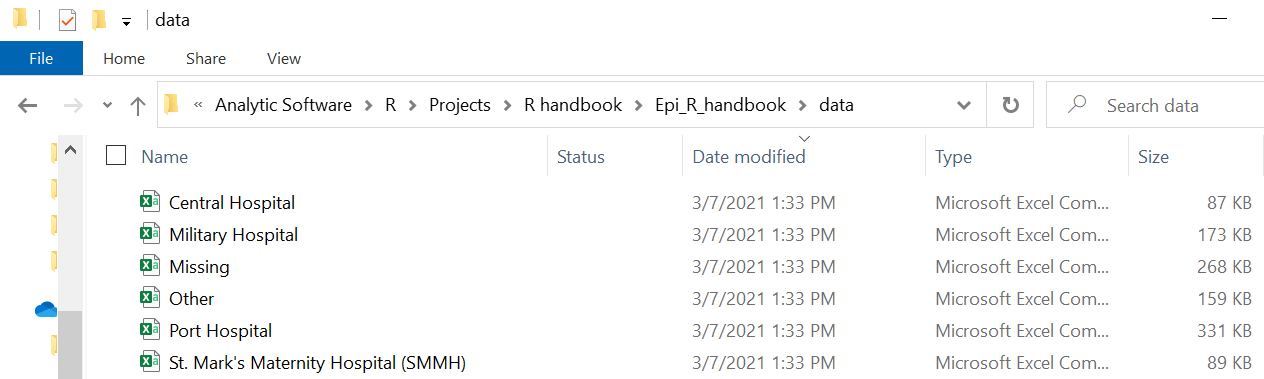
Vamos demonstrar com uma tarefa comum de um epidemiologista: - Você deseja importar um arquivo do Excel com dados do caso, mas os dados são divididos em diferentes abas com nomes. Como você importa e combina com eficiência as planilhas em um data frame?
Digamos que recebemos arquivo Excel abaixo. Cada folha contém casos de um determinado hospital.
Aqui está uma abordagem que usa a função map():
-
map()a funçãoimport()para que seja executada para aba do Excel - Combine em um os data frames importados usando
bind_rows() - Ao longo do caminho, preserve o nome da aba original para cada linha, armazenando essas informações em uma nova coluna no data frame final
Primeiro, precisamos extrair os nomes das abas e salvá-los. Fornecemos o caminho do arquivo Excel para a função excel_sheets() do pacote readxl, que extrai os nomes das abas Nós os armazenamos em um vetor de caracteres chamado sheet_names.
sheet_names <- readxl::excel_sheets("hospital_linelists.xlsx")Aqui estão os nomes:
sheet_names[1] "Central Hospital" "Military Hospital"
[3] "Missing" "Other"
[5] "Port Hospital" "St. Mark's Maternity Hospital"Agora que temos esse vetor de nomes, map() pode fornecê-los um a um para a função import(). Neste exemplo, os sheet_names são .x e import() é a função .f.
Lembre-se da página Importar e exportar que quando usado em arquivos do Excel, import() pode aceitar o argumento which = (qual) especificando a aba a ser importada. Dentro da função .f import(), fornecemos which = .x, cujo valor mudará a cada iteração através do vetor sheet_names - primeiro “Central Hospital” (“Hospital Central”), depois “Military Hospital” (“Hospital Militar”) etc.
Nota - porque usamos map(), os dados em cada planilha do Excel serão salvos como um data frame separado dentro de uma lista. Queremos que cada um desses elementos de lista (data frame) tenha um nome, então antes de passarmos sheet_names para map(), passamos por set_names() de purrr, o que garante que cada elemento da lista recebe o nome apropriado.
Salvamos a lista de saída como o objeto combined.
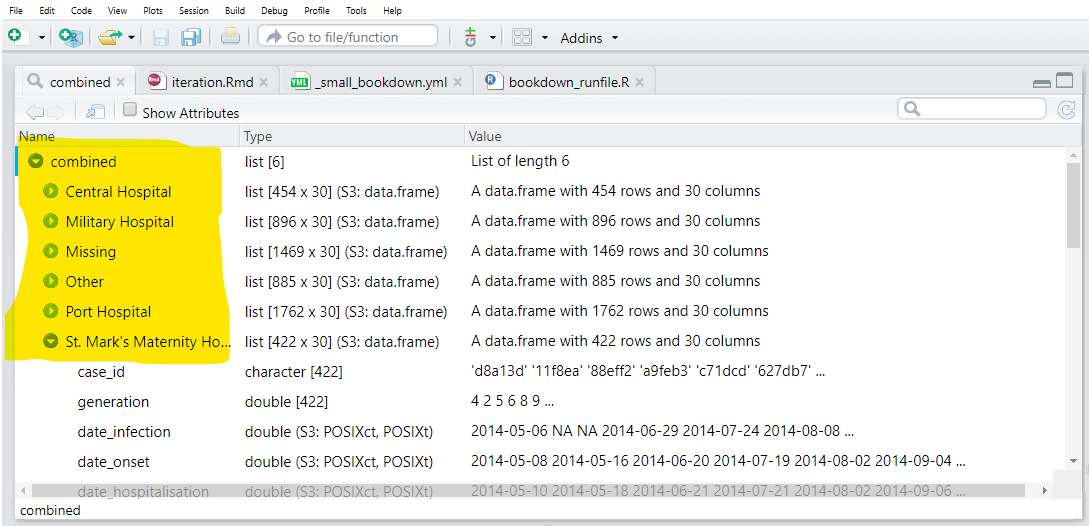
combined <- sheet_names %>%
purrr::set_names() %>%
map(.f = ~import("hospital_linelists.xlsx", which = .x))Quando inspecionamos o resultado, vemos que os dados de cada aba do Excel são salvos na lista com um nome. Isso é bom, mas ainda não terminamos.
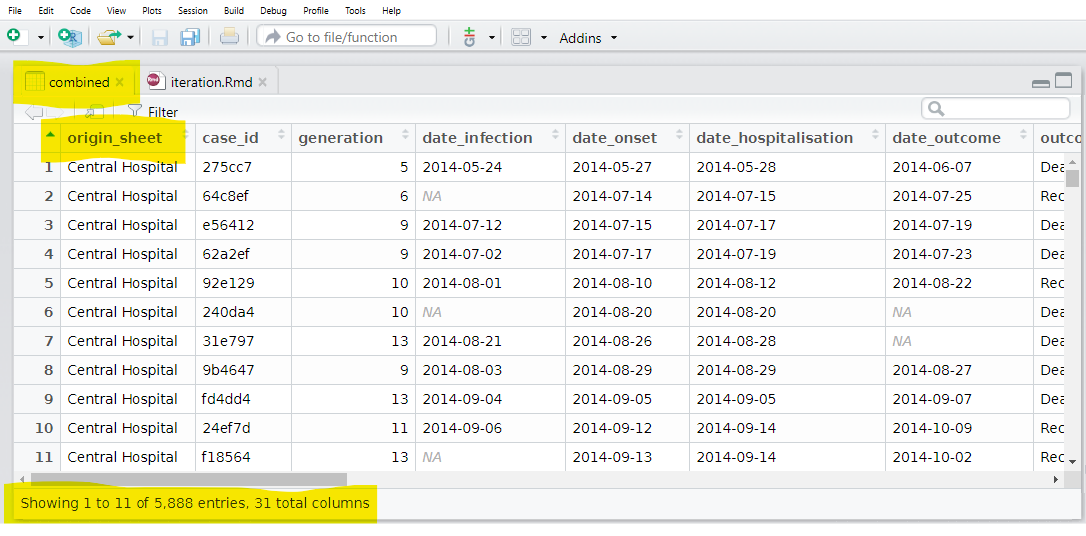
Por fim, usamos a função bind_rows() (do dplyr) que aceita a lista de data frames com estrutura semelhante e os combina em um único data frame. Para criar uma nova coluna a partir do elemento names da lista, usamos o argumento .id = e fornecemos o nome desejado para a nova coluna.
Abaixo está toda a sequência de comandos:
sheet_names <- readxl::excel_sheets("hospital_linelists.xlsx") # extraindo o nome das abas
combined <- sheet_names %>% # começando com os nomes das abas
purrr::set_names() %>% # definindo seus nomes
map(.f = ~import("hospital_linelists.xlsx", which = .x)) %>% # iterando, importando, salvando na lista
bind_rows(.id = "origin_sheet") # combinar lista de data frames, preservando a origem em uma nova colunaE agora temos um data frame com uma coluna contendo a aba de origem!
Existem variações da map() que você deve conhecer. Por exemplo, map_dfr() retorna um data frame, não uma lista. Assim, poderíamos tê-lo usado para a tarefa acima e não ter que vincular linhas. Mas aí não teríamos conseguido capturar de qual aba (hospital) veio cada caso.
Outras variações incluem map_chr(), map_dbl(). Estas são funções muito úteis por duas razões. Em primeiro lugar, elas convertem automaticamente a saída de uma função iterativa em um vetor (não uma lista). Em segundo lugar, elas podem controlar explicitamente a classe em que os dados voltam - você garante que seus dados voltem como um vetor de caracteres com map_chr(), ou vetor numérico com map_dbl(). Vamos voltar a eles mais tarde na seção!
As funções map_at() e map_if() também são muito úteis para iteração - elas permitem que você especifique em quais elementos de uma lista você deve iterar! Estes funcionam simplesmente aplicando um vetor de índices/nomes (no caso de map_at()) ou um teste lógico (no caso de map_if()).
Vamos usar um exemplo em que não queríamos ler a primeira aba de dados do hospital. Usamos map_at() em vez de map(), e especificamos o argumento .at = para c(-1) que significa não usar o primeiro elemento de .x. Alternativamente, você pode fornecer um vetor de números positivos, ou nomes, para .at = para especificar quais elementos usar.
sheet_names <- readxl::excel_sheets("hospital_linelists.xlsx")
combined <- sheet_names %>%
purrr::set_names() %>%
# excluindo a primeira aba
map_at(.f = ~import( "hospital_linelists.xlsx", which = .x),
.at = c(-1))Observe que o nome da primeira aba ainda aparecerá como um elemento da lista de saída - mas é apenas um nome de caractere único (não um data frame). Você precisaria remover esse elemento antes de vincular as linhas. Abordaremos como remover e modificar elementos de uma lista em uma seção posterior.
Divida o conjunto de dados e exporte
Abaixo, damos um exemplo de como dividir um conjunto de dados em partes e, em seguida, usar a iteração map() para exportar cada parte como uma aba separada do Excel ou como um arquivo CSV separado.
Conjunto de dados dividido
Digamos que temos o caso completo linelist como um data frame e agora queremos criar uma linelist separada para cada hospital e exportar cada um como um arquivo CSV separado. Abaixo, fazemos os seguintes passos:
Use group_split() (do dplyr) para dividir o data frame linelist por valores únicos na coluna hospital. A saída é uma lista contendo um data frame por subconjunto de hospital.
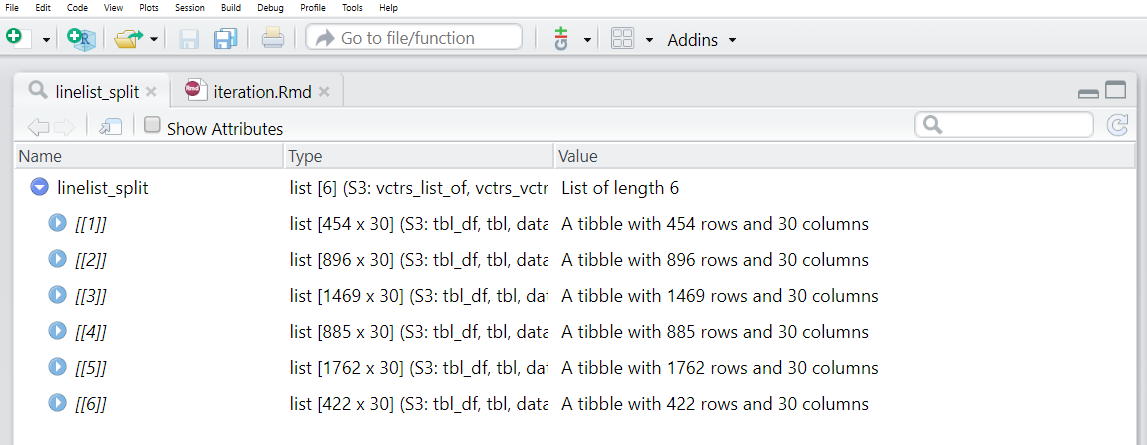
linelist_split <- linelist %>%
group_split(hospital)Podemos executar View(linelist_split) e ver que esta lista contém 6 data frames (“tibbles”), cada um representando os casos de um hospital.
No entanto, observe que os data frames na lista não possuem nomes por padrão! Queremos que cada um tenha um nome e, em seguida, use esse nome ao salvar o arquivo CSV.
Uma abordagem para extrair os nomes é usar pull() (do dplyr) para extrair a coluna hospital de cada data frame na lista. Então, por segurança, convertemos os valores em caracteres e usamos unique() para obter o nome desse data frame específico. Todas essas etapas são aplicadas a cada data frame via map().
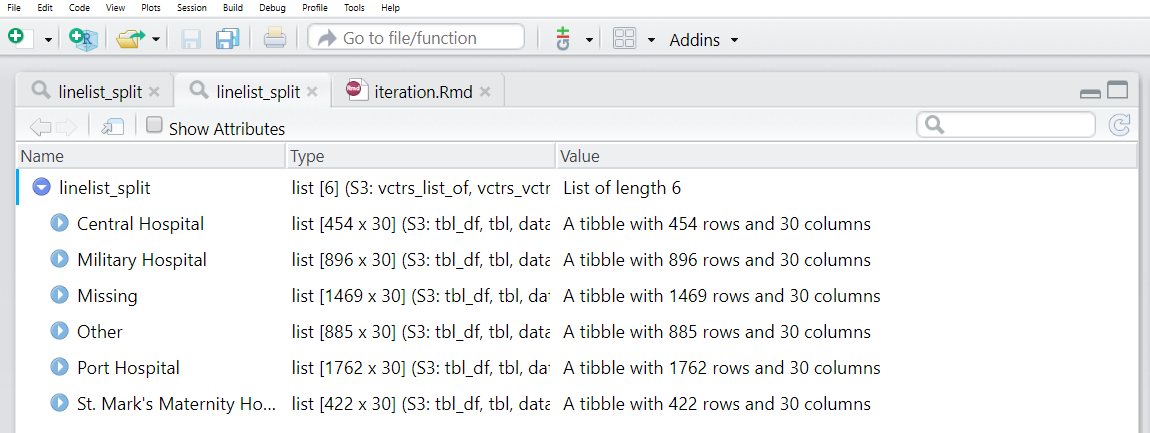
names(linelist_split) <- linelist_split %>% # Atribuindo os nomes de data frames listados
# Extraia os nomes fazendo o seguinte para cada data frame:
map(.f = ~pull(.x, hospital)) %>% # Puxe a coluna do hospital
map(.f = ~as.character(.x)) %>% # Converta em caractere, apenas por garantia
map(.f = ~unique(.x)) # Pegue o nome exclusivo do hospitalAgora podemos ver que cada um dos elementos da lista tem um nome. Esses nomes podem ser acessados via names(linelist_split).
names(linelist_split)[1] "Ausente"
[2] "Central Hospital"
[3] "Military Hospital"
[4] "Other"
[5] "Port Hospital"
[6] "St. Mark's Maternity Hospital (SMMH)"Mais de uma coluna group_split()
Se você quiser dividir a linelist por mais de uma coluna de agrupamento, como para produzir uma lista de linelist pela interseção de hospital E sexo, precisará de uma abordagem diferente para nomear os elementos da lista. Isso envolve coletar as “chaves de grupo” exclusivas usando group_keys() do dplyr - elas são retornadas como um data frame. Então você pode combinar as chaves de grupo em valores com unite() como mostrado abaixo, e atribuir esses nomes de conglomerados a linelist_split.
# dividindo a linelist por combinações exclusivas de hospital-gênero
linelist_split <- linelist %>%
group_split(hospital, gender)
# extraindo group_keys() como um data frame
groupings <- linelist %>%
group_by(hospital, gender) %>%
group_keys()
groupings # mostrando agrupamentos únicos# A tibble: 18 × 2
hospital gender
<chr> <chr>
1 Ausente f
2 Ausente m
3 Ausente <NA>
4 Central Hospital f
5 Central Hospital m
6 Central Hospital <NA>
7 Military Hospital f
8 Military Hospital m
9 Military Hospital <NA>
10 Other f
11 Other m
12 Other <NA>
13 Port Hospital f
14 Port Hospital m
15 Port Hospital <NA>
16 St. Mark's Maternity Hospital (SMMH) f
17 St. Mark's Maternity Hospital (SMMH) m
18 St. Mark's Maternity Hospital (SMMH) <NA> Agora combinamos os agrupamentos, separados por traços, e os atribuímos como os nomes dos elementos da lista em linelist_split. Isso leva algumas linhas extras, pois substituimos NA por “Ausente”, usamos unite() do dplyr para combinar os valores das colunas (separados por traços) e, em seguida, convertemos em um vetor sem nome para que ele pode ser usado como nomes de linelist_split.
# Combinando em um valor de um nome
names(linelist_split) <- groupings %>%
mutate(across(everything(), replace_na, "Ausente")) %>% # substituindo NA por "Ausente" em todas as colunas
unite("combined", sep = "-") %>% # unindo todos os valores da coluna em um
setNames(NULL) %>%
as_vector() %>%
as.list()Exportando como abas do Excel
Para exportar as linelists do hospital como um arquivo do Excel com uma linelist por aba, podemos apenas fornecer a lista nomeada linelist_split para a função write_xlsx() do pacote writexl. Isso tem a capacidade de salvar um arquivo Excel com várias abas. Os nomes dos elementos da lista são aplicados automaticamente como os nomes das abas
linelist_split %>%
writexl::write_xlsx(path = here("data", "hospital_linelists.xlsx"))Agora você pode abrir o arquivo Excel e ver que cada hospital tem sua própria aba
Exportando como arquivos CSV
É um comando um pouco mais complexo, mas você também pode exportar cada linelist específica do hospital como um arquivo CSV separado, com um nome de arquivo específico para o hospital.
Novamente usamos map(): pegamos o vetor de nomes de elementos da lista (mostrado acima) e usamos map() para iterar por eles, aplicando export() (do pacote rio, veja página Importar e exportar) no data frame na lista linelist_split que tem esse nome. Também usamos o nome para criar um nome de arquivo exclusivo. Aqui está como funciona:
Começamos com o vetor de nomes de caracteres, passado para
map()como.xA função
.féexport(), que requer um data frame e um caminho de arquivo para gravarA entrada
.x(o nome do hospital) é usada dentro de.fpara extrair/indexar aquele elemento específico da listalinelist_split. Isso resulta em apenas um data frame por vez sendo fornecido paraexport().Por exemplo, quando
map()itera para “Military Hospital” (“Hospital Militar”), entãolinelist_split[[.x]]é na verdadelinelist_split[["Military Hospital"]], retornando assim o segundo elemento delinelist_split- que são todos os casos do Hospital Militar.-
O caminho do arquivo fornecido para
export()é dinâmico através do uso destr_glue()(consulte a página Caracteres e strings):-
here()é usado para obter a base do caminho do arquivo e especificar a pasta “data” (observe as aspas simples para não interromper as aspas duplasstr_glue())
-
Em seguida, uma barra
/, e novamente o.xque imprime o nome do hospital atual para tornar o arquivo identificávelFinalmente a extensão “.csv” que
export()usa para criar um arquivo CSV
names(linelist_split) %>%
map(.f = ~export(linelist_split[[.x]], file = str_glue("{here('data')}/{.x}.csv")))Agora você pode ver que cada arquivo é salvo na pasta “data” do R Project “Epi_R_handbook”!
Customizar funções
Você pode querer criar sua própria função para fornecer ao map().
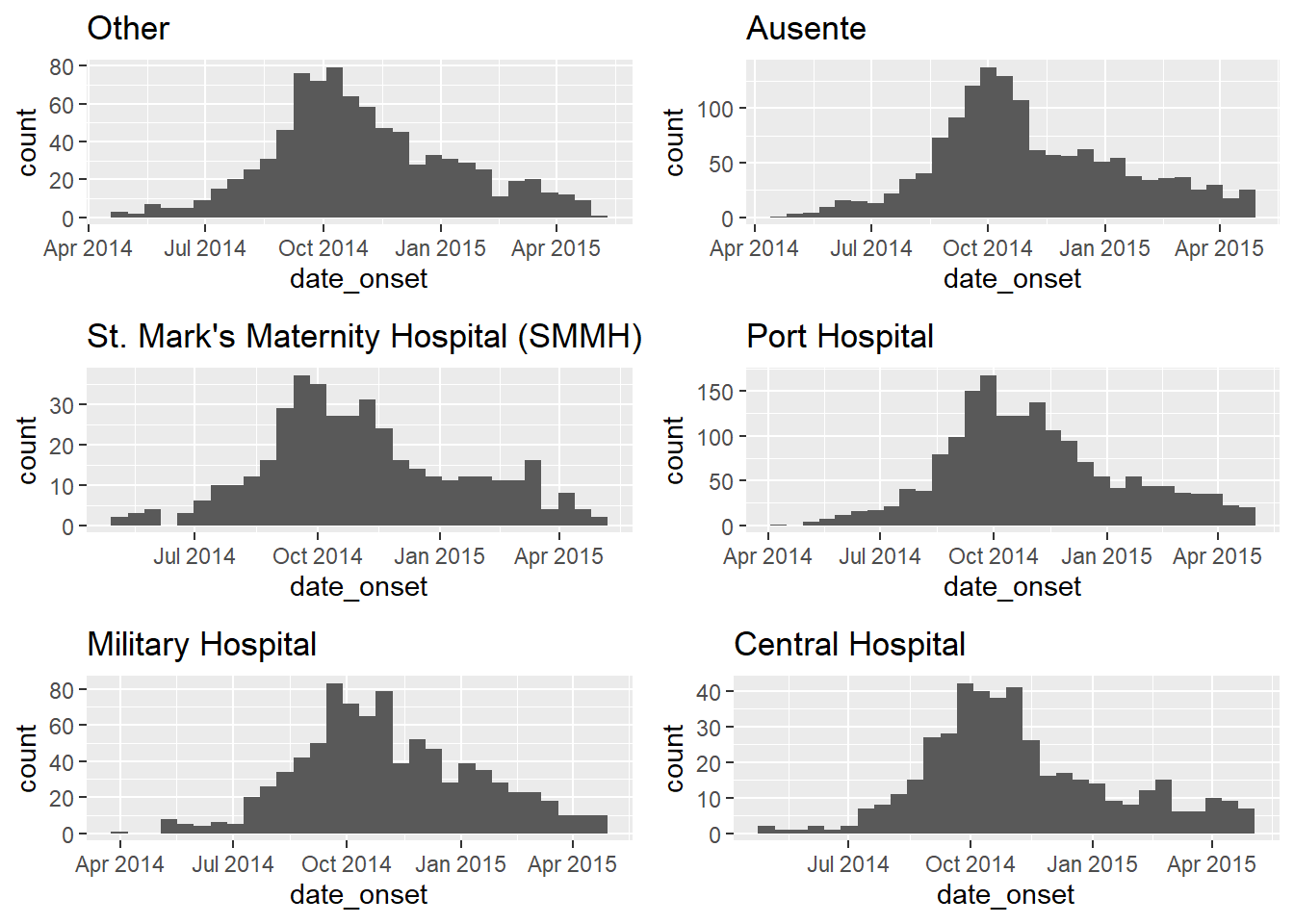
Digamos que queremos criar curvas epidêmicas para os casos de cada hospital. Para fazer isso usando purrr, nossa função .f pode ser ggplot() e extensões com + como de costume. Como a saída de map() é sempre uma lista, os gráficos são armazenados em uma lista. Por serem gráficos, eles podem ser extraídos e plotados com a função ggarrange() do pacote ggpubr (documentação ).
# carregando pacote para plotar elementos da lista
pacman::p_load(ggpubr)
# mapeando o vetor de 6 "nomes" de hospitais (criados anteriormente)
# usando a função ggplot especificada
# a saída é uma lista com 6 ggplots
hospital_names <- unique(linelist$hospital)
my_plots <- map(
.x = hospital_names,
.f = ~ggplot(data = linelist %>% filter(hospital == .x)) +
geom_histogram(aes(x = date_onset)) +
labs(title = .x)
)
# exibindo os ggplots (eles são armazenados em uma lista)
ggarrange(plotlist = my_plots, ncol = 2, nrow = 3)Se este código map() parecer muito confuso, você pode obter o mesmo resultado salvando seu comando ggplot() específico como uma função personalizada definida pelo usuário, por exemplo, podemos chamá-lo de make_epicurve()). Esta função é então usada dentro do map(). .x será substituído iterativamente pelo nome do hospital e usado como hosp_name na função make_epicurve(). Consulte a página sobre Escrevendo funções.
# Criando a função
make_epicurve <- function(hosp_name){
ggplot(data = linelist %>% filter(hospital == hosp_name)) +
geom_histogram(aes(x = date_onset)) +
theme_classic()+
labs(title = hosp_name)
}# mapeando
my_plots <- map(hospital_names, ~make_epicurve(hosp_name = .x))
# exibindo os ggplots (eles são armazenados em uma lista)
ggarrange(plotlist = my_plots, ncol = 2, nrow = 3)Mapeando uma função ao longo de colunas
Outro caso de uso comum é mapear uma função ao longo de muitas colunas. Abaixo, mapeamos (map()) a função t.test() em colunas numéricas no data frame linelist, comparando os valores numéricos por gênero.
Lembre-se da página em Testes estatísticos simples que t.test() pode receber entradas em um formato de fórmula, como t.test(coluna numérica ~ coluna binária). Neste exemplo, fazemos o seguinte:
As colunas numéricas de interesse são selecionadas de
linelist- elas se tornam as entradas.xparamap()A função
t.test()é fornecida como a função.f, que é aplicada a cada coluna numérica-
Dentro dos parênteses de
t.test():- o primeiro
~precede o.fquemap()irá iterar sobre.x - o
.xrepresenta a coluna atual sendo fornecida para a funçãot.test() - o segundo
~faz parte da equação do teste t descrita acima - a função
t.test()espera uma coluna binária no lado direito da equação. Nós fornecemos o vetorlinelist$genderindependentemente e estaticamente (observe que ele não está incluído emselect()).
- o primeiro
map() retorna uma lista, então a saída é uma lista de resultados do teste t - um elemento de lista para cada coluna numérica analisada.
# Resultados são salvos como uma lista
t.test_results <- linelist %>%
select(age, wt_kg, ht_cm, ct_blood, temp) %>% # mantendo apenas algumas colunas numéricas para mapear
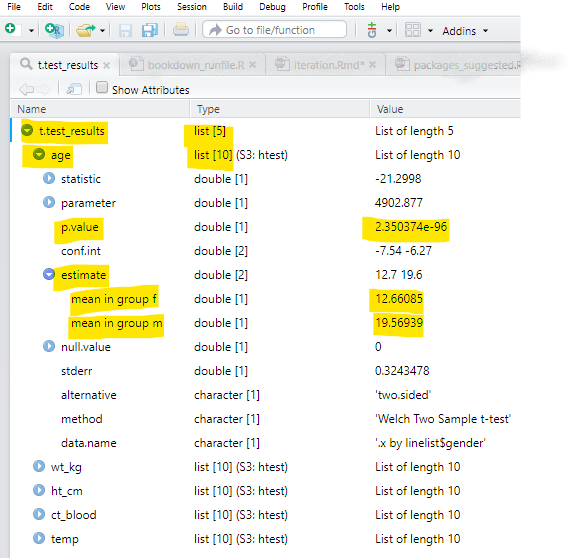
map(.f = ~t.test(.x ~ linelist$gender)) # função t.test com equação NUMERIC ~ CATEGORICALAqui está a aparência da lista t.test_results quando aberta (Visualizada) no RStudio. Destacamos partes que são importantes para os exemplos nesta página.
- Você pode ver no topo que a lista inteira é chamada de
t.test_resultse tem cinco elementos. Esses cinco elementos são nomeadosage,wt_km,ht_cm,ct_blood,tempapós cada variável que foi usada em um teste t comgenderdalinelist. - Cada um desses cinco elementos são listas, com elementos dentro deles, como
p.valueeconf.int. Alguns desses elementos comop.valuesão números únicos, enquanto alguns comoestimateconsistem em dois ou mais elementos (média no grupo femédia no grupo m).
Nota: Lembre-se que se você deseja aplicar uma função a apenas certas colunas em um data frame, você também pode simplesmente usar mutate() e across(), conforme explicado na página Limpeza de dados e principais funções. Abaixo está um exemplo de aplicação de as.character() apenas para as colunas “age”. Observe o posicionamento dos parênteses e vírgulas.
# convertendo colunas com nome da coluna contendo "idade" para classe Character
linelist <- linelist %>%
mutate(across(.cols = contains("age"), .fns = as.character)) Extraindo de listas
Como map() produz uma saída da classe List (lista), vamos gastar algum tempo discutindo como extrair dados de listas usando funções acompanhantes do purrr. Para demonstrar isso, usaremos a lista t.test_results da seção anterior. Esta é uma lista de 5 listas - cada uma das 5 listas contém os resultados de um teste t entre uma coluna do data frame linelist e sua coluna binária gender. Veja a imagem na seção acima para uma visualização da estrutura da lista.
Nomes dos elementos
Para extrair os nomes dos próprios elementos, simplesmente use names() do R base. Neste caso, usamos names() em t.test_results para retornar os nomes de cada sub-lista , que são os nomes das 5 variáveis que tiveram testes t realizados.
names(t.test_results)[1] "age" "wt_kg" "ht_cm" "ct_blood" "temp" Elementos por nome ou posição
Para extrair elementos da lista por nome ou por posição, você pode usar colchetes [[ ]] conforme descrito na página Introdução ao R. Abaixo usamos colchetes duplos para indexar a lista t.tests_results e exibir o primeiro elemento que é o resultado do teste t em age (idade).
t.test_results[[1]] # primeiro elemento por posição
Welch Two Sample t-test
data: .x by linelist$gender
t = -21.3, df = 4902.9, p-value < 2.2e-16
alternative hypothesis: true difference in means between group f and group m is not equal to 0
95 percent confidence interval:
-7.544409 -6.272675
sample estimates:
mean in group f mean in group m
12.66085 19.56939 t.test_results[[1]]["p.value"] # retorna o elemento nomeado "p.value" do primeiro elemento$p.value
[1] 2.350374e-96No entanto, abaixo vamos demonstrar o uso das funções simples e flexíveis do purrr map() e pluck() para alcançar os mesmos resultados.
pluck()
pluck() extrai elementos por nome ou por posição. Por exemplo - para extrair os resultados do teste t para idade, você pode usar pluck() assim:
t.test_results %>%
pluck("age") # alternativamente, use pluck(1)
Welch Two Sample t-test
data: .x by linelist$gender
t = -21.3, df = 4902.9, p-value < 2.2e-16
alternative hypothesis: true difference in means between group f and group m is not equal to 0
95 percent confidence interval:
-7.544409 -6.272675
sample estimates:
mean in group f mean in group m
12.66085 19.56939 Indexe níveis mais profundos especificando os níveis adicionais com vírgulas. O código abaixo extrai o elemento chamado “p.value” (o p-valor)da lista age de dentro da lista t.test_results. Você também pode usar números em vez de nomes de caracteres.
t.test_results %>%
pluck("age", "p.value")[1] 2.350374e-96Você pode extrair esses elementos internos de todos os elementos de primeiro nível usando map() para executar a função pluck() em cada elemento de primeiro nível. Por exemplo, o código abaixo extrai os elementos “p.value” de todas as listas dentro de t.test_results. A lista de resultados do teste t é o .x iterado, pluck() é a função .f sendo iterada e o valor “p-value” é fornecido para a função.
t.test_results %>%
map(pluck, "p.value") # retorna cada p-valor$age
[1] 2.350374e-96
$wt_kg
[1] 2.664367e-182
$ht_cm
[1] 3.515713e-144
$ct_blood
[1] 0.4473498
$temp
[1] 0.5735923Como outra alternativa, map() oferece uma abreviação onde você pode escrever o nome do elemento entre aspas, e ele irá “arrancá-lo”. Se você usar map() a saída será uma lista, enquanto que se você usar map_chr() será um vetor de caractere nomeado e se você usar map_dbl() será um vetor numérico nomeado.
t.test_results %>%
map_dbl("p.value") # retorna o p-valor como um vetor numérico com nome age wt_kg ht_cm ct_blood temp
2.350374e-96 2.664367e-182 3.515713e-144 4.473498e-01 5.735923e-01 Você pode ler mais sobre pluck() em sua documentação purrr. Ele tem uma função irmã chuck() que retornará um erro em vez de NULL se um elemento não existir.
Convertendo uma lista em um data frame
Este é um tópico complexo - consulte a seção Recursos para tutoriais mais completos. No entanto, demonstraremos a conversão da lista de resultados do teste t em um data frame. Criaremos um data frame com colunas para a variável, seu p-valor e as médias dos dois grupos (masculino e feminino).
Aqui estão algumas das novas abordagens e funções que serão usadas:
-
A função
tibble()será usada para criar um tibble (como um data frame)- Envolvemos a função
tibble()com chaves{ }para evitar que todo ot.test_resultsseja armazenado como a primeira coluna do tibble
- Envolvemos a função
-
Dentro de
tibble(), cada coluna é criada explicitamente, semelhante à sintaxe demutate():- O
.representat.test_results - Para criar uma coluna com os nomes das variáveis do teste t (os nomes de cada elemento da lista) usamos
names()conforme descrito acima - Para criar uma coluna com os p-valores, usamos
map_dbl()conforme descrito acima para extrair os elementosp.valuee convertê-los em um vetor numérico
- O
t.test_results %>% {
tibble(
variables = names(.),
p = map_dbl(., "p.value"))
}# A tibble: 5 × 2
variables p
<chr> <dbl>
1 age 2.35e- 96
2 wt_kg 2.66e-182
3 ht_cm 3.52e-144
4 ct_blood 4.47e- 1
5 temp 5.74e- 1Mas agora vamos adicionar colunas contendo as médias para cada grupo (masculino e feminino).
Precisaríamos extrair o elemento estimate, mas na verdade ele contém dois elementos dentro dele (média no grupo f e média no grupo m). Portanto, não pode ser simplificado em um vetor com map_chr() ou map_dbl(). Em vez disso, usamos map(), que usado dentro de tibble() criará uma coluna da lista de classes dentro do tibble! Sim, isso é possível!
t.test_results %>%
{tibble(
variables = names(.),
p = map_dbl(., "p.value"),
means = map(., "estimate"))}# A tibble: 5 × 3
variables p means
<chr> <dbl> <named list>
1 age 2.35e- 96 <dbl [2]>
2 wt_kg 2.66e-182 <dbl [2]>
3 ht_cm 3.52e-144 <dbl [2]>
4 ct_blood 4.47e- 1 <dbl [2]>
5 temp 5.74e- 1 <dbl [2]> Depois de ter essa coluna de lista, há várias funções tidyr (parte do tidyverse) que ajudam a “retangular” ou “desaninhar” essas colunas de “lista aninhada”. Leia mais sobre eles aqui, ou executando vignette("rectangle"). Em resumo:
-
unnest_wider()- dá a cada elemento de uma coluna de lista sua própria coluna -
unnest_longer()- dá a cada elemento de uma coluna de lista sua própria linha -
hoist()- funciona comounnest_wider()mas você especifica quais elementos serão desaninhados
Abaixo, passamos o tibble para unnest_wider() especificando a coluna means do tibble (que é uma lista aninhada). O resultado é que means é substituído por duas novas colunas, cada uma refletindo os dois elementos que estavam anteriormente em cada célula means.
t.test_results %>%
{tibble(
variables = names(.),
p = map_dbl(., "p.value"),
means = map(., "estimate")
)} %>%
unnest_wider(means)# A tibble: 5 × 4
variables p `mean in group f` `mean in group m`
<chr> <dbl> <dbl> <dbl>
1 age 2.35e- 96 12.7 19.6
2 wt_kg 2.66e-182 45.8 59.6
3 ht_cm 3.52e-144 109. 142.
4 ct_blood 4.47e- 1 21.2 21.2
5 temp 5.74e- 1 38.6 38.6Descartar, manter e compactar listas
Como trabalhar com purrr geralmente envolve listas, exploraremos brevemente algumas funções do purrr para modificar listas. Consulte a seção Recursos para tutoriais mais completos sobre as funções purrr.
-
list_modify()tem muitos usos, um dos quais pode ser remover um elemento da lista -
keep()retém os elementos especificados para.p =, ou onde uma função fornecida para.p =é avaliada como TRUE -
discard()remove os elementos especificados para.p, ou onde uma função fornecida para.p =é avaliada como TRUE -
compact()remove todos os elementos vazios
Aqui estão alguns exemplos usando a lista combined criada na seção acima em usando map() para importar e combinar vários arquivos (contém 6 data frames de linelist):
Elementos podem ser removidos por nome com list_modify() e definindo o nome igual a NULL.
combined %>%
list_modify("Central Hospital" = NULL) # remove elemento da lista por nomeVocê também pode remover elementos por critérios, fornecendo uma equação de “predicado” para .p = (uma equação que avalia como TRUE ou FALSE). Coloque um til ~ antes da função e use .x para representar o elemento da lista. Usando keep() os elementos da lista que forem avaliados como TRUE serão mantidos. Inversamente, se estiver usando discard(), os elementos da lista que forem avaliados como TRUE serão removidos.
# mantenha apenas elementos de lista com mais de 500 linhas
combined %>%
keep(.p = ~nrow(.x) > 500) No exemplo abaixo, os elementos da lista são descartados se suas classes não forem data frames.
# descartando elementos que não são data frame
combined %>%
discard(.p = ~class(.x) != "data.frame")Sua função de predição também pode referenciar elementos/colunas dentro de cada item da lista. Por exemplo, abaixo, os elementos da lista onde a média da coluna ct_blood é superior a 25 são descartados.
# mantenha apenas elementos onde a média da coluna ct_blood é maior que 25
combined %>%
discard(.p = ~mean(.x$ct_blood) > 25) Este comando remove todos os elementos vazios da lista:
# remove todos os elementos vazios da lista
combined %>%
compact()pmap()
ESTA SEÇÃO ESTÁ EM CONSTRUÇÃO
16.4 funções Apply
A família de funções “apply” é uma alternativa do R base ao purrr para operações iterativas. Você pode ler mais sobre eles aqui.
16.5 Recursos
A página R for Data Science sobre iteração
Vinheta sobre gravação/leitura de arquivos Excel
Um tutorial purrr por jennybc
Outro purrr tutorial por Rebecca Barter
Um tutorial purrr para map, pmap e imap